
[Python]過去11年分47都道府県別の健診結果データから地域差を可視化してみた:協会けんぽの「健診結果基本情報」
はじめに
こんにちは、機械学習勉強中のあおじるです。
以前、全国健康保険協会(協会けんぽ)の健診データを使った記事を書きましたが、もう少し詳細な健診結果の時系列データが掲載されていましたので使ってみました。
言語はPython、環境はGoogle Colaboratoryを使用しました。
使用するデータ
データは、全国健康保険協会(協会けんぽ)の生活習慣病予防健診の結果の年度、都道府県、性、年齢階級別の時系列データ「健診結果基本情報」を用います。データレイアウトはこちらです。
# データ
import pandas as pd
df_kenshin = pd.read_csv('./df_kenshin.csv', encoding='shift_jis')
print(df_kenshin.shape)
# (145888, 8)
df_kenshin = df_kenshin.iloc[:,[0,1,3,4,5,6,7]]
df_kenshin.columns = ['y','t','s','n','k','X','P']
print(df_kenshin.shape)
# (145888, 7)
print(df_kenshin.columns)
# Index(['y', 't', 's', 'n', 'k', 'X', 'P'], dtype='object')$$
\def\arraystretch{1.5}
\begin{array}{c:c:c:c:c|c:c}
\textsf{y} & \textsf{t} & \textsf{s} & \textsf{n} & \textsf{k} & \textsf{X} & \textsf{P} \\ \hline
2010 & 1 & 1 & 1 & 腹囲 & {} & {} \\
2010 & 1 & 1 & 2 & 腹囲 & {} & {} \\
\vdots & \vdots & \vdots & \vdots & \ddots & \vdots & \vdots \\
2020 & 47 & 2 & 8 & 体重 & {} & {}
\end{array}
$$
y:年度
2010~2020 の11年度分t:都道府県
1:北海道、・・・、47:沖縄 の47都道府県s:性別
1:男性、2:女性n:年齢階級(5歳階級)
1:35~39歳、2:40~44歳、3:45~49歳、4:50~54歳、5:55~59歳、6:60~64歳、7:65~69歳、8:70歳以上k:健診項目
腹囲:腹囲(cm)、
BMI:BMI(kg/m2)、
収縮期血圧:収縮期血圧(mmHg)、
拡張期血圧:拡張期血圧(mmHg)、
総コレステロール:総コレステロール(TC)(mg/dL)、
中性脂肪:中性脂肪(血清トリグリセリド)(TG)(mg/dL)、
HDL:HDLコレステロール(mg/dL)、
LDL:LDLコレステロール(mg/dL)、
GOT:AST(GOT)(U/L)、
GPT:ALT(GPT)(U/L)、
γGTP:γ-GT(γ-GTP)(U/L)、
空腹時血糖:空腹時血糖(FPG)(mg/dL)、
HbA1c:HbA1c(ヘモグロビンA1c)(NGSP値)(%)、
尿酸:尿酸(UA)(mg/dL)、
血清クレアチニン:血清クレアチニン(Cr)(mg/dL)、
eGFR:eGFR(mL/min)、
身長:身長(cm)、
体重:体重(kg)X:検査値の合計
P:検査人数
ヒートマップ表示
1.年度別都道府県別平均値
18個ある健診項目のうち、HbA1cは2014年度以降しかデータがないようですので、11年度分すべてそろっている健診項目(17項目)のみを用いることにします。
k_names = df_kenshin.query('y == 2020') \
.drop_duplicates(subset=['k']).loc[:,'k'] \
.to_list()
k_names
# ['腹囲',
# 'BMI',
# '収縮期血圧',
# '拡張期血圧',
# '総コレステロール',
# '中性脂肪',
# 'HDL',
# 'LDL',
# 'GOT',
# 'GPT',
# 'γGTP',
# '空腹時血糖',
# 'HbA1c',
# '尿酸',
# '血清クレアチニン',
# 'eGFR',
# '身長',
# '体重']
for i, k_name in enumerate(k_names):
print(i, k_name)
print(df_kenshin.query('k == "{}"'.format(k_name)) \
.iloc[:,:2] \
.groupby(['y'], as_index=False) \
.count().T)
# 0 腹囲
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 1 BMI
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 2 収縮期血圧
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 3 拡張期血圧
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 4 総コレステロール
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 5 中性脂肪
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 6 HDL
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 7 LDL
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 8 GOT
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 9 GPT
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 10 γGTP
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 11 空腹時血糖
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 12 HbA1c
# 0 1 2 3 4 5 6
# y 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752
# 13 尿酸
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 14 血清クレアチニン
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 15 eGFR
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 16 身長
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
# 17 体重
# 0 1 2 3 4 5 6 7 8 9 10
# y 2010 2011 2012 2013 2014 2015 2016 2017 2018 2019 2020
# t 752 752 752 752 752 752 752 752 752 752 752
k_names_2 = [k_name for k_name in k_names if k_name != 'HbA1c']
k_names_2
# ['腹囲',
# 'BMI',
# '収縮期血圧',
# '拡張期血圧',
# '総コレステロール',
# '中性脂肪',
# 'HDL',
# 'LDL',
# 'GOT',
# 'GPT',
# 'γGTP',
# '空腹時血糖',
# '尿酸',
# '血清クレアチニン',
# 'eGFR',
# '身長',
# '体重']健診項目ごとに、年度(y)、都道府県(t)別の平均値を計算します。
それをクロス表の形して、seaborn の heatmap でヒートマップ表示します。
# %matplotlib inline
import matplotlib.pyplot as plt
import seaborn as sns
# 平均値計算(性年齢調整前)
for i, k_name in enumerate(k_names_2):
print(i, k_name)
df = df_kenshin.query('k == "{}"'.format(k_name))
# y,t別 平均値計算
df_yt_XP = df.loc[:,['y','t','X','P']] \
.groupby(['y','t'], as_index=False) \
.sum()
print(df_yt_XP.shape)
# (517, 4)
df_yt_XperP = df_yt_XP.assign(XperP = df_yt_XP.X / df_yt_XP.P) \
.loc[:,['y','t','XperP']]
print(df_yt_XperP.shape)
# (517, 3)
# y,t別 クロス表
df_cross = df_yt_XperP.pivot(index=['t'], columns=['y'], values=['XperP']) \
.set_axis(range(2010,2020+1,1), axis='columns')
print(df_cross.shape)
# (47, 11)
# ヒートマップ表示
plt.figure(figsize=(14,14))
sns.heatmap(df_cross, annot=True, fmt='.2f', cmap='bwr', cbar=True,
cbar_kws={'pad': 0.05, 'shrink': 0.5, 'aspect': 20})
plt.tick_params(left=False, bottom=False, labeltop=True, labelright=True)
plt.xticks(rotation=0)
plt.yticks(rotation=0)
plt.xlabel('y') # plt.xlabel(None)
plt.ylabel('t') # plt.ylabel(None)
plt.show()

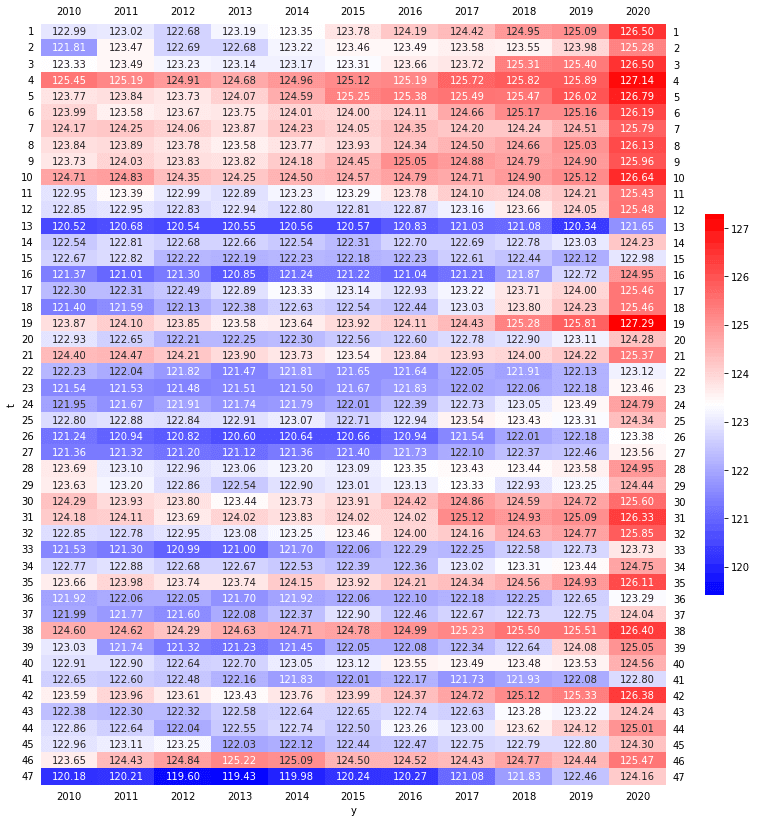














2020年度は健診の受診控えもあって受診者の背景が他の年度と違ったのか、少し数値の出方が違う検査項目もあるようです。
2.性・年齢調整平均値
健診の数値は、性・年齢による影響を受けると思われますので、性・年齢構成を調整してみることにします。
調整方法は、以前の記事と同じ直接法を用いることとし、性・年齢構成は2010年度(データの初年度)の性・年齢構成を基準としてそれにそろえます。
結果は1.と同様にヒートマップ表示します。
# 性・年齢調整値計算
for i, k_name in enumerate(k_names_2):
print(i, k_name)
# y,t別 性・年齢調整計算
df = df_kenshin.query('k == "{}"'.format(k_name))
df_ytsn_XP = df.loc[:,['y','t','s','n','X','P']] \
.groupby(['y','t','s','n'], as_index=False) \
.sum()
df_ytsn_XP = df_ytsn_XP.assign(XperP = df_ytsn_XP.X / df_ytsn_XP.P)
print(df_ytsn_XP.shape)
# (8272, 7)
df_sn_P = df_kenshin.query('k == "{}" & y == 2010'.format(k_name)) \
.loc[:,['s','n','P']] \
.groupby(['s','n'], as_index=False) \
.sum() \
.rename(columns={'P': 'Psn'})
print(df_sn_P.shape)
# (16, 3)
df_P = df_kenshin.query('k == "{}" & y == 2010'.format(k_name)) \
.loc[:,['P']] \
.sum()
df_P.shape
# (1,)
df_sn_W = df_sn_P.assign(P = df_P.P,
Wsn = df_sn_P.Psn / df_P.P)
print(df_sn_W.shape)
# (16, 5)
df_ytsn_XPW = pd.merge(df_ytsn_XP, df_sn_W,
on=['s','n'], how='left') \
.loc[:,['y','t','s','n','XperP','Wsn']]
df_ytsn_adjXperP = df_ytsn_XPW.assign(adjXperP = df_ytsn_XPW.XperP * df_ytsn_XPW.Wsn) \
.loc[:,['y','t','adjXperP']] \
.groupby(['y','t'], as_index=False) \
.sum()
print(df_ytsn_adjXperP.shape)
# (517, 3)
# y,t別 クロス表
df_cross = df_ytsn_adjXperP.pivot(index=['t'], columns=['y'], values=['adjXperP']) \
.set_axis(range(2010,2020+1,1), axis='columns')
print(df_cross.shape)
# (47, 11)
# ヒートマップ表示
plt.figure(figsize=(14,14))
sns.heatmap(df_cross, annot=True, fmt='.2f', cmap='bwr', cbar=True,
cbar_kws={'pad': 0.05, 'shrink': 0.5, 'aspect': 20})
plt.tick_params(left=False, bottom=False, labeltop=True, labelright=True)
plt.xticks(rotation=0)
plt.yticks(rotation=0)
plt.xlabel('y') # plt.xlabel(None)
plt.ylabel('t') # plt.ylabel(None)
plt.show()









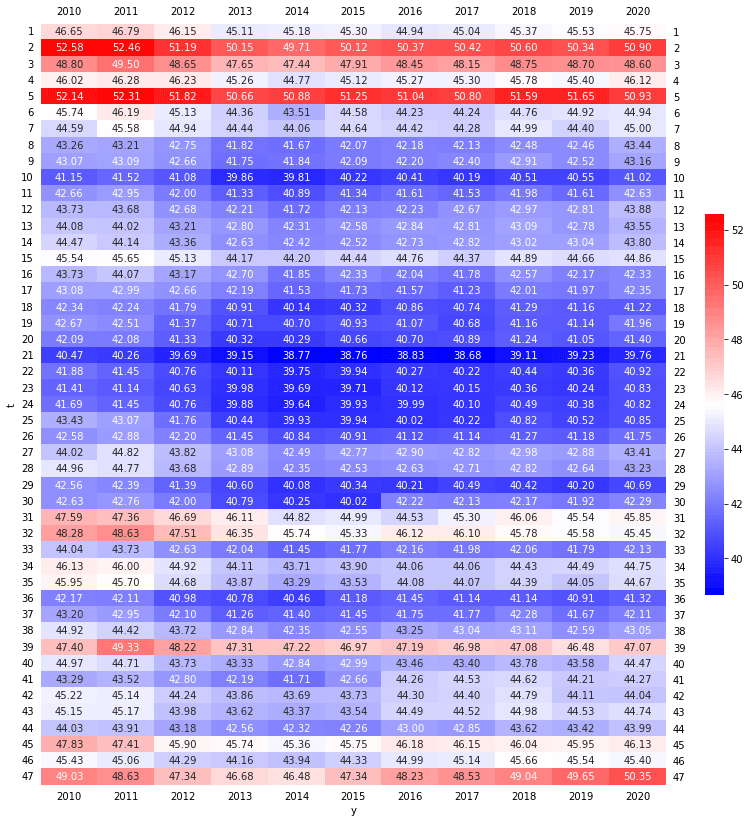






2010年度から2020年度にかけて時間を経るにしたがって、どの健診項目も概ね全体的に(全国的に)数値が悪化する方向にシフトしているように見えます。
3.全国平均からの乖離率
各都道府県の特徴を見るために、各年度の全国平均からの乖離率を計算してみます。
# 全国平均からの乖離率計算
for i, k_name in enumerate(k_names_2):
print(i, k_name)
df = df_kenshin.query('k == "{}"'.format(k_name))
# y,t別 性・年齢調整計算
df_ytsn_XP = df.loc[:,['y','t','s','n','X','P']] \
.groupby(['y','t','s','n'], as_index=False) \
.sum()
df_ytsn_XP = df_ytsn_XP.assign(XperP = df_ytsn_XP.X / df_ytsn_XP.P)
print(df_ytsn_XP.shape)
# (8272, 7)
df_sn_P = df_kenshin.query('k == "{}" & y == 2010'.format(k_name)) \
.loc[:,['s','n','P']] \
.groupby(['s','n'], as_index=False) \
.sum() \
.rename(columns={'P': 'Psn'})
print(df_sn_P.shape)
# (16, 3)
df_P = df_kenshin.query('k == "{}" & y == 2010'.format(k_name)) \
.loc[:,['P']] \
.sum()
df_P.shape
# (1,)
df_sn_W = df_sn_P.assign(P = df_P.P,
Wsn = df_sn_P.Psn / df_P.P)
print(df_sn_W.shape)
# (16, 5)
df_ytsn_XPW = pd.merge(df_ytsn_XP, df_sn_W,
on=['s','n'], how='left') \
.loc[:,['y','t','s','n','XperP','Wsn']]
df_yt_adjXperP = df_ytsn_XPW.assign(adjXperP = df_ytsn_XPW.XperP * df_ytsn_XPW.Wsn) \
.loc[:,['y','t','adjXperP']] \
.groupby(['y','t'], as_index=False) \
.sum()
print(df_yt_adjXperP.shape)
# (517, 3)
# y別 全国平均の性年齢調整計算
df_ysn_XP = df.loc[:,['y','s','n','X','P']] \
.groupby(['y','s','n'], as_index=False) \
.sum()
df_ysn_XP = df_ysn_XP.assign(XperP = df_ysn_XP.X / df_ysn_XP.P)
print(df_ysn_XP.shape)
# (11, 6)
df_ysn_XPW = pd.merge(df_ysn_XP, df_sn_W,
on=['s','n'], how='left') \
.loc[:,['y','s','n','XperP','Wsn']]
df_y_adjXperP = df_ysn_XPW.assign(adjXperPy = df_ysn_XPW.XperP * df_ysn_XPW.Wsn) \
.loc[:,['y','adjXperPy']] \
.groupby(['y'], as_index=False) \
.sum()
print(df_y_adjXperP.shape)
# (11, 2)
# y,t別 全国平均からの乖離率計算
df_yt_D = pd.merge(df_yt_adjXperP, df_y_adjXperP,
on=['y'], how='left')
df_yt_D = df_yt_D.assign(D = (df_yt_D.adjXperP - df_yt_D.adjXperPy) / df_yt_D.adjXperPy) \
.loc[:,['y','t','D']]
print(df_yt_D.shape)
# (517, 3)
# y,t別 クロス表
df_cross = df_yt_D.pivot(index=['t'], columns=['y'], values=['D']) \
.set_axis(range(2010,2020+1,1), axis='columns')
print(df_cross.shape)
# (47, 11)
# ヒートマップ
plt.figure(figsize=(14,14))
sns.heatmap(df_cross, annot=True, fmt='.2%', cmap='bwr', cbar=True,
cbar_kws={'pad': 0.05, 'shrink': 0.5, 'aspect': 20})
plt.tick_params(left=False, bottom=False, labeltop=True, labelright=True)
plt.xticks(rotation=0)
plt.yticks(rotation=0)
plt.xlabel('y') # plt.xlabel(None)
plt.ylabel('t') # plt.ylabel(None)
plt.show()
















時系列を並べてみることで、年度を経ても変わらない都道府県の特徴がかなりはっきりと出ている気がします。
47沖縄は、全体的に数値がよくないのが目立ちます(特に、腹囲、BMI、中性脂肪、HDLコレステロール、ALT(GPT)、γ-GTP、尿酸)。
その他、目立つところは以下のとおりです。
・03岩手~10群馬、19山梨、30和歌山、31鳥取、38愛媛は血圧が高め
・14神奈川、29奈良、30和歌山はコレステロールが高め
・03岩手、05秋田は中性脂肪が高め
・07福島、08茨城はHDLコレステロールが低め
・29奈良、35山口はLDLコレステロールが高め
・01北海道~07茨城(特に02青森、03岩手、05秋田)は肝機能が悪め
・02青森、03岩手、05秋田、39高知は血糖値が高め
・39高知は尿酸値が高め(47沖縄が飛び抜けて高いがその次に)
・19山梨、20長野は腎機能が悪め
おわりに
今回は、健診結果の都道府県ごとの時系列データをヒートマップ表示してみました。
データ初年度(2010年度)を基準として性・年齢調整した結果を見ると、どの健診項目も全体的に(全国的に)数値が悪化しているようでした。
全国平均からの乖離率を見ると、どの健診項目も年度を経ても変わらない都道府県の特徴がはっきり見えました。
最後まで読んでいただき、ありがとうございました。
お気づきの点等ありましたら、コメントいただけますと幸いです。
#地域差 , #地域間格差 , #都道府県 , #健診 , #健康診断 , #健診項目 , #健診結果 , #健診データ , #生活習慣病予防健診 , #腹囲 , #BMI , #血圧 , #コレステロール , #中性脂肪 , #血糖 , #尿酸 , #肝機能 , #腎機能 , #クレアチニン , #eGFR , #身長 , #体重 , #年齢調整 , #性年齢調整 , #ヒートマップ , #heatmap , #seaborn , #Python , #協会けんぽ , #noteで数式
