
RNA-seq解析をウェブブラウザ上でできる!〜iDEPを使ってみた
こんにちは。NGS解析をしたいと思い立ってR言語を勉強し始めた生命科学系研究者のえいこです。生命科学分野の便利ツールが紹介されている「統合TV」のサイトを見ていたら、RNA-seqの解析をウェブブラウザ上で解析できるiDEPの紹介動画がありました。
NGS解析に関する知識はまだまだで勉強途中で、実践に使えるのはまだまだ先。ウェブブラウザ上で動くんだったらすぐに使えて、便利なのでは?と思ったので、どんなものか試しに使ってみました。
参考にした統合TVの動画はこちら。
iDEPでどんなことができるのかまとめていきます。
----------
◇ iDEPはデータがあればすぐに解析できる!
iDEPはウェブブラウザ上でRNA-seqデータ解析が簡単にできるツールです。バックで動いているのはR。Rの可能性を感じられるサイトになっています。まずは、iDEPのサイトにアクセスしてみましょう。
シンプルでわかりやすいインターフェイス。アップロードできるデータは、カウントデータ、p値・FDR値が入ったデータ、サンプルデータ、実験条件データなどです。データのフォーマットの詳細については、こちらをご覧ください。
今回はiDEPのサイトで用意されているデモデータを使って、どんなことができるのか見ていきたいと思います。サイトの左上の"Click here to load demo data"をクリックすると右側の画面にデータセットが表示されます。

上側のカラムには各実験の条件やサンプルについての情報が、下側のカラムには遺伝子名とカウント情報が載っています。
次に、簡単にデータの概要を見てみましょう。一番上のツールバーの"Pre Process"をクリックしましょう。


各サンプルの全カウント数や、2サンプル間の発現の違いを見るためのScatterグラフを描いてくれます。ScatterグラフのX軸とY軸はピンクの〇で囲ったところで変えられます。
また、個々の遺伝子発現の違いを見るためには左側のメニューバーの"Plot one or more genes"(ピンクの〇)をクリックして、オレンジの〇の部分に遺伝子IDを入れると見ることができます。

これで着目している遺伝子がどう動いているかを見ることができますね。
さて、"Pre Process"でデータの概要を見てきました。次はiDEPでどんな解析ができるのか、ざっと見ていきましょう。
◇ iDEPでできる解析
iDEPは上のツールバーをクリックすることで、解析が一発でできます。
できる解析は、
・Heatmap作成
・k-Means
・PC解析
・DEG(変動遺伝子)
・Pathway解析
・Genome
・Bicluster
... etc.
とまぁ、論文を書くために必要な解析はカバーしているのではないかと思います。太文字で描いた解析について、どんな感じで解析されるのか見てみましょう。
・Heatmap作成

ツールバーの"Heatmap"をクリックすると自動的にHeatmapを作ってくれます。微調整などは左側のメニューバーからできます。
・PC解析

論文でよく見る主要成分解析(PC解析)も簡単にできます。
・DEG(変動遺伝子)
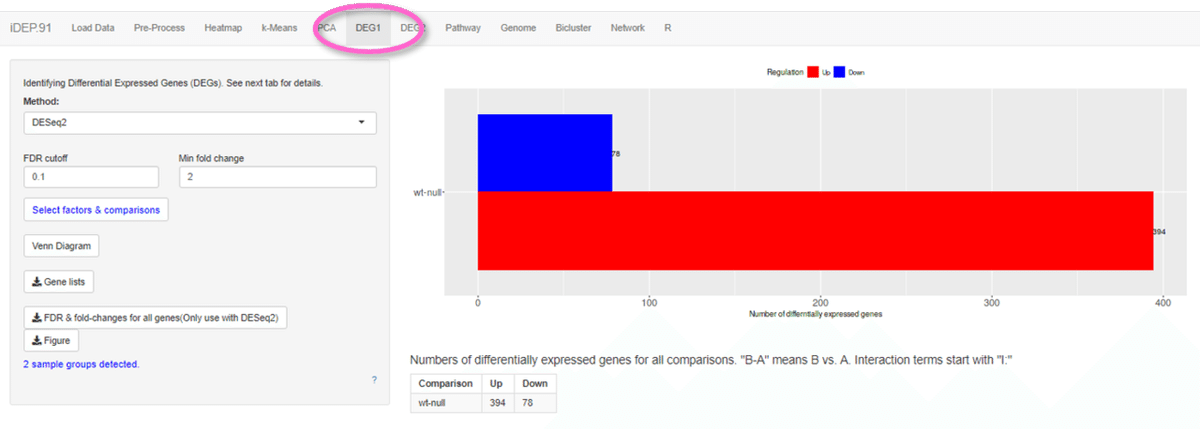
ツールバーの"DEG1"をクリックすると発現上昇した遺伝子と発現低下した遺伝子の数を表示してくれます。"DEG2"との違いはまだよくわかりません。
・Pathway解析
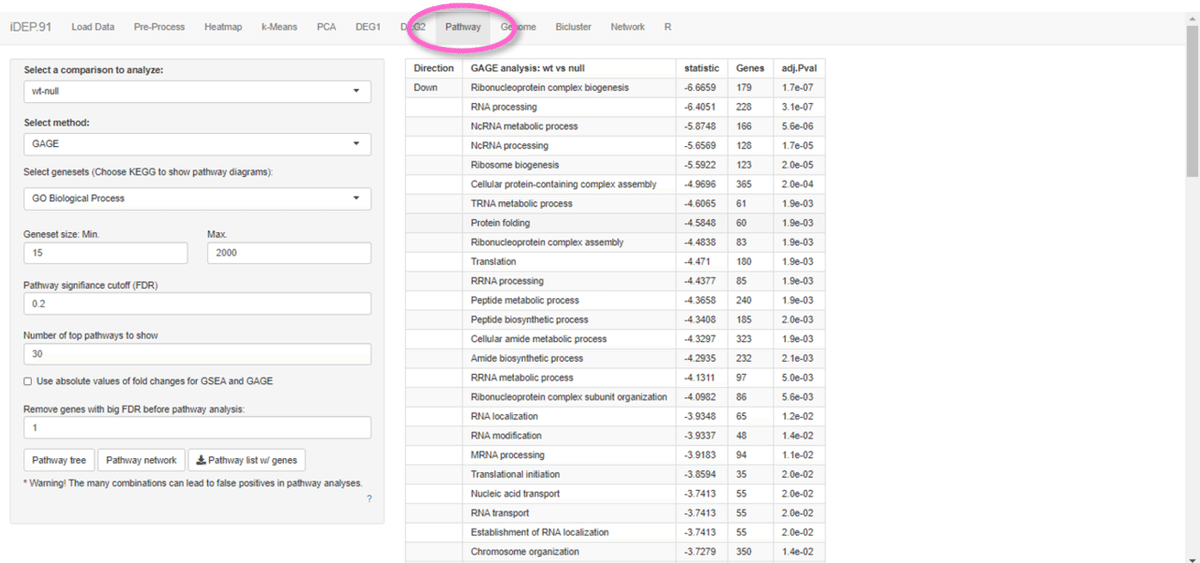
変動した遺伝子で濃縮できるPathwayを多い順に表示してくれます。デフォルトはGo pathway解析になっています。
また、抽出されたPathwayの関係性も、

・Genome

ゲノム上のどこに、変動遺伝子が集積しているのかビジュアル化もできます。
・Bicluster


クラスターごとの遺伝子の一覧や、濃縮されているPathwayも見ることができます。
と、ボタン一つでこれだけのことができるようになるんだったらもうRの勉強しなくても良いのでは?とも思っています…RNA-seqの解析に関してはまだまだ分からないことが多いので勉強するつもりですが。ちなみに、iDEPでのRのコードは全て公開されています。
最後に…
これだけ便利なツールを作って維持するのはかなり大変。解析に使ったときは必ず下記の論文を引用しましょう!
Ge SX, Son EW, Yao R: iDEP: an integrated web application for differential expression and pathway analysis of RNA-Seq data. BMC Bioinformatics 2018, 19(1):534. PMID:30567491
それでは、また!
いいなと思ったら応援しよう!

