
[プログラム・コード公開] コピペだけで15の変数選択手法を一気に実行して結果を比較する! (R言語)
[New] 実業家として有名な堀江貴文さん(ホリエモン)のメルマガ[Vol.359]における副業紹介において、このプログラム・コード販売が、なんと 1番 に紹介され評価していただきました!
こんにちは!大学教員ブロガーのねこしです。http://univprof.com/
仕事や研究において、重要な変数を決めるため、いろいろな変数選択手法を検討する方もいらっしゃいます。重要な変数を選ぶときに、いくつかの変数選択手法を実行して、どんな変数が何回選ばれたかを確認する方法はこちらに書きました。
http://univprof.com/archives/17-02-09-12105097.html
しかし、重要な変数を選択する方法はわかっても、実際に変数選択ができるようになるわけではありません。ネットや本でプログラミングを説明しているものはありますが、データの読み込み方とか結果の出し方とか、他にも調べてやらなくちゃいけないこと、多いんですよね・・・。手間と時間がかかります。
そこで、すぐに15の変数選択手法を一気に実行して結果を比較できるプログラムを作りました。R言語で実行ができます。
15の変数選択手法はこちらです。
■相関係数による変数選択:すべての変数の間の相関係数を計算し、その絶対値が大きい変数の組の一方を削除する。最初は相関係数の絶対値の閾値を0.9にしているが、変更もできる。
■非線形の相関係数(Maximum Information Coefficient, MIC)による変数選択:すべての変数の間のMICを計算し、その値が大きい変数の組の一方を削除する。最初はMICの閾値を0.9にしているが、変更もできる。
■Variable Importance in Projection (VIP):Partial Least Squares (PLS) を実施した後のVIPという指標を用いて、VIPの値が大きい変数のみを選択する。最初はVIPの値の閾値を1にしているが、変更もできる。
■PLS-β:PLSを実施した後の標準回帰係数の絶対値を用いて、その値が大きい変数を選択する。最初は閾値を標準回帰係数の絶対値の中央値にしているが、変更もできる。
■PLSの繰り返しによる変数選択:不要な変数を一つずつ削除しながら、ダブルクロスバリデーション( http://univprof.com/archives/16-06-12-3889388.html )の結果が良くなるように変数を選択する手法。詳しくはこちら( http://univprof.com/archives/16-11-22-8587099.html )。
■Least Absolute Shrinkage and Selection Operator (LASSO):LASSOではモデルに寄与しない変数の標準回帰係数の値が0になりやすい。標準回帰係数の値が0でない変数のみ選択する。
■Elastic Net (EN):ENでもLASSOと同様に標準回帰係数の値が0になりやすい。0でない変数を選択する。
■Random Forest (RF) の変数の重要度に基づく変数選択:RFを実行した後の変数の重要度が大きい変数を選択する。
■Genetic Algorithm-based Partial Least Squares(GAPLS):生物の進化の仮定を模倣して、PLSのクロスバリデーションの結果がよくなるような変数の組み合わせを選択する。
■Stepwise Backward Akaike’s Information Criterion (AIC):モデル構築を繰り返して、変数を1つずつ削除しながら、AIC(赤池情報量規準)の値がよくなるように変数の組み合わせを選択する。
■Stepwise Backward Bayesian Information Criterion (BIC):モデル構築を繰り返して、変数を1つずつ削除しながら、BIC(ベイズ情報量規準)の値がよくなるように変数の組み合わせを選択する。
■Stepwise Forward Akaike’s Information Criterion (AIC):モデル構築を繰り返して、変数を1つずつ追加しながら、AICの値がよくなるように変数の組み合わせを選択する。
■Stepwise Forward Bayesian Information Criterion (BIC):モデル構築を繰り返して、変数を1つずつ追加しながら、BICの値がよくなるように変数の組み合わせを選択する。
■Stepwise Forward-Backward Akaike’s Information Criterion (AIC):モデル構築を繰り返して、変数を1つずつ追加したり削除しながら、AICの値がよくなるように変数の組み合わせを選択する。
■Stepwise Forward-Backward Bayesian Information Criterion (BIC):モデル構築を繰り返して、変数を1つずつ追加したり削除しながら、BICの値がよくなるように変数の組み合わせを選択する。
データ形式・必要なソフトウェア
以下の記事に示す形式のデータ(回帰分析用のdata.csv)さえ準備すれば、R言語で15の変数選択手法を一気に実行することが可能です。なお今回はdata_prediction1.csv, data_prediction2.csvは必要ありません。
R言語のために必要なソフトウェアは以下の記事をご覧ください。
実行結果
実行結果を下に示します。最後に以下のcsvファイルが同じディレクトリ(フォルダ)に保存されます。
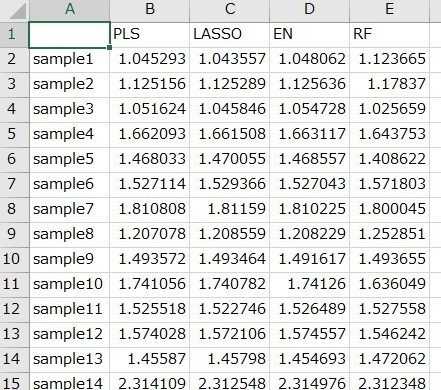
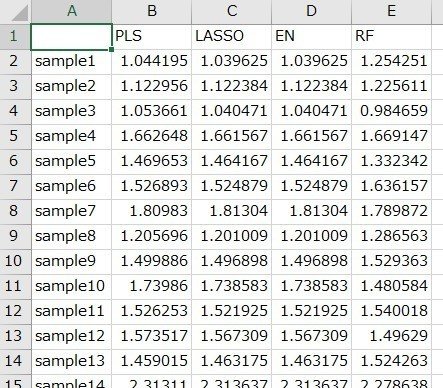
■CalculatedY.csv ・・・ それぞれの回帰分析手法(PLS, LASSO, EN, RF,)におけるモデル構築用データの目的変数の計算値
■PredictedYcv.csv ・・・ それぞれの回帰分析手法(PLS, LASSO, EN, RF)におけるモデル構築用データの目的変数のクロスバリデーション予測値
■StatisticsAll.csv ・・・ それぞれの回帰分析手法におけるモデル構築用データのr^2・RMSE、クロスバリデーション後のr^2cv・RMSEcvの値
■SelectedVariable.csv ・・・ 15の変数選択手法で選択された変数と、変数ごとの選択された回数
■ScoreAll.csv ・・・ 以下のような各変数選択手法における変数ごとのスコアの値
・PLSの標準回帰係数の絶対値
・VIPの値
・LASSOの標準回帰係数の絶対値
・ENの標準回帰係数の絶対値
・RFにおける変数ごとの重要度




このプログラムからスタートしてさらにプログラミングを進めたいと考えている方にもぜひ利用していただければと思います。
プログラム公開
ここまでお読みいただきありがとうございます。
R言語のプログラムは有料コンテンツとします。ただこれにより、こちらに記載した複数の変数選択手法をすぐに実行できます。
http://univprof.com/archives/17-02-09-12105097.html
こちらからプログラムのzipファイル自体はダウンロードできます。
http://univprofblog.html.xdomain.jp/code/varselall_analysis_all_e_r_pass.zip
購入していただくと解凍のためのパスワードがありますのでそちらをご利用ください。
またこちらのzipファイルに必要なスクリプトと関数があります。パスワードはかけていません。購入後に使い方の説明があります。
http://univprofblog.html.xdomain.jp/code/R_scripts_functions.zip
ここから先は
¥ 9,800
この記事が気に入ったらチップで応援してみませんか?
