
【R Studio】生存曲線の描き方
概要
何らかの生物を用いて寿命を測り、生存率を比較する実験では、生存曲線を図として作成することになる。昨今はChatGPTに指示すればコードを書いてもらえるが、書いてもらったコードをコピペしてもエラーを起こすことがしばしばある。そこで本記事では、Rでsurvivalというパッケージを用いて簡便に生存曲線を描画する方法を紹介する。
R version 4.3.0 , R Studio 2024.04.2+764 で実行
データの準備
元となる寿命測定のデータでは、グループ名と寿命の2項目が必要となる。たとえば下のExcelファイルのように、groupの列に記された各個体(Aの1個体目、2個体目、・・・・・・、100個体目、Bの1個体目、2個体目、・・・・・・、100個体目)に対し、それぞれに対応する寿命をlifespanの列に記載する。どの個体がどれくらい生きたかの対応ができていればよいので、グループごとのまとまりを作る必要はない(たとえばデータの並びがAABABBBABABのようになっていても構わない)。
このファイルを読み込む。
library(openxlsx)
data <- read.xlsx("sample.xlsx")生存率を比較したり、生存曲線を描画したりするには、survivalパッケージを使用する。
install.packages("survival")
library(survival)2群間の生存率の違いを検証
では、survdiff関数を使って、2群の生存率に有意差があるかを検証する。この関数ではLog-rank検定と呼ばれる検定を行っている。
デフォルトだとp値が小数第一位までしか表示されないので、digitsを指定して詳細な数値が表示されるようにしておく。
死亡か打ち切りかのパラメータを加えたstatusという項目が使われることがあるが、今回は全個体が死亡したものとして扱うため、その項目はない(線虫やショウジョウバエのような数ヶ月で死亡する動物は全個体が死亡するまで観測するのが容易であるが、マウスやヒトなどの何年も生きる動物では打ち切りを考慮する必要が出てくるだろう)。
options(digits = 10)
survdiff(Surv(lifespan) ~ group, data = data)結果は、
Call:
survdiff(formula = Surv(lifespan) ~ group, data = data)
N Observed Expected (O-E)^2/E (O-E)^2/V
group=A 100 100 144.4964 13.7023 59.7275
group=B 100 100 55.5036 35.6720 59.7275
Chisq= 59.7 on 1 degrees of freedom, p= 1.089e-14 最後に表示されているp値が非常に小さいことから、AとBの生存率は有意に異なっていると考えられる。
生存曲線の描画
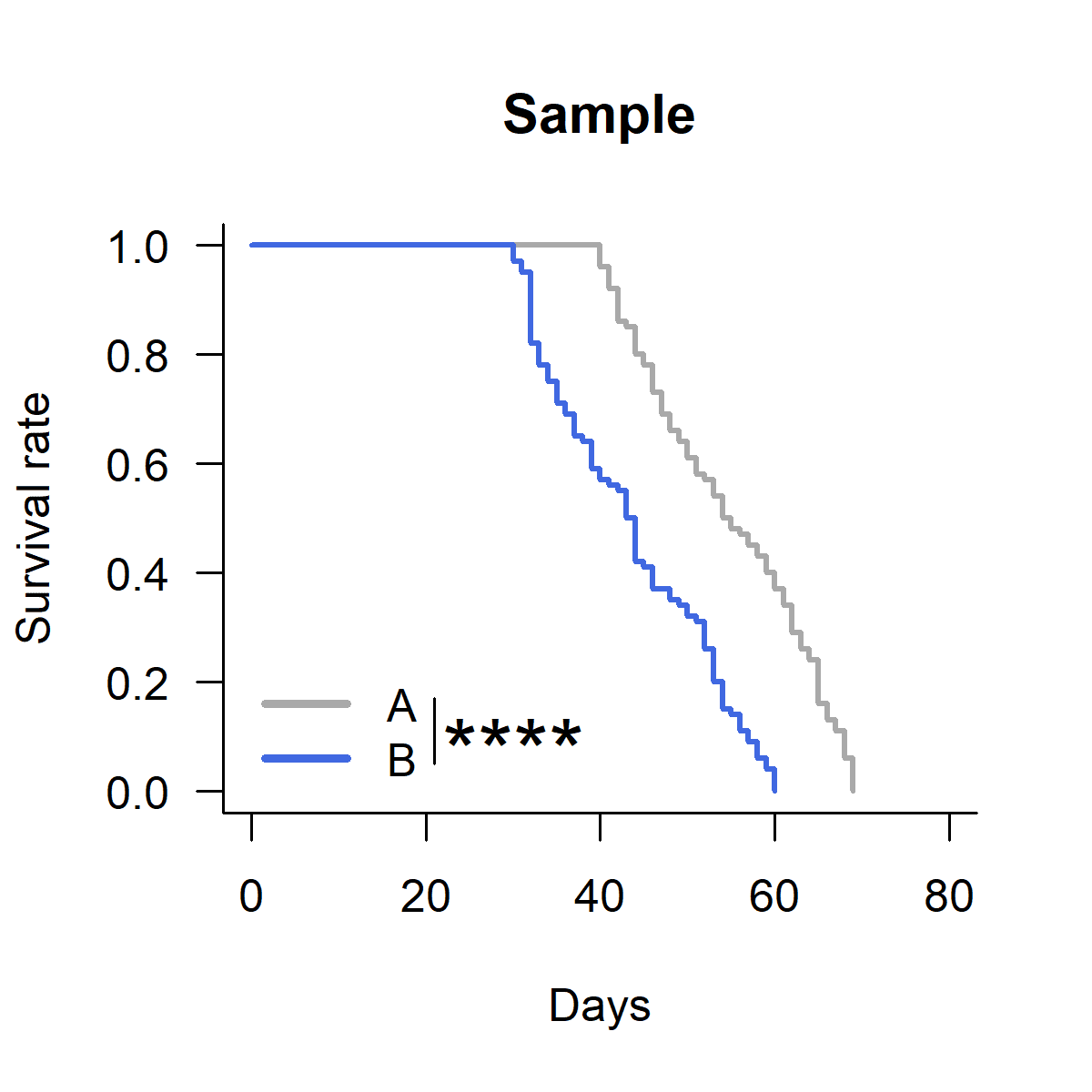
続いて、生存曲線を描画する。survfit関数を用いて生存率を推定し、それをプロットする(図1)。lasはx軸とy軸に表示される数字の向き、lwdは線の太さ、btyは座標軸の形を指定している。
survfit <- survfit(Surv(lifespan) ~ group, data = data)
plot(survfit, main = "Sample", las = 1, lwd = 2, bty = "L", xlim = c(0,80), xlab = "Days", ylab = "Survival rate", col = c("darkgrey", "royalblue"))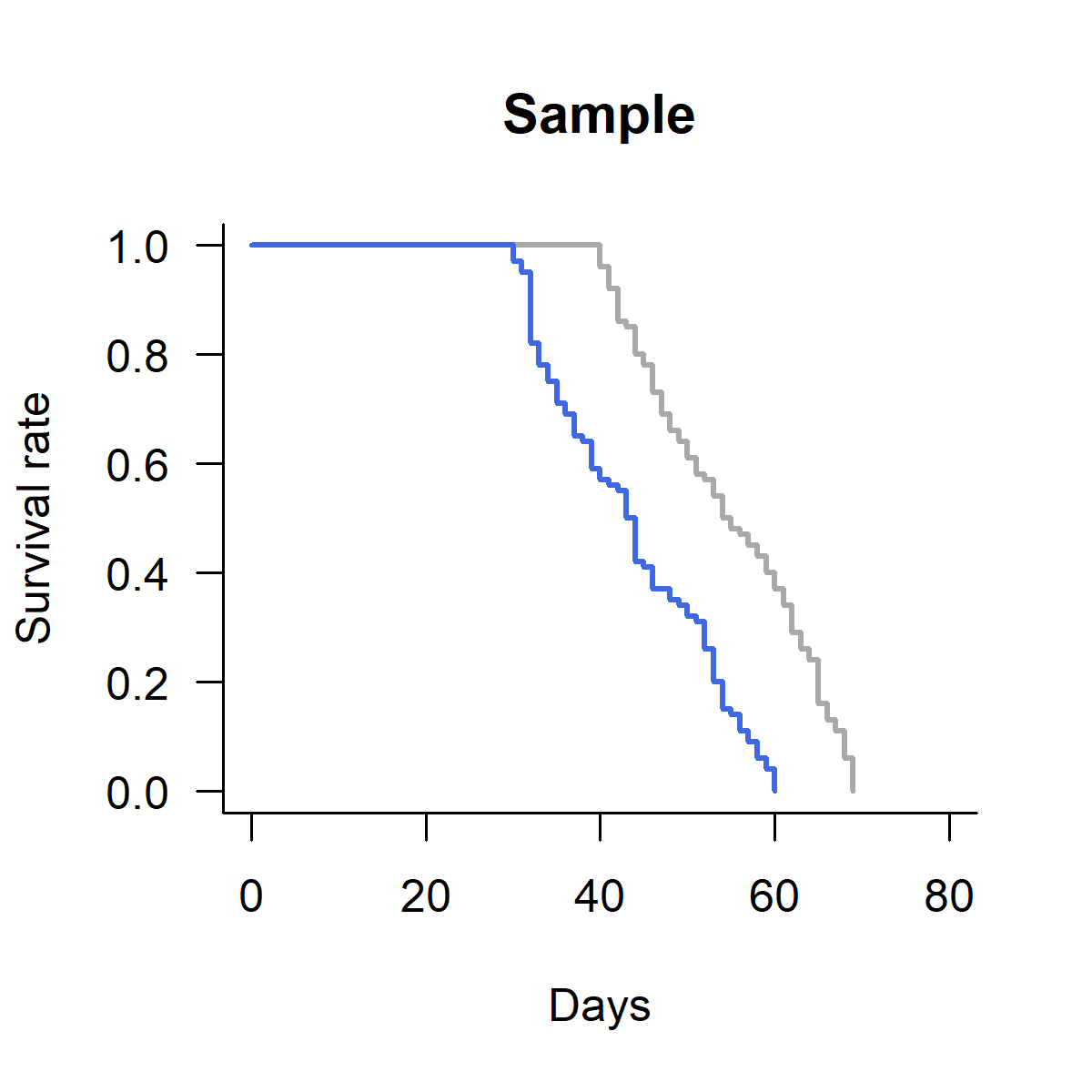
いわゆるカプラン・マイヤー曲線を描くことができた。
描画はグループ名のアルファベット順に行われるので、グラフの色を指定する順番に注意する必要がある。上ではdarkgreyがA群、royalblueがB群のグラフである。
次に凡例を加える(図2)。ここでは凡例の表示順をコード内で指定しているので、色の順番はそれに合わせればよい。
legend("bottomleft", legend = c("A", "B"), bty = "n", lty = 1, lwd = 3, cex = 1.0, col = c("darkgrey","royalblue"))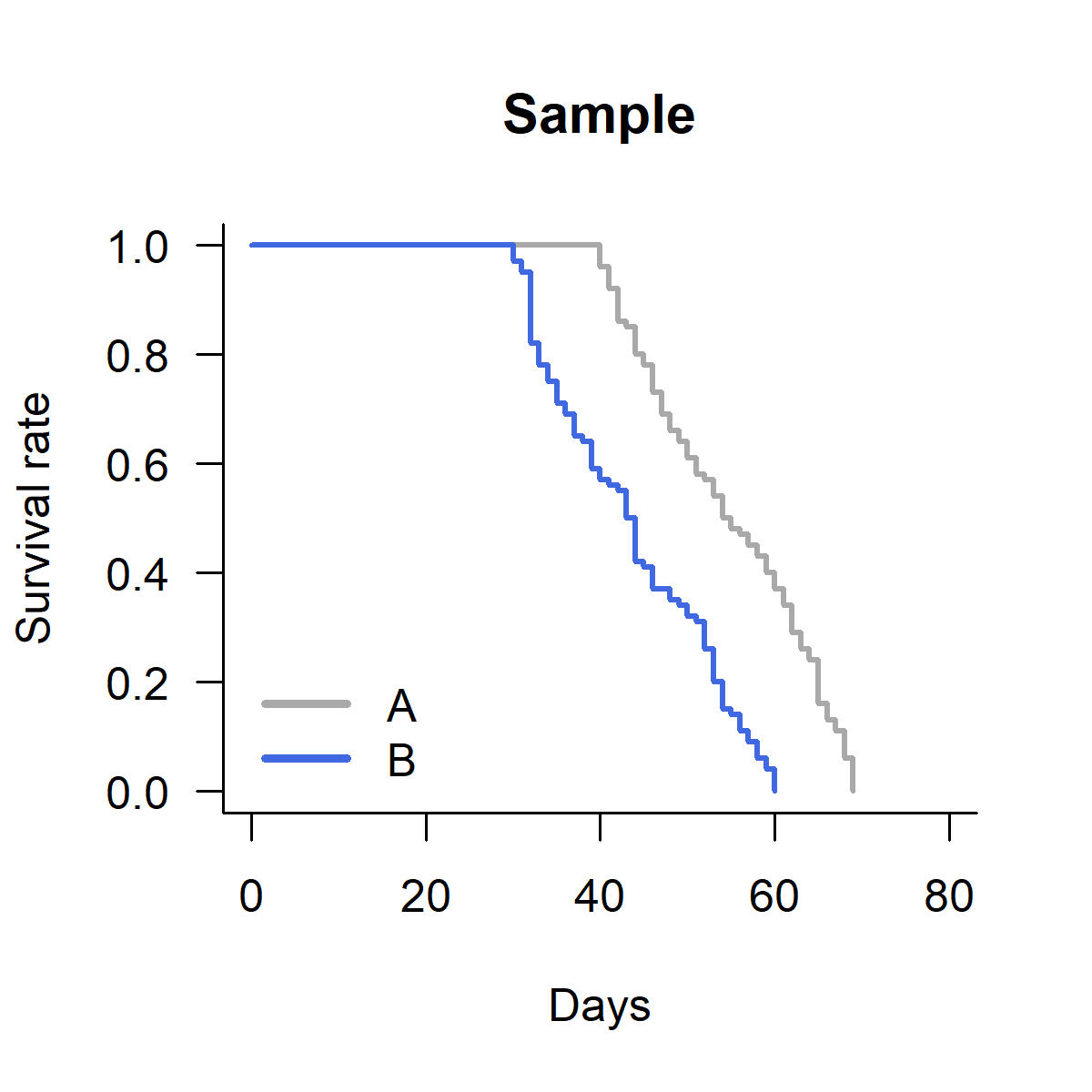
有意差があることをグラフ中に示すには、凡例の部分に線を引いてアスタリスクを追加する方法がある(図3)。
(※このコードを実行しても、Rの画面上では図3のようには見えず、ずれて表示されると思われる。これは画面上の表示と出力後の画像の見え方が必ずしも一致しないという仕様に因る。)
segments(21, 0.05, 21, 0.17)
text(x = 30, y = 0.11, "****", cex = 2)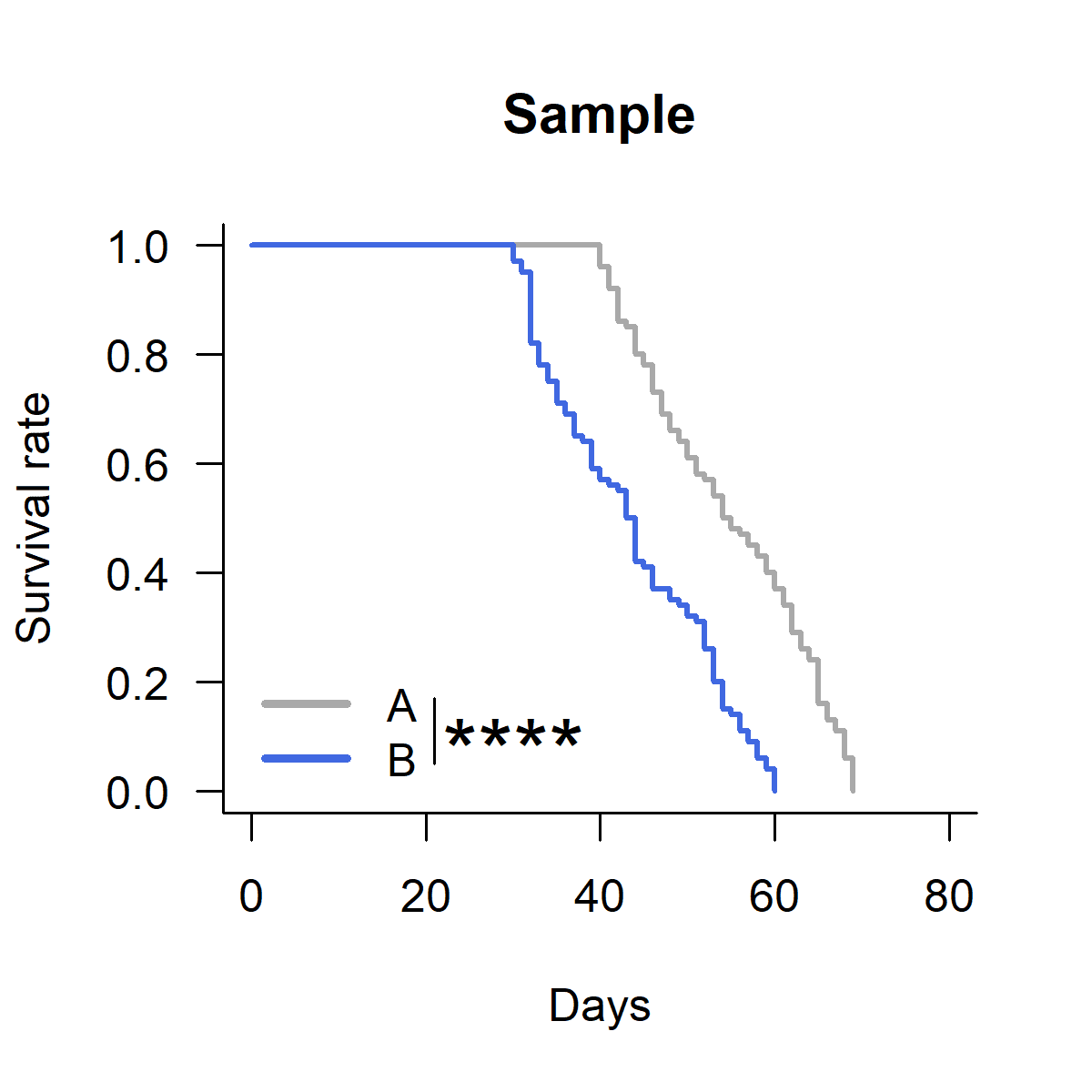
ただし、画像として出力した際に凡例のサイズや位置がRの画面上と異なることが多く、調整する手間がかかる。そのため、余白に単にp値を書いたほうが楽だし、2群間比較であれば差があることが十分に伝わる。その例は下のようになる(図4)。※図3のときのsegmentとtextは用いず、図2の上に重ねている
text(x = 0, y = 0.5, "p < 0.0001", cex = 2)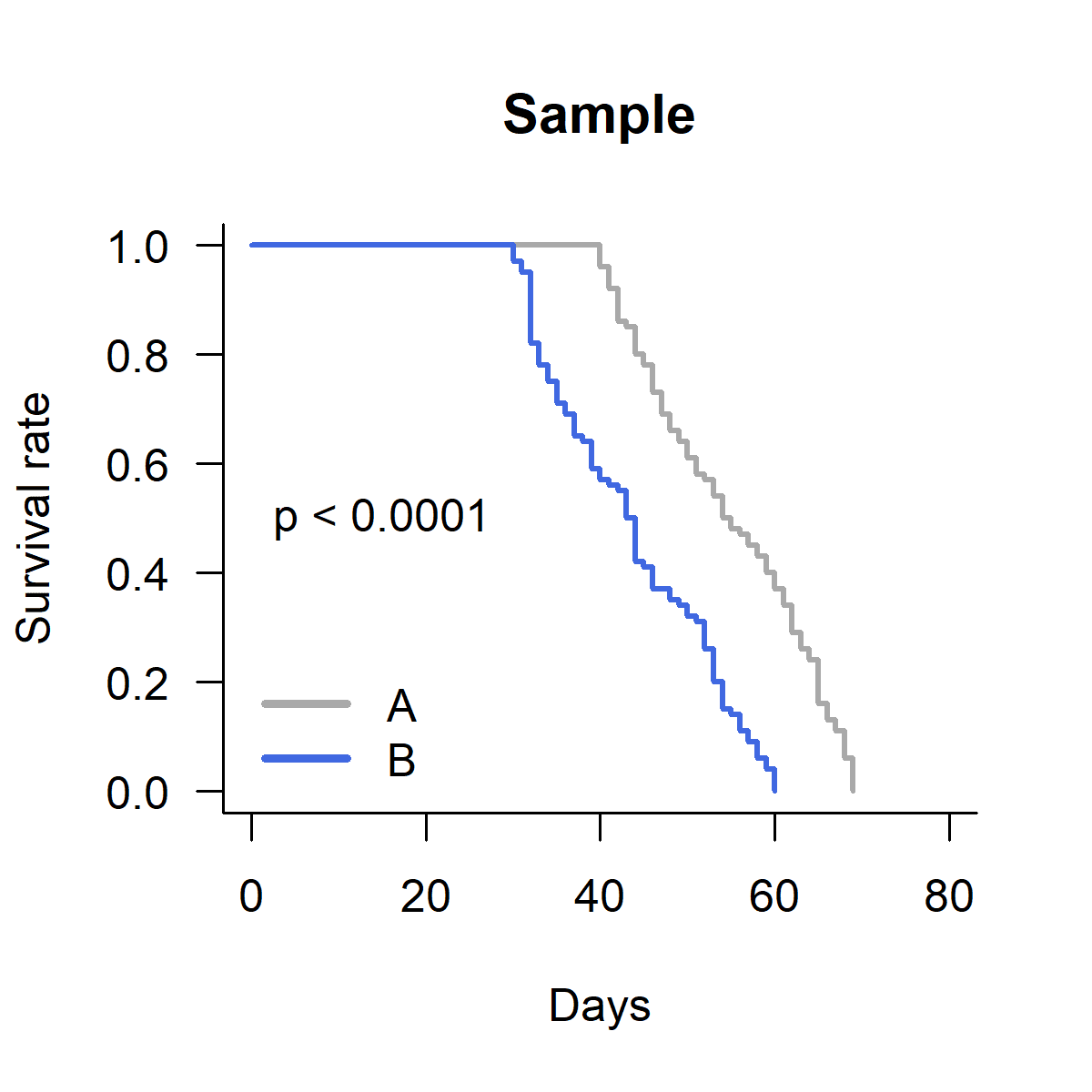
画像として出力する際は、次のようなコードを用いる。
プロット描画のコードを画像出力コードで挟んだ形になっている。
png("Sample.png", width = 1200, height = 1200, res = 300)
plot(survfit, main = "Sample", las = 1, lwd = 2, bty = "L", xlim = c(0,80), xlab = "Days", ylab = "Survival rate", col = c("darkgrey", "royalblue"))
legend("bottomleft", legend = c("A", "B"), bty = "n", lty = 1, lwd = 3, cex = 1.0, col = c("darkgrey","royalblue"))
segments(21, 0.05, 21, 0.17)
text(x = 30, y = 0.11, "****", cex = 2)
dev.off()これで、図3と同じものが出力されたはずである。
おわりに
以上で、Rを用いて生存曲線を描画することができた。本記事が読者の研究活動の一助となれば幸いである。
本記事では、グラフ作成を簡便に行うために最低限の情報のみを記載した。より詳細な解析方法などは専門の書籍や他の方のブログ等を参考にされたい。
