【概要】Structural biology in the time of COVID-19: perspectives on methods and milestones /コロナ禍における構造生物学:方法、出来事からの観点
本論文は、IUCrJ 国際結晶学連合(IUCr)が発行するフルオープンアクセスの査読付きジャーナルに2021年3月18日付で承認され、同年4月13日にアクセプトされている。
https://journals.iucr.org/m/issues/2021/03/00/mf5052/
【概要】
重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)によるCOVID-19パンデミックを構造生物学的観点から読み解く。
①構造解析法
タンパク質の構造解析における主な手法は2種類、高分子X線結晶構造解析(MX)と低温電子顕微鏡(CryoEM)。
・高分子X線結晶構造解析(MX)とは、
結晶化したタンパク質はX線に照射して、散乱されたX線を観測することで、物質の中の電子の分布、すなわち、物質の3次元構造を知る手法。多くのタンパク質構造を得るために全機械化のハイスループットスクリーニングなどの方法もある。
・低温電子顕微鏡(CryoEM)とは、
透過型電子顕微鏡法の一種で、試料を染色せずに、液体窒素程度の温度で凍結させることで「固定」し観察、解析する手法。
一般的に、低分子量の生体分子は主にMX法で解かれ、高分子量のものは主にCryoEM法で解かれている。
SARS-CoV-2の最初のゲノム配列が公開(Wu et al.、2020)され、その1週間後にメインプロテアーゼの3次元構造がMX法で解かれた。

https://www.nature.com/articles/s41586-020-2223-y.pdf (Jin et al.、2020)
最初のCryoEM構造はスパイクプロテインで、ゲノム配列公開、数週間後に、Protein Data Bank(PDB)に登録された(PDBエントリー6vsb; Wrapp et al.、2020年)。
さらには構造の視点からウイルスはどのような仕組みなのか、ウイルスタンパク質とヒトの宿主タンパク質とはどのように相互作用するか、医薬品になり得るリガンドとウイルスタンパク質が結合するか、それは果たしてどう結合するのかを理解するのに重要な学問と言える。
例えば、ワクチン開発の主要なターゲットとなるスパイクプロテインと、ヒトACE2受容体との複合体やヒト抗原体との複合体の構造的アプローチを行っている。

https://www.nature.com/articles/s41586-020-2852-1.pdf (Barnesら、2020年)
(Shangら、2020年、Wallsら、2020年、Wrappら、2020年、Yuanら、2020年 etc.)
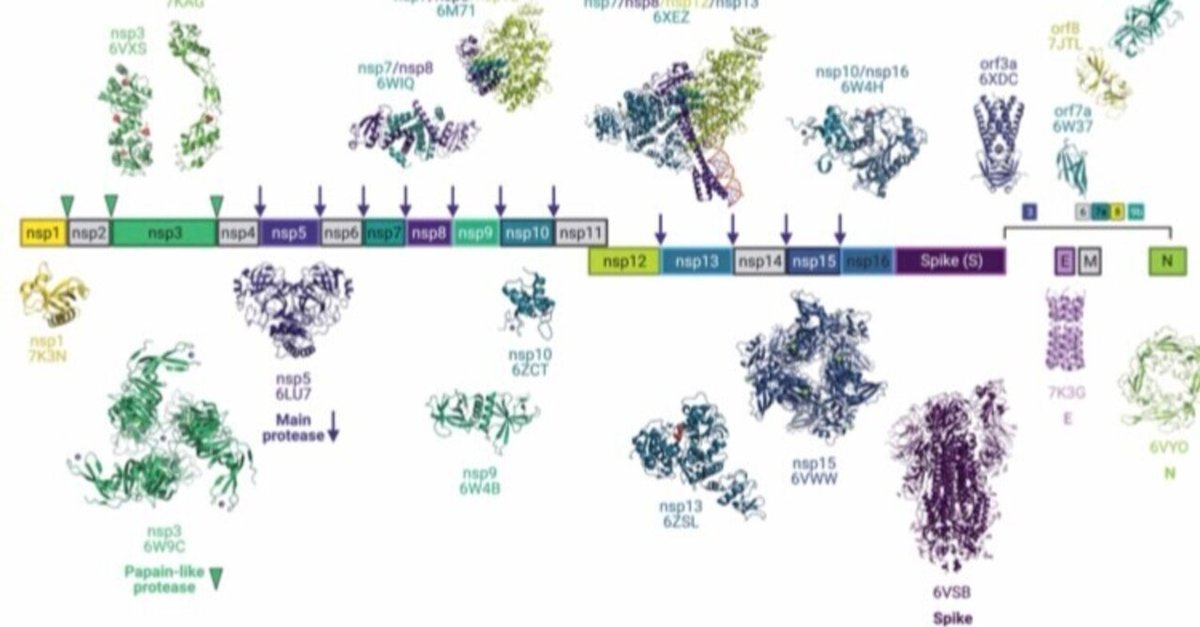
他にもターゲットとなり得るタンパク質が存在する。それは、コロナウイルスでは最長の遺伝子orf1a,b内に16の非構造タンパク質(nsp1-16)がコードされており、その内のパパイン様プロテアーゼ(nsp3)とメインプロテアーゼ(nsp5)という2つのシステインプロテアーゼが前駆体として機能し、宿主細胞からSARS-CoV2を複製、転写、ウイルスの組み立てを行う役目がある酵素や、RNAポリメラーゼ、ヘリカーゼ、エンドヌクレアーゼといったタンパク質も治療薬開発として、さらには阻害剤やフラグメントの結合を構造学から解析するというなんとも魅力あるターゲットとなる。

https://www.nature.com/articles/s41467-020-18709-w.pdf (Douangamath et al.、2020年)
(Gao et al, 2020年;Gordonら、2020年;Jinら、2020年;Kneller、Phillips、O'Neillら、2020年;Newmanら、2021年;Shinら、2020年;Schullerら、2021年;Yinら、2020年;Zhang、Linら、2020年、Newmanら、2021年、Schullerら、2021年 etc.)
論文やPDBから見ても、COVID-19パンデミックを受けて、SARS-CoV-2の研究に劇的に舵を切ったことを示す説得力のある数字を出している。
1年が経過した時点で、SARS-CoV-2に関する構造情報の77%はMXから、23%はCryoEMから得られており、ウイルスの標的タンパク質とagonist、antagonistのリガンドとどのように相互作用するかは、91%をもMXから構造が得られている。
②シュミレーション法
SARS-CoV-2の各種タンパク質の構造は、①の実験的手法によって得られる。しかし、創薬やワクチン開発の基盤となる原子レベルの詳細を明らかにするためには、in silico構造学も不可欠だ。
初期に公開されたゲノム(Wu et al.、2020)から、ソフトウェア C-I-TASSERやDeepMind社のAlphaFoldを活用し、迅速に、タンパク質構造予測の作成を行った。

https://zhanglab.dcmb.med.umich.edu/papers/2020_2.pdf (Zhang, 2020)
(Zhang, Zheng et al., 2020; Jumper et al., 2020; Senior et al., 2020 etc.)
このような分子動力学シミュレーションは、実験的な構造データの有用性を大幅に向上させている。
例.1
SARS-CoV-2の主要なウイルスタンパク質に対するin silico変異導入の迅速な解析
(Casalino, Dommerら、2020年、Rynkiewiczら、2021年、Sheik Amamuddyら、2020年)
例.2
スパイクプロテインのグリコシル化のモデリング
(Casalino, Gaiebら、2020年、Wooら、2020年)
例.3
宿主-病原体間のタンパク質相互作用(HPI)の可能性を探るために、複数のHPIデータベースを活用して、ネットワークベースおよび機械学習ベースで推定相互作用の評価
(Dey et al.、2020年、Messina et al.、2020年)
例.4
タンパク質の動的なふるまいをシミュレートすることで、様々な疾患に対する新しい治療法を開発できるのを支援するのを目的とした分散コンピューティングプロジェクトFolding@Homeでは、クラウドソーシングを利用してエクサスケールのコンピューティングリソースにアクセスし、複数のSARS-CoV-2シミュレーションプロジェクト
(Achdout et al.2020; Zimmerman & Bowman, 2021)
①構造解析法と②シュミレーション法の考察
実験手法と計算モデルの相乗効果によって、SARS-CoV-2ウイルスのメカニズムの詳細を解明する上での基本的な進歩がもたらされたのはもちろん、特にタンパク質のダイナミクス、グリコシル化、タンパク質の相互作用研究の分野で、両方のアプローチを前進させました。
しかし、SARS-CoV-2のタンパク質構造には未解決のものがあり、また新たに出現した亜種もあるため、構造生物学コミュニティの継続的な関心と技術が必要とされている。
