
ろう・難聴脳データベース研究のMRI解析プロトコール
Surface-Basedの機能的結合解析
機能的結合、Resting-state fMRIとは
機能的結合は、異なる場所で発生する神経活動が同期する現象を指します。2つの異なる脳領域での活動が統計的に関連している場合、これらの領域は機能的に結合していると解釈されます。この考え方の背景には、脳の異なる領域が同時に活動することで、一つの機能やネットワークを形成していると考えられます。これらの脳ネットワークは、脳内の血流の変化を通じて観察され、これにより血中酸素レベル依存(BOLD)信号が生じ、fMRIを使用して測定することができます。脳の活動は固有のものであり、外部からのタスクがない場合でも存在します。Resting-state fMRIは、明確なタスクを実行していない休息状態やタスク非実行状態でのBOLD信号の自発的な変動を測定し、脳の領域間の相互作用(機能的結合)を評価します。
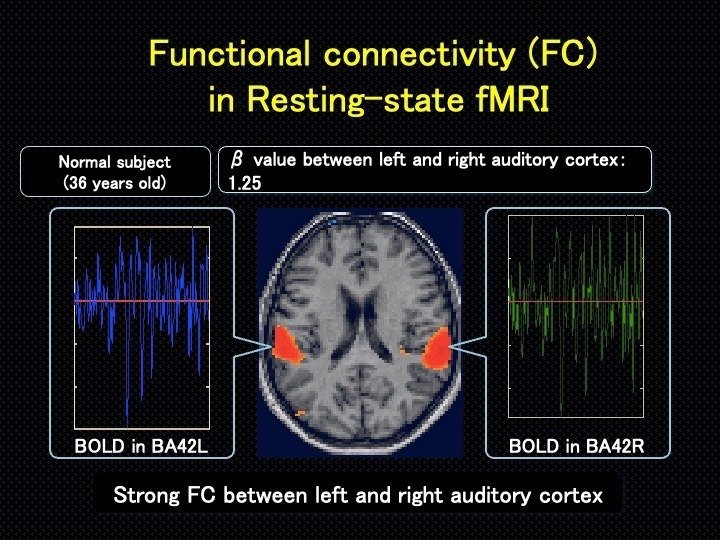
上の図は、10年前の私の脳のResting-state fMRIです。左右の聴覚野のBOLD信号の揺らぎが、統計学的に強い相関を示し同期していることがわかります。つまり左右の聴覚野は、強い機能的結合があると言えます。また、聴覚関連領域の機能的結合は、年齢で変化すること、耳鳴患者さんでは聴力が正常であっても低下することを報告しています。

聴覚関連領域の機能的結合が年齢とともに変化することを報告

耳鳴患者さんでは、健聴であっても、聴覚関連領域の機能的結合が低下していることを報告
Surface-based解析とは
画像は特定の長さ、幅、高さで取得された個々のボクセル(Voxel)を持ちます。下の図のように、2つの近接した脳回は、Voxel-basedだと数ミリメートルしか離れていないかもしれませんが、一つの脳回から脳溝に下り、もう一つの脳回に上がる実際の距離は、それの何倍も長いことになります。この距離は、画像を表面空間に再構築することでより正確に表現されます。これを、Surface-based解析と言います。

ろう・難聴脳データベース研究では、T1強調画像とResting-State fMRI画像を用いて、Surface-basedで機能的結合解析を行なっています。
MRI撮像プロトコール
MRI撮像プロトコールは「革新脳・脳プロ 脳3T-MRI標準プロトコルガイドライン Ver1.0 2015.4.26」を用いています。T1強調画像とResting-state fMRIを撮像します。
T1強調画像

Resting-state fMRI

解析プロトコール
必要なアプリケーション
Surface-Basedの機能的結合解析を行うためには、以下の主要なアプリケーションが必要です。これらのアプリケーションは、MRIデータの前処理、解析、および可視化をサポートします。それぞれのツールは、特定のタスクや解析のステップに特化しており、一緒に使用することで、複雑なfMRIデータの解析を効果的に行うことができます。
MRIcroGLは、画像の表示、編集、および変換のためのソフトウェアツールです。dcm2niixが組み込まれており、DICOM形式の画像をNIfTI形式に変換する機能が備わっています。
FreeSurferは、構造MRIデータから脳の皮質表面を再構築するためのソフトウェアツールセットです。このツールは、脳の皮質の厚さやボリュームを計測するためにも使用されます。FreeSurferは、脳の各領域の自動セグメンテーションや、表面ベースの統計解析もサポートしています。
CONNは、fMRIデータの機能的および構造的結合解析を行うためのMATLABベースのツールボックスです。CONNは、前処理、第一レベルおよび第二レベルの統計解析、および結果の可視化をサポートしています。
MATLABは、数値計算とプログラミングのための高性能な言語と環境です。CONN、SPMは、MATLAB上で動作します。
FSLは、機能的および構造的脳画像解析のためのソフトウェアライブラリです。FSLには、前処理、統計解析、および結果の可視化のための多くのツールとユーティリティが含まれています。
SPM (Statistical Parametric Mapping)
SPMは、fMRI、PET、およびMRIデータの統計的解析を行うためのMATLABベースのソフトウェアパッケージです。SPMは、前処理、統計モデリング、および結果の可視化をサポートしています。
Step 0: DICOMからNIfTIへ
DICOM画像のソート(必須ではないが、DICOMを撮像条件順にソートできる)
dcm2niix -r y [出力ディレクトリ] [DICOMファイルが格納されているディレクトリ]dcm2niixの実行
dcm2niix -o [出力ディレクトリ] [DICOMファイルが格納されているディレクトリ]fMRIの複数のNIfTI画像を一つのファイルにまとめる(必要があれば)
fslmerge -tr [出力ファイルの名前] [連結する入力ファイル(ワイルドカード*を使用して複数のファイルを指定する)] [TR(Repetition Time)今回は2.5]
Step 1: FreeSurferでRecon-all
recon-all -s <subject_name> -i [解析するT1強調画像MRI] -all
Step 2: Surface-basedデータをCONNに読み込む
Matlabターミナルを開き、CONNツールボックスを開く。
Setup > Structuralで、先ほどのrecon-allで作成したSurface-basedデータ(/mri/T1.mgz)をImportする。
Setup > Functionalで、Resting-state fMRIデータをImportする。
Setup > ROIsで、FreeSurferのROI(/label/lh.aparc.annot)を読み込む。
Step 3: 前処理(Preprocessing)の実行
Setup > Optionsで、Analysis space (voxel-level)をSurface: same as template (Freesurfer fsaverage)に変更する。
Preprocessingで、preprocessing pipeline for surface-based analyses (in subject-space)を選択する。using nonlinear coregistrationのオプションをつけた方が、より正確なリサンプリングを生成するが、時間がかかる。解剖学的画像はすでにFreeSurferによってセグメント化されており、セグメンテーションがインポートされているので、structural Segmentation (Grey/White/CSF tissue estimation)は削除する。
前処理終了後、ROIsセクションに戻り、前で読み込んだFreeSurferのROIを選択。Compute average timeseriesの下で、secondary dataset #4 (surface-space data)を選択する。
上記をすべて終了したら、Setupパイプラインを実行するためのDoneボタンをクリックする。

主に次の2つのサイトを参考にして解析を行なっています。
K-Lab:分担研究者に入っていただいている筑波大学医学医療系精神医学の根本清貴先生のブログ。
Andy’s Brain Book!:Michigan大学のAndrew Jahn先生のブログ。
ろう・難聴脳データベース研究の参加者を募集しています!
一緒に共同研究をやってくださる施設や先生方も募集しています!

