皮膚表面の細菌DNAは生存可能な皮膚マイクロバイオームを過大評価する
微生物学と感染症
皮膚表面の細菌DNAは生存可能な皮膚マイクロバイオームを過大評価する
https://elifesciences.org/articles/87192?utm_source=twitter&utm_medium=social&utm_campaign=organic
エレン・M・アコスタ
キャサリン・A・リトル
ゼマー・ギタイ
他 著者リストを展開する
プリンストン大学分子生物学科(米国
プリンストン大学ルイス・シグラー統合ゲノミクス研究所(米国
バンダービルト大学メディカルセンター病理学・免疫学・微生物学教室(米国
バンダービルト感染・免疫・炎症研究所(アメリカ合衆国
ペンシルバニア大学皮膚科(米国
他、著者リストを展開
2023年6月30日
https://doi.org/10.7554/eLife.87192
オープンアクセス
著作権情報
記録版
著者らは、このバージョンの論文を「Version of Record」であると宣言する。
イーライフのプロセスについて
ダウンロード
論文または論文の一部を様々な形式でダウンロードするためのリンクの2部構成のリストです。
ダウンロード(論文をPDFでダウンロードするためのリンク)
論文PDF
図表PDF
引用文献を開く(この記事の引用文献を様々なオンライン参考文献管理サービスで開くためのリンク)
メンデレー
この論文を引用する(様々なレファレンス・マネージャー・ツールと互換性のあるフォーマットでこの論文からの引用をダウンロードするためのリンク)
エレン・M・アコスタ
キャサリン・A・リトル
ベンジャミン・P・ブラットン
ハイメ・G・ロペス
毛暁明
エイミー・S・ペイン
モハメド・ドニア
ダネル・デベンポート
ゼマー・ギタイ
(2023)
皮膚表面の細菌DNAは生存可能な皮膚マイクロバイオームを過剰に表現する
eLife 12:RP87192.
https://doi.org/10.7554/eLife.87192
BibTeXをダウンロード
.RISをダウンロード
引用
共有
コメントオープンアノテーション(現在このページには0件のアノテーションがあります)。
記録の公開バージョン
2023年6月30日(本バージョン)
全文
図表
概要
皮膚マイクロバイオームはヒトの健康に不可欠な役割を果たしている。しかしながら、その構成細菌の空間的な組織と生存率は不明なままである。ここでは、ヒトおよびマウスの皮膚サンプルに培養、画像化、および分子生物学的アプローチを適用し、皮膚表面には、細菌のDNA量から予測されるよりも少ない数の生菌がコロニー形成していることを見出した。その代わりに、皮膚に存在する生菌は、毛包やその他の皮膚浸潤部に多く存在している。さらに、皮膚マイクロバイオームは、他のヒトマイクロバイオーム部位と比較して、生存細菌の割合が特異的に低いことを示し、皮膚表面のほとんどの細菌DNAが生存細胞に関連していないことを示した。最後に、ヒトボランティアを用いてin vivo皮膚マイクロバイオーム摂動-回復研究を行った。細菌16S rRNA遺伝子配列解析の結果、皮膚マイクロバイオームは積極的な摂動を受けても驚くほど安定している一方で、皮膚表面の再増殖は、根底に存在する生存集団によって促進されることが明らかになった。皮膚表面の細菌DNAは一過性に擾乱を受けるが、その下層に存在する安定した生菌集団によって補充されるため、今回の研究結果は、皮膚マイクロバイオームの擾乱のダイナミクスを説明する一助となる。これらの結果は、皮膚マイクロバイオームの生物学における複数の未解決の問題を解決するものであり、今後の研究と操作の取り組みに重要な示唆を与えるものである。
eLifeの評価
この重要な研究において、著者らは、皮膚細菌を対象とした現在のDNAベースの微生物ゲノム解析では、配列決定されたDNAの出所や、それが生菌由来なのか非生存菌由来なのかを常に検出することはできないという説得力のある証拠を示している。さらに著者らは、ヒトとマウスにおいて、生菌のほとんどが皮膚表面ではなく毛包内に存在することを実証した。全体として、この研究は一つの学問分野を超えた意義があり、マイクロバイオームを研究する人々にとって興味深いものとなるだろう。
https://doi.org/10.7554/eLife.87192.2.sa0
eLifeの評価について
はじめに
皮膚は人体最大の臓器であり、宿主と微生物の潜在的相互作用におよそ25平方メートルを提供する(Gallo, 2017)。皮膚は、世界との触覚的相互作用を促進し、日常生活の危険から私たちを分離し、20~30日ごとに自己再生するという驚くべき能力を持っている(Maeda, 2017)。ヒトの皮膚には、皮膚マイクロバイオームを構成する生物も生息しており、ヒトの健康に重要な役割を果たすことが明らかになっている。例えば、ヒトの皮膚マイクロバイオームは、免疫系の教育(Scharschmidtら、2015;Polak-Witkaら、2020)、創傷治癒(Kalanら、2019;Kimら、2019;Loescheら、2017)、コロニー形成抵抗性(Byrdら、2018)、皮膚における遺伝子発現の調節(Meiselら、2018b)に影響し、発生に関与している可能性がある(Meiselら、2018a)。ヒトの健康に対する皮膚マイクロバイオームの多くの貢献にもかかわらず、従来の方法では対処されていない重要な疑問がある: 縦断的研究において、皮膚マイクロバイオームが数カ月にわたって安定しているにもかかわらず、水泳のような一過性の環境変化で容易に乱されるのはなぜか(Nielsen and Jiang, 2019)。なぜ空気にさらされる臓器には嫌気性細菌が多いのか?また、擦過傷のような強い摂動がなければ、新しい微生物で皮膚を安定的にコロニー化することが難しいのはなぜだろうか?皮膚マイクロバイオームを構成する細菌のサンプリング、培養、配列決定を行う従来の方法は、皮膚マイクロバイオーム分野の確立に不可欠であった。ここでは、これらの重要な疑問の解決に着手するため、生存可能な細菌と生存不可能な細菌を区別するためのアプローチを追加し、これらの基礎研究を拡張する。
研究結果
蛍光in situハイブリダイゼーションでは、皮膚表面の細菌はほとんど検出されなかった。
皮膚マイクロバイオーム研究の主な方法は、綿棒で皮膚から採取したDNAの塩基配列を決定することである(Huttenhower et al.) しかし、皮膚マイクロバイオームはしばしば皮膚表面の微生物がよく混ざったコーティングとして描かれるにもかかわらず(Byrdら、2018;Grice and Segre、2011)、皮膚表面からのDNAが皮膚マイクロバイオームの根本的な生物学を反映しているかどうかは依然として不明である。さらに、シークエンシングや培養に基づく研究では、細菌が皮膚の深部まで広がっていることが示されているが、皮膚マイクロバイオーム全体としての空間分布はよく特徴付けられていない(Polak-Witkaら、2020;Belkaid and Tamoutounour、2016;Lousadaら、2021)。このギャップを解決するために、我々は細菌16S rRNAにハイブリダイズする万能細菌蛍光in situハイブリダイゼーション(FISH)プローブEUB338を用いて、皮膚における細菌細胞の空間分布を決定した(Amann et al.)
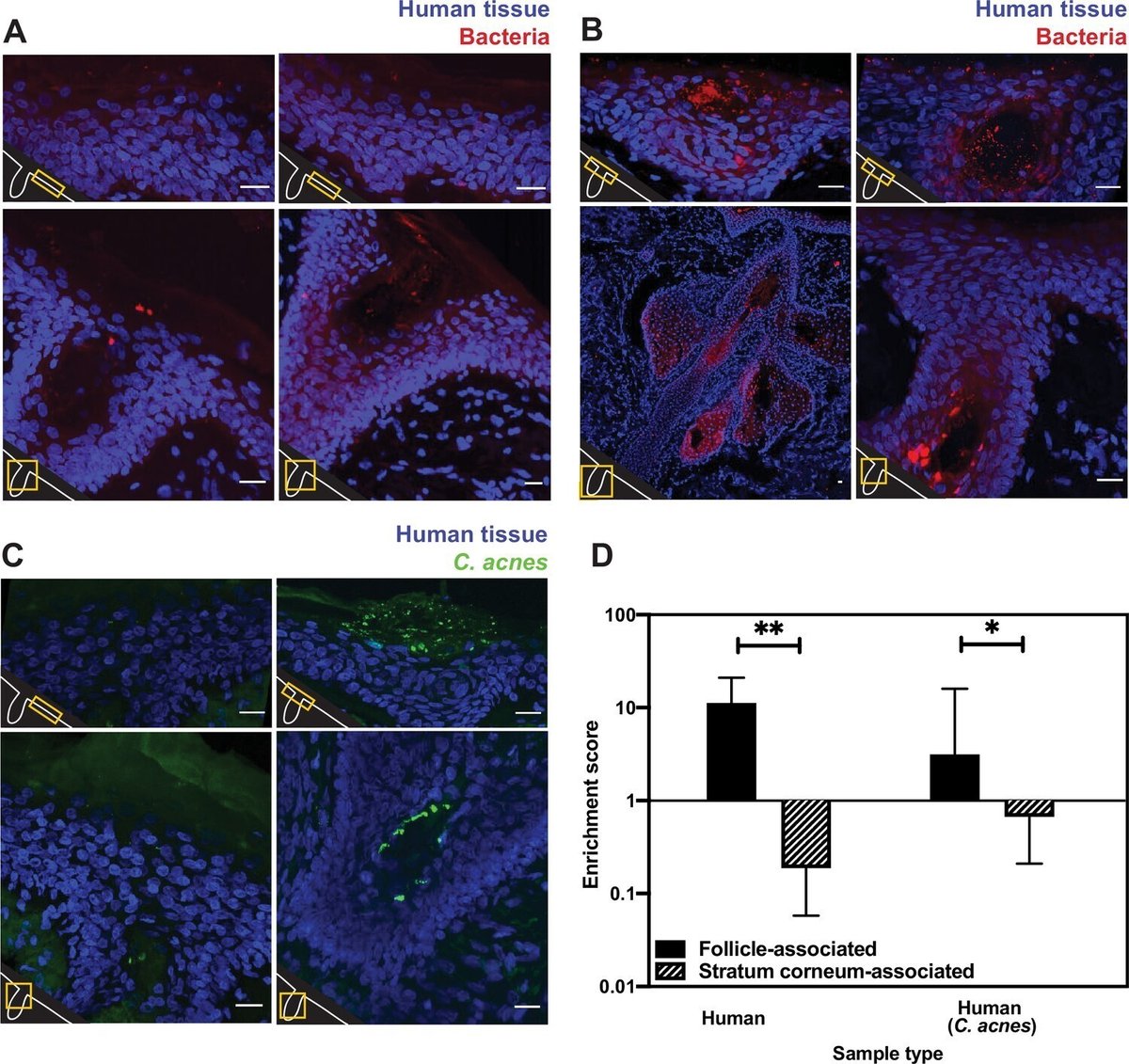
我々はまず、生検した健康な成人ヒト顔面組織でEUB338 FISHを使用し、皮膚表面には細菌がほとんど存在しないことを発見した(図1A)。対照的に、コメドのような毛包やその他の皮膚構造内には、細菌のクラスターが見られた(図1B)。皮膚表面にFISH染色がないことについては、いくつかの説明が可能である。例えば、生存可能な表面細菌が生検部位の滅菌によって除去された可能性がある(この可能性については後述する)。あるいは、表面細菌はEUB388ハイブリダイゼーション可能なリボソームが少ない定常期のような状態で存在するか、皮膚マイクロバイオーム細菌種がFISHプローブのハイブリダイゼーションを起こしにくくなっている可能性もある。しかしながら、複数の皮膚マイクロバイオーム細菌種(Cutibacterium acnes、Staphylococcus epidermidis、Micrococcus luteus、Corynebacterium striatum)の定常期細胞において、EUB338染色がうまく機能することが確認された(図1-図1)。
図1-補遺1
アセットをダウンロード アセットを開く
ヒト組織の細菌蛍光in situハイブリダイゼーション(FISH)染色。
(A-C)スケールバー=20μm。各図の左下は毛包の模式図を白で、各画像フレームの解剖学的位置を黄色で示す。DAPI染色は... もっと見る
図1-図1
アセットをダウンロードする アセットを開く
定常期の皮膚マイクロバイオーム細菌種の蛍光in situハイブリダイゼーション(FISH)。
すべての細菌種を適切な条件で定常期まで増殖させた。汎細菌FISHプローブEUB338を用いた。ハイブリダイゼーションは赤で示す。
ヒト皮膚のFISH染色を定量化するために、関心領域(毛包、皮膚表面、その他の皮膚構造)内の平均蛍光と関心領域外の平均蛍光の比を計算した。この比率を濃縮スコアと呼ぶ。ヒト毛包の濃縮スコアの中央値は11.24であった(図1D)。一方、皮膚表面(角質層)の濃縮スコアはわずか0.188であった。毛包では多くの細菌が観察されるが、皮膚表面ではほとんど観察されないというこのパターンは、ヒトの皮膚マイクロバイオームで最も豊富な細菌種の一つであるC. acnesに特異的なFISHプローブを用いても同様であった(図1C)。C.acnesの濃縮スコアは、毛包では3.14であったのに対し、角質層では0.67であった(図1D)。無傷の皮膚関連菌の大部分は皮膚表面と関連していなかったが、皮膚表面、特に表層下構造に近い部分には目に見える細菌が存在することに注目したい(図1AおよびB)。このことは、FISH染色が皮膚表面の細菌を検出できることを示している。このように、今回の結果は、皮膚表面のぬぐい液から細菌を培養できるというこれまでの報告と一致しているが、さらにこれらの知見を拡張して、皮膚表面には深部の皮膚構造よりも無傷の細菌が少ないことを実証している。
皮膚表面の細菌DNAは生菌数を過大評価する
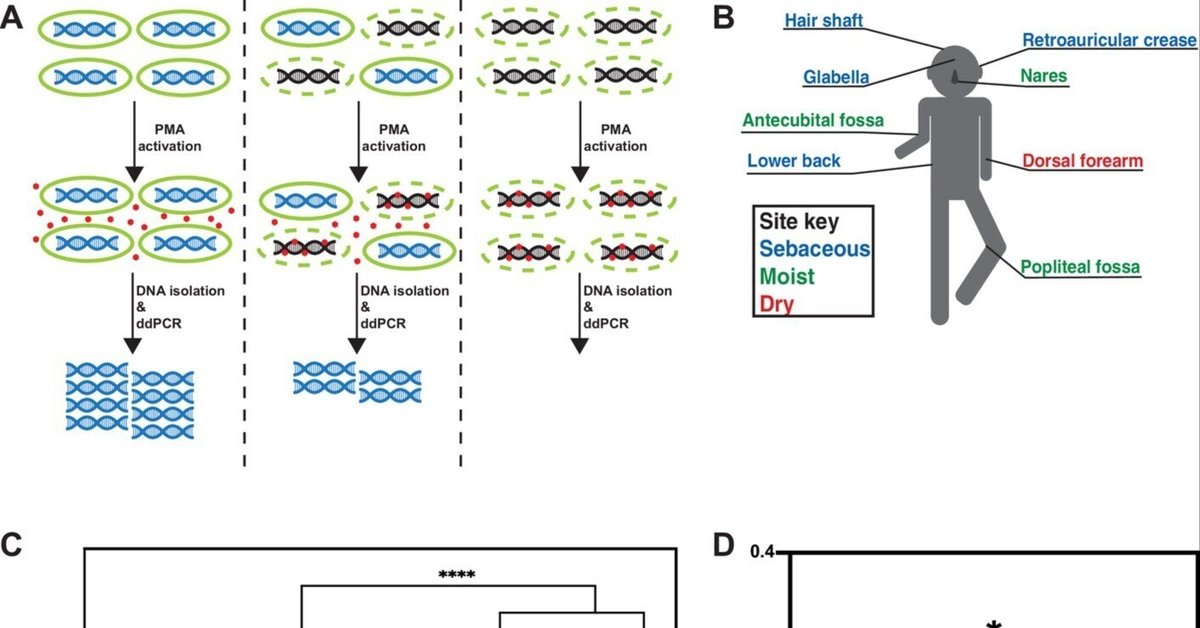
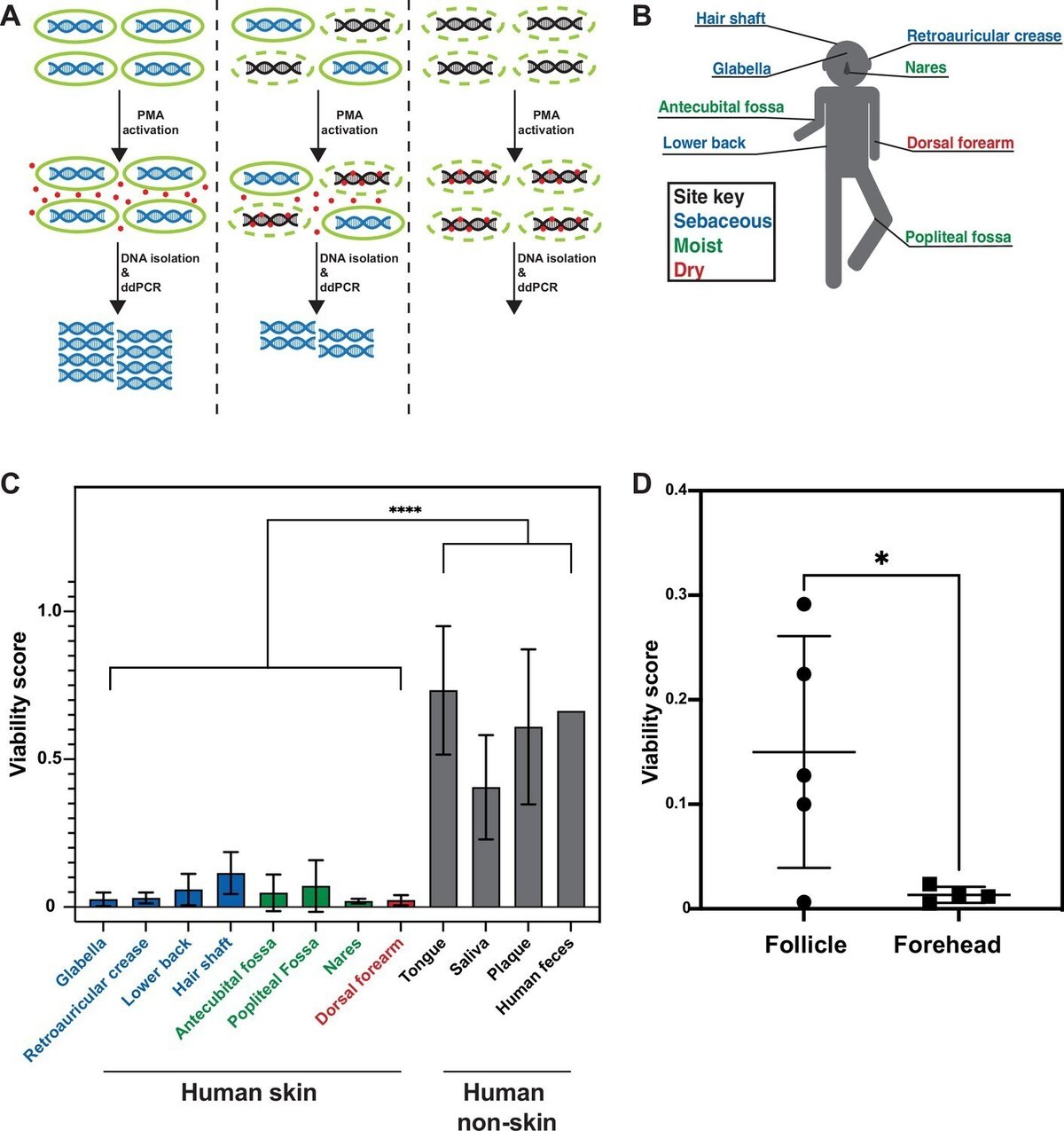
皮膚表面をスワブして採取したサンプルのDNA配列決定は、簡便で非侵襲的であり、他のサンプリング方法よりも高い一貫性が得られることが示されているため、皮膚マイクロバイオームの評価に用いられる最も一般的な方法である(Bjerre et al.) しかしながら、従来のメタゲノムおよび16S rRNA遺伝子配列決定では、生存可能な細菌と生存不能な細菌のDNAを区別することができない。そこで私たちは、サンプル中のDNAのうち、無傷の細菌に由来するDNAの割合を定量的に決定できる方法を導入した。生細胞と生存不能細胞を分子的に区別することができれば、未処理の生きた皮膚を用いた直交法を用いてFISHの結果を検証することができ、またFISHで観察された無傷の表面細菌の数が少なかったのは生検滅菌によるものであるという可能性に対処することができる。この目的のために、細胞不透過性の低分子プロピジウムモノアジド(PMA)を利用した。PMAは、光活性化によって二本鎖DNAに不可逆的に結合し、PCR増幅を阻害する(図2A;Nocker et al.) DNA単離の細胞溶解ステップの前にPMA光活性化を行うと、PMAは細胞不透過性であるため、生存細菌内のゲノムDNAはPMA結合から保護されるが、無細胞DNAや透過化細菌内のDNAはPMA結合となる。細菌のDNAを定量化するために、PMAと液滴デジタルPCR(PMA-ddPCR)を組み合わせた。PMAなしのサンプルとPMAありのサンプルとのddPCRカウントの比率を計算することで、任意の細菌集団の生存率スコアを作成することができました。同様のアプローチは、環境廃水サンプルからの細菌の生存性を評価するために使用されています(Yang et al.)
図2と2つの補足
アセットをダウンロード アセットを開く
プロピジウムモノアジド-液滴デジタルPCR(PMA-ddPCR)とヒト皮膚および非皮膚マイクロバイオームの生存率スコア。
(A)PMA-ddPCRワークフローの概略図。(B)サンプリングされた各皮膚部位を示すサンプリングスキーム。色は部位のタイプを示す(青は皮脂、緑は湿潤、赤は乾燥)。(C)皮膚上のPMA-ddPCR...もっと見る
図2-図2
アセットをダウンロード アセットを開く
プロピジウムモノアジド(PMA)を使用しない場合(A)と使用した場合(B)の20μL液滴デジタルPCR(ddPCR)反応あたりのコピー数。
(A)と(B)のデータは図2に示した生存率スコアの算出に使用した。エラーバーは標準偏差を表す。
図2-図1
アセットをダウンロード アセットを開く
Propidium monoazide-droplet digital PCR(PMA-ddPCR)とサンプリングコントロール。
(A) 熱キルした大腸菌培養液と指数関数的に増殖した大腸菌培養液の既知の比率でのPMA-ddPCR検証コントロール。指数関数的に増殖する細胞集団に対してPMA-ddPCRを実施した結果、... さらに表示
PMA-ddPCRにより、集団中の生存細胞の割合を確実に測定できることを確認するため、まず、指数関数的に増殖する大腸菌培養物または定常期の大腸菌培養物と熱キルした大腸菌培養物との既知の比率を用いて、PMA-ddPCRが期待される結果を生成することを検証した(図2-図1A)。PMA-ddPCRが皮膚常在細菌種にも有効かどうかを調べるため、最も一般的な皮膚マイクロバイオーム細菌の4種、S. epidermidis、C. acnes、M. luteus、およびC. striatumにもPMA-ddPCRを適用した。各細菌培養の連続希釈を行い、PMAの有無にかかわらず、コロニー形成単位(CFU)プレーティングとddPCRの両方を並行して行った。いずれの場合も、サンプル中のDNA量(PMA-ddPCRによる評価)は、細菌量の多いシナリオでも少ないシナリオでも、培養可能な細菌数とよく相関していた(図2-図1B)。これらの結果から、PMA-ddPCRは無傷の細菌内に存在するサンプル中のDNA量を評価する信頼性の高い方法であることが確認されました。
次に、健康なヒトボランティア4人の皮膚を8箇所(口唇、後口唇溝、腰、毛幹、前肩甲骨窩、膝窩、鼻腔、前腕背)綿棒で採取し、PMA-ddPCRをヒト皮膚マイクロバイオームサンプルに適用した(図2B、図2-図2AおよびB)。PMA-ddPCRの結果、これらの部位の生存率スコアは0.02~0.12(0は完全に生存していない集団、1.0は完全に生存している集団)であり、皮膚表面で見つかった細菌DNAの大部分は生存している細胞とは関連していないことがわかった(図2C)。これが皮膚に特異的な現象であるかどうかを調べるため、皮膚以外のマイクロバイオーム部位(舌、唾液、歯垢、糞便)をいくつかテストした。その結果、皮膚以外のすべてのマイクロバイオーム部位において、生存率スコアは皮膚よりも有意に高く、0.4(唾液)から0.87(糞便)の範囲であった(図2C)。
次に、PMA-ddPCRの生菌数スコアが皮膚表面の生菌数を正確に表しているかどうかを調べた。皮膚表面上の検出可能な細菌数が少ない(FISHまたはPMA-ddPCRのいずれかによって)ことは、皮膚表面が無菌であることを意味するのではなく、むしろ皮膚表面上の細菌DNAの大部分は、これらの生存細胞由来ではないことを意味する。実際、皮膚から直接細菌を培養することは一般的である(Byrd et al.) 生菌数を直接定量化するために、皮膚微生物培養の標準条件(好気的および嫌気的に培養したトリプシン大豆アガロースプレート中の5%ヒツジ血液)を用いて、各サンプルを少量ずつプレーティングした。PMA-ddPCRと従来のddPCRのどちらが皮膚マイクロバイオームの生菌数をよく表しているかを調べるため、S. epidermidisの既知菌数で作成した標準曲線と結果を比較した。各サンプルについて、PMA-ddPCRによる定量はこの標準曲線とほぼ一致した一方、PMAを欠くサンプルでは重複が見られなかった(図2-図1CおよびD)。最も存在量の多い皮膚マイクロバイオーム種は容易に培養可能であるため、皮膚表面の生菌からDNAを定量することで、プレーティング時のCFUを正確に予測できるはずである。PMAを使用せずに皮膚マイクロバイオームサンプル中の細菌DNAを定量した結果、DNA量は平均して標準曲線による予測値の82倍となったが、PMAを使用することでこの値はわずか1.3まで低下した(図2-図1E)。また、CFU を予測するために ddPCR カウントを使用した場合、PMA 非存在下での ddPCR では、CFU カウントが実測値の 58.5 倍になると予測されたのに対し、PMA-ddPCR では、CFU カウントが実際の培養 CFU の平均 1.28 倍になると予測された(図 2-図 1F)。この結果は、PMA-ddPCR の方が PMA を含まないアプローチよりも生菌数をよく反映しているという結論をさらに裏付けるものである。
生検された皮膚から得られたFISHの結果から、生菌は皮膚表面下で保護されている可能性が示唆された。未処置の皮膚でこの仮説を検証するため、顔面毛包内容物を採取し、PMA-ddPCRを用いて顔面毛包内容物と皮膚表面サンプルの生存率を比較した(図2D)。毛包内容物の平均生存率は皮膚表面よりも10倍以上高く、毛包内の細胞は皮膚表面の細胞よりも生存率が高いことが確認された。生検のFISHと皮膚ぬぐい液のPMA-ddPCRの結果を合わせると、皮膚表面には生存可能な細菌細胞はほとんど存在せず、滅菌されていない健康なヒトの皮膚表面はまばらにコロニー形成されているという結論が裏付けられた。これらのデータは、皮膚表面の細菌DNAは主に生存細菌細胞とは関連していないことを示しているが、われわれの結果は、皮膚表面に生存細菌細胞が存在しないことを示唆しているわけではないことに留意されたい。むしろ、我々のデータは、皮膚表面の細菌DNAの大部分は細菌内には存在しないことを示しており、PMAを用いることで、生存可能な皮膚マイクロバイオームをより正確に推定できることを示している。
従来のシーケンス法は皮膚マイクロバイオームの豊富さと多様性を過大評価する
多くのマイクロバイオームと同様に、皮膚マイクロバイオームに関する既存の知見は、細菌16S rRNA遺伝子アンプリコンシーケンスに大きく依存しており、培養法によってもたらされるバイアスを回避しながら細菌集団を評価するために開発された。しかし、我々の知見によると、皮膚マイクロバイオームの研究に16S rRNA遺伝子アンプリコンシーケンスを用いても、サンプル中のDNAのほとんどが生菌由来ではなく、従来の16S rRNA遺伝子アンプリコンシーケンスでは生細胞由来か死細胞由来かの区別がつかないため、完全に偏りがないとは言えないことが示唆された。16S rRNA遺伝子アンプリコンシークエンシングがこれら2種類の細菌集団を区別できないことは、この方法の潜在的な欠点として言及されている(Byrd et al.) 従来の16S rRNA遺伝子アンプリコンシークエンシングがどの程度正確に生きた皮膚マイクロバイオーム組成を捉えるかを評価するために、我々はPMAに続いて16S rRNA遺伝子アンプリコンシークエンシング(PMA-seqと呼ぶアプローチ)を利用した(Nocker et al.) PMA処理(PMA-seq)とPMA処理なし(従来のシーケンス)のマッチしたサンプルのペアをシーケンスすることで、従来のシーケンス法で得られたマイクロバイオーム組成が、PMA-seqで得られた生存可能なマイクロバイオーム組成にどれだけ近いかを調べることができた(図3A)。これらの実験から、サンプリングした各皮膚部位において、従来のシーケンス法と比較して、PMA処理したサンプルはリッチネス(豊富度、Rは、同定可能な細菌分類群の数を示す指標)および多様性(多様性、Hは、シャノン多様性指数で測定)が低いことが確認された(図3BおよびC)。さらに、従来のシーケンス(Rtrad)ではリッチネスが大きかったサンプルは、PMA-seq(RPMAとHPMA)ではリッチネスとシャノン多様性の減少が比例して大きかった(図3BとC)。これらの結果は、従来のシーケンスでは皮膚部位によってリッチネス値の幅が広い(1~30種の分類群)ように見えるが、実際には異なる身体部位の皮膚マイクロバイオーム全体のリッチネスは比較的類似しており、低い(1~10種の分類群)ことを示唆している。したがって、従来のシーケンシングでは皮膚マイクロバイオームには幅広い多様性があるように見えるが、PMA-seqでは、これは一般に任意の皮膚部位における過大評価であることが示された。実際、我々の結果は、ほとんどの部位において、生存可能な皮膚マイクロバイオームは比較的少数の分類群に支配される傾向があることを示唆している。コンタミネーションコントロールを含む5人の健康なボランティアからの襟足サンプルの独立した解析では、コンタミネーション除去の有無にかかわらず、従来のシーケンスでは多様性と豊富性の両方が過剰に表現されるという同じ傾向が明らかになった(図6-図3)。
図3と1つの補足
アセットをダウンロードする オープンアセット
PMA-seqと比較した従来のシーケンスの相対存在量とリッチネスおよび多様性の変化。
(A)ファミリーレベルで配列決定された全細菌分類群の相対存在量。一対のバーは、従来のシーケンス(左)とPMA-seq(右)のデータを表す。サンプルは...続きを見る
図3-図1
アセットをダウンロード アセットを開く
同定された分類群の全リストと対応する色。
(A)図3に示した同定された細菌群の全リスト。
ほとんどの細菌群が従来のシーケンス法で過剰発現している
豊富性と多様性の指標は、皮膚マイクロバイオームの構成が従来のシーケンスとPMA-seqの間で異なる身体部位でどのように変化するかに関する重要な情報を提供する。さらに、特定の細菌分類群の相対的存在量の変化を理解したいと考えた。分類群レベルのPMA依存性変化を定量化するために、各細菌分類群についてPMAインデックス(IPMA)を開発した: IPMA = APMA(A|PMA)+(A|trad)����+����� ここで、APMAはPMA-seqによる相対存在量、Atradは従来のシーケンスによる相対存在量である。PMA指数が低い(<0.5)場合は、当該分類群がトラディショナルシーケンシングで過剰発現していることを示し、PMA指数が高い(>0.5)場合は、当該分類群がトラディショナルシーケンシングで過小発現していることを示す(図4A)。これらの値は、他の配列に対する濃縮度を表し、生存率スコアとは異なる測定値であることに留意されたい。PMAインデックスを計算すると、ほとんどの分類群が0に近いPMAインデックス値を示したことから、任意の身体部位におけるほとんどの細菌分類群の存在量は、従来のシーケンスでは過大評価されていることが明らかになった(図4A)。16S rRNAコピー数はこれらの値に影響を与える可能性があるが、最も豊富な種の16Sコピー数はすべてほぼ2倍の範囲内であったため、この影響は観察された傾向と比較すると軽微である(Stoddard et al.)
図4と1つの補足
アセットをダウンロード オープンアセット
プロピジウムモノアジド(PMA)インデックスと、従来とPMA-seq間の相対存在量の変化。
(A)少なくとも4サンプルに存在した各細菌分類群のPMAインデックスを、図3に示した同じサンプル部位のサンプル間の平均としてここに示す。色はPMAインデックスを示す。
図4-図1
アセットをダウンロード アセットを開く
リゾスタフィンとブドウ球菌特異的PCRプライマーを用いた皮膚3部位の生菌スコア。
(A)最も豊富な細菌DNAを有する3つの皮膚部位を示す。各サンプルの半分をDNA分離前にリソスタフィンで処理し、バイアビリティスコアがどのように変化するかを評価した。... もっと見る
最も豊富な4つの細菌ファミリー(Propionibacteriaceae、Staphylococcaceae、Corynebacteriaceae、Micrococcaceae)は、全シーケンシングリードの93%(PMA-seqリードの96%、従来のシーケンシングリードの91%)を占め、ファミリーレベルの興味深いPMAインデックスパターンを示した。プロピオニバクテリウム科には、皮膚マイクロバイオームの主要な構成要素であるC. acnesが含まれており、従来のシーケンスでは、部位のタイプにかかわらず、皮膚マイクロバイオームの50%以上を構成することが示されている(Byrd et al.) PMA-seqにより、従来のシーケンシングは皮脂部位におけるプロピオニバクテリウム科の存在量を正確に表している(PMAインデックスが0.5に近いことで実証)が、湿潤部位および乾燥部位ではプロピオニバクテリウム科を過剰に表していることが明らかになった(PMAインデックスが0.2~0.3)。さらに、プロピオニバクテリウム科細菌は皮脂分泌部位を支配していたが(ほとんどの皮脂分泌サンプルで全生存細菌の75%以上を占めた)、湿潤部位や乾燥部位は支配していなかった(これらのサンプルのいずれにおいても生菌数は全生存細菌の50%を超えなかった)(図4AおよびB)。プロピオニバクテリウム科と同様に、ブドウ球菌科の細菌はすべての体部位に広く分布しているようであった(図4AおよびB、図4-図1)。
コリネバクテリウム科の細菌も皮膚マイクロバイオームの主成分と考えられているが、我々の結果は、従来のシークエンシングでは、鼻腔を除くすべての皮膚部位でコリネバクテリウム科の存在量が過大評価されていたことを示している。例えば、従来のシークエンシングでは、膝窩にコリネバクテリウム属細菌が多く存在することが同定されたが、PMA-seqでは、これらのリードは大部分が不育症由来であることが示された(図4B)。以前の研究で、コリネバクテリアは鼻腔分離株から容易に培養できることが証明されており、この分類群の生存可能なメンバーが鼻腔に豊富に存在するが、他のほとんどの皮膚部位には存在しないというPMA-seqの結果を裏付けている(Liu et al.)
興味深いことに、Micrococcaceaeは毛幹を除くすべての部位で、従来のシークエンシングで過剰発現していた。毛幹では、MicrococcaceaeはPMA-seqで豊富に検出されたが、従来のシーケンスではほとんど検出されなかった。図4Bに示すように、PMA-seqで検出された生存可能なMicrococcaceaeの相対的割合の増加は、生存可能なプロピオニバクテリウム科細菌の減少に対応しており、Micrococcaceaeは、生存不能なプロピオニバクテリウム科細菌のDNAが多いため、従来のシーケンスでは検出されなかった可能性が示唆される。これらの結果は、ほとんどの皮膚部位は比較的少数の細菌科によってコロニー形成され、異なる細菌科は異なる皮膚部位に明瞭にコロニー形成され、皮膚表面の追加的な細菌DNAの大部分は不育症菌由来であることを示唆している。
マウスの皮膚マイクロバイオームの空間的構成と生存率はヒトのそれと類似している。
ヒトの皮膚マイクロバイオームのFISHとPMAの研究は強く一致しているが、同じサンプルで2つのアプローチを直接比較することはできない。同じサンプルでFISHとPMA-ddPCRの両方を実施できるシステムとして、我々はマウスの皮膚に注目した。K14-H2B-GFPマウスの組織を用いて、汎用細菌EUB338 FISHプローブを用いて、マウス皮膚組織における細菌細胞の空間分布を評価した。その結果、ヒト組織で見られたのと同じ細菌分布が観察された:毛包に細菌が多く(濃縮スコア15.26)、皮膚表面には細菌が比較的少ない(濃縮スコア0.21)(図5AおよびD)。毛皮が皮膚表面の細菌の存在にどのような影響を与えるかを調べるため、ヌードマウス(SKH1-Hrhr Elite)の皮膚でFISH染色を行ったところ、同様の細菌分布が認められた(毛包関連の濃縮スコアは10.79であったのに対し、皮膚表面の濃縮スコアは1.13であった)(図5BおよびD)。毛包に相当数の細菌が観察されたことから、表面細菌が存在しないのは、単に実験用マウスの飼育環境が比較的清潔であるためだけではないことが示唆された。マウスの組織は解剖前に滅菌剤で処理されていないため、これらの結果は、ヒト組織サンプルで認められた皮膚表面関連細菌の数が少ないことが、単に生検前の滅菌の結果ではないことも示唆している。
図5
ダウンロード資産 オープン資産
マウス組織の細菌蛍光in situハイブリダイゼーション(FISH)染色(A-D)およびマウスの生存率スコアとヒトの生存率スコアの比較。
(A). EUB338で染色したK14-H2B-GFPマウスの組織は、毛包に豊富な細菌シグナルを示すが、皮膚表面にはない。(B)SKH1-Hrhrエリートヌードマウスからの組織も細菌シグナルを示した。
試験した皮膚サンプルの表面には多くの細菌が検出されなかったので、FISH染色で皮膚表面の細菌が存在する場合、それを可視化できることを確認するための陽性対照を探した。例えば、皮膚の乾燥した酸性の性質がFISHの効果に影響し、皮膚表面での染色がほとんどできない可能性がある。この目的のために、動物から取り出したマウスの背側皮膚組織に大腸菌細胞を塗布した。この組織をヒトや他のマウス組織と同様に処理した。FISH染色により、これらのサンプルの表面に多くの細菌が確認され、この技術を用いて皮膚表面の細菌を確実に可視化できることが確認された(図5C)。陰性対照として、EUB338 FISHプローブの逆相補体をコードするプローブ(NONEUB338)が皮膚表面や毛包に有意にハイブリダイズしないことも確認した(図5C)。これらの結果は、EUB338染色が皮膚表面という生物学的背景でうまく機能することを実証し、ヒト組織でのFISH実験の結論を支持するものである。
次に、PMA-ddPCRを用いてマウスの皮膚マイクロバイオームにおける細菌の生存率を評価した。我々のこれまでの知見がヒト特異的なものではないことを示すさらなる証拠として、マウス皮膚マイクロバイオーム部位のPMA-ddPCRベースの生存率スコアは、ヒト皮膚部位の平均生存率スコア(それぞれ0.066と0.045)と同程度であり、マウスまたはヒト糞便マイクロバイオームの生存率スコア(それぞれ0.98と0.66)よりもはるかに低かった(図5E)。これらの結果は、皮膚生物学が異なるにもかかわらず、ヒトもマウスも、生存細胞とは関連しない細菌DNAが皮膚表面に豊富に存在することを示している。
皮膚表面の再増殖は、皮膚表面下に存在する安定した細菌群集によって促進される
最後に、皮膚マイクロバイオーム表面の摂動と再増殖という観点から、今回の知見の機能的意義を明らかにしようとした。具体的には、健康なボランティアの額の皮膚マイクロバイオームを時間T=0でサンプリングしてベースラインの微生物群集を確立し、塩化ベンザルコニウムで滅菌した後、3時間後、24時間後、48時間後にサンプリングして回復をモニターするという摂動-回復実験を行った(図6A)。各サンプリング時点において、DNA単離とその後の16S rRNA遺伝子配列決定に先立ち、サンプルの半分をPMAで処理した。PMA未処理のサンプルに注目すると、5人のボランティアのうち4人は、滅菌によってDNAの絶対量が有意に減少した(図6B)が、皮膚マイクロバイオームの全体的な細菌組成は比較的安定していることがわかった(図6A、図6-図2.) ボランティア4で発見された皮膚マイクロバイオーム組成は、他のボランティアで同定された群集とは著しく異なるが、同定された種は依然として健康な皮膚マイクロバイオームの一般的な構成要素であることに留意すべきである。さらに、ボランティア4は全体的に細菌DNAの存在量が少なく、これがプロピオニバクテリウム科の相対的存在量の低さにつながっている可能性がある。
図6 3つの補足
アセットをダウンロード アセットを開く
皮膚マイクロバイオームの摂動と回復。
(A)摂動後48時間における各個体の細菌相対量。0時間はベースライン、摂動前の群集を表す。サンプルがプロピジウムモノアジドで処理されたかどうか...続きを見る
図6-図3
アセットをダウンロード アセットを開く
600ntシーケンスデータで行ったコンタミネーション除去。
従来のシーケンス(Htrad)とPMA-seq(HPMA)の間のシャノン多様性の変化を、Htradに対する多様性の変化(∆H)をプロットすることで示した(A-C)。従来の配列決定(Htrad)とPMA-seq(HPMA)の間のリッチ... もっと見る
図6-図2
資産をダウンロードする 資産を開く
皮膚マイクロバイオームの摂動と回復。
(A)摂動後48時間における各個体のBray-Curtis非類似度。赤いデータポイントは、プロピジウムモノアジド(PMA)処理サンプルとPMA未処理のベースラインサンプルを比較したものである。... もっと見る
図6-図1
アセットをダウンロード アセットを開く
摂動回復で同定された細菌のリスト。
ここに列挙した細菌群は、図6に示したシーケンスデータセット全体に対応する。細菌は相対的存在量の高い順に記載されている。
我々の発見は、皮膚マイクロバイオームが短いタイムスケールでは不安定であり、長いタイムスケールでは安定であるという、以前に報告されたパラドックスを再現している(Nielsen and Jiang, 2019; Oh et al.) 重要なことは、PMA処理したサンプルの解析により、回復ダイナミクスを理解することができたことである。具体的には、Bray-Curtis非類似度を用いて個体群回復の程度をモニターした(図6C)。その結果、PMAを使用したサンプルと使用しなかったサンプルをPMA処理したベースライン・コミュニティと比較すると、すべての個体で一貫したパターンになることがわかりました。実験の経過とともに、コミュニティはPMA処理したベースライン・サンプルに収束していったが、PMA未処理のベースライン・サンプルに対する類似性の一貫したパターンは示さなかった(図6-図2A)。この結果は、皮膚表面の細菌DNAは容易に除去され、皮膚表面下の保護されたリザーバー内の生存可能な細菌集団によって継続的に補充されるという仮説を支持するものである。
再増殖の動態をASVレベルでより詳細に調べると、表面滅菌時に消失したASVが時間の経過とともに再び出現することが多く、表面再増殖は表面下の集団によって駆動されるというモデルと一致することがわかった。具体的には、表面不妊手術後、皮膚は不妊手術前と同じASVによって再増殖されることがわかった(図6-図2B)。図6補遺2Bでは、あるASVが表面殺菌前(T0)に生細胞集団(+PMA)に出現し、殺菌後(T3)に生細胞集団から消失し、その後24時間後または48時間後に再び生細胞集団に出現する具体例を強調している。これらの結果から、細菌は皮膚表面下の保護された領域で増殖し、その死骸のDNAが皮膚表面に蓄積することが示唆される。このように、皮膚表面の細菌DNAは、その下の群集の指紋として機能する。
考察
ここで我々は、イメージングとPMAに基づく方法の両方を用いて、皮膚表面には細菌がまばらにコロニー形成していることを証明した。この中心的な知見は、ヒトの生検、健康な綿棒ボランティア、ヘアリーマウス、ヌードマウスの皮膚に共通して当てはまる。皮膚マイクロバイオームは、免疫系を教育し、病原体と闘い、創傷治癒を促進する手段として大きな注目を集めており、複数のグループが皮膚プロバイオティクスを追求している(Yu et al.) 今回の発見は、こうした皮膚マイクロバイオームの機能の根底にあるメカニズムや、皮膚マイクロバイオームの組成を操作する能力にとって重要な意味を持つ。例えば、皮膚マイクロバイオームによる免疫教育の重要な機能は毛包内で起こることを示唆する先行研究(Polak-Witka et al. 私たちの発見はまた、個々の毛穴がクローン性の細菌集団によってコロニー形成されていることを示す以前の研究結果(Conwillら、2022年)を裏付けている。一方、今回の結果は、外因性細菌を皮膚表面に安定的にコロニー形成させることが困難であり、しばしば擦過を必要とする理由を説明する一助となるかもしれない(Scharschmidt et al.、2015)。皮膚剥離の際に生じる角質層(皮膚表層)の破壊は、安定した細菌集団が存在する組織深層へのアクセスを可能にする可能性がある。したがって、毛包内の細菌を標的とすることは、皮膚マイクロバイオームを安定的に操作したり、免疫系を教育したりするための、より優れた戦略である可能性がある。細菌が毛包に生息することは以前から知られていたが(Polak-Witkaら、2020;Lousadaら、2021)、今回の発見はこの知識を拡張し、皮膚マイクロバイオームの生菌は主にこの部位に限定されることを示唆している。このことは、皮膚表面から細菌を容易に培養することができるが、皮膚上に存在する細菌のDNA量から示唆される量よりもはるかに少ない量であるという我々の発見によってさらに裏付けられる。
皮膚マイクロバイオームの組成は、長い時間スケールで安定している一方で、一過性の擾乱の影響を受けやすいことが判明している(Nielsen and Jiang, 2019; Oh et al.) 皮膚は主に毛包単位やその他の皮膚侵入部において生存細菌によってコロニー形成されており、皮膚表面の細菌DNAはほとんどが死滅した細菌のものである。われわれは、皮膚表面の細菌DNAは容易に洗い流される可能性がある一方で、皮膚表面の細菌DNAは洗浄に影響されることなく残存し、新しい細菌DNAの小さいながらも安定した供給源として存続し、時間の経過とともに皮膚表面に補充されることを示した。このモデルは、皮膚マイクロバイオームに関連する偏性嫌気性細菌種が逆に多いことも説明している。ヒトの皮膚細胞もまた、皮膚表面の下で生まれ、死ぬと同時に表面に押し出される。このように、皮膚マイクロバイオームのライフサイクルは、皮膚の上皮細胞のターンオーバー過程に似ているのかもしれない。
今回の発見はまた、皮膚表面のコロニー形成が不十分である理由に焦点を当てた今後の研究によって解決されるべき根本的な疑問を提起している。皮膚以外のマイクロバイオームにおけるアクセス可能なDNAは、一般的に生存可能な細菌細胞を代表している。これは、皮膚マイクロバイオームが0.02から0.12の生菌数であったのとは対照的である。多くの抗菌性化合物を含む唾液でさえ(van 't Hof et al., 2014)、生存性スコアは皮膚マイクロバイオームの最高生存性スコアの4倍近くあった。皮膚に関連する生存性スコアが特異的に低いのは、細菌細胞が死滅した後、細菌のDNAが長い間皮膚に付着しているような受動的メカニズムによって説明できるかもしれない。あるいは、上皮細胞によって産生される抗菌ペプチドのような要因による皮膚表面での細菌死滅、細菌種間の競争、飢餓、紫外線、乾燥のような過酷な環境要因への曝露のような能動的メカニズムも考えられる。もう一つの未解決の疑問は、真菌やウイルスのような皮膚マイクロバイオームの非細菌成分についても同様の傾向が当てはまるかどうかである。
材料と方法
ヒトおよびマウスの組織処理
詳細なプロトコールを請求する
ヒト組織は、ペンシルバニア大学のSkin Biology and Diseases Resource-based Center(SBDRC)内のSkin Translational Research Coreを通じて入手した。正常な成人ヒト皮膚は、皮膚癌手術の際に得られた。癌組織を切除した後、美容的な修復を確実にするために正常な(癌でない)組織も切除しなければならない。これらの組織部分は通常廃棄されるが、画像化のために採取された。手術前に外科用スクラブ液(アルコール/ヨードまたはクロルヘキシジン)を使用した。組織は切除後すぐに室温のOCTに包埋し、-80℃で保存した。組織は32歳女性の額と81歳男性の頬から採取した。組織はSBDRCのCore B- Skin Translational Research Coreを通じて、'Fresh normal and diseased human skin'サービスの一環として入手した。
すべてのマウスは公認動物施設で飼育・管理され、すべての実験はUSA Public Health Service Policy of Humane Care and Use of Laboratory Animalsに従って行われた。すべてのプロトコールは、Institutional Animal Care and Use Committeeのプロトコール#1867(Princeton University)により承認された。蛍光実験にはK14-H2B-GFP、C57BL/6J、SKH1-Eliteヌードマウスの背部皮膚を用いた。K14-H2B-GFPおよびC57BL/6Jマウスの毛皮は、解剖前にバリカンで剃毛した。背部組織を除去し、薄い(~5 mm)ストリップに切断し、室温OCTに包埋した。OCTブロックはドライアイスの上に置いて凍結させ、その後-80℃で保存した。雌雄ともに使用した。すべての動物は3~6週齢で、非殺菌の標準的なげっ歯類用飼料を与えた。施設スタッフによる定期的な健康診断で病原体は確認されなかった。マウスは1ケージあたり1~5匹で飼育され、寝具とエンリッチメントはベンチレーテッド・ソーレン・ラックを使用した。マウスの安楽死はCO2吸入法(AVMA Guidelines for the Euthanasia of Animals: 2020 Editionによる)で行い、その後頚椎脱臼を行った。解剖はエタノールで滅菌した器具を用いて行った。
蛍光in situハイブリダイゼーションおよびイメージング
詳細プロトコールを請求する
ヒトとマウスの組織は同じように処理した。30μmの組織切片を切り出し、Leica CM3050Sクライオスタットを用いてスライドにマウントした。スライド上の組織を1×PBS中4%ホルムアルデヒドで20分間固定した。固定後、組織を1×PBSで5分間洗浄し、10mg/mLのリゾチームと1μg/μLのFISHプローブを含むハイブリダイゼーションバッファー(0.9M NaCl、20mM Tris-HCl、10%ホルムアミド)中で47℃、暗所で2~3時間インキュベートした。FISHプローブの塩基配列はSupplementary file 1にある。ハイブリダイゼーション後、スライドを洗浄バッファー(0.9M NaCl、20mM Tris-HCl)中、暗所、47℃で1時間インキュベートした。その後、スライドを1×PBSで10分間洗浄した。組織核を可視化するために、組織を1μg/mLのDAPIで室温で10分間染色した。その後、スライドを1×PBSで10分間ずつ3回洗浄した。最後の洗浄ステップの後、組織をグリセロールベースのアンチフェード非硬化マウンティングメディアでマウントした。カバースリップは、ワセリン、ラノリン、パラフィンの1:1:1:1混合液で密封した。画像はNIS Elementsソフトウェアで制御されたNikon A1R-Si HD共焦点顕微鏡で取得した。画像処理にはImageJとMATLAB(The MathWorks, Natwick, MA)を用いた。蛍光濃縮スコアを計算するために、Zスタックの最大投影を使用した。幅Wの長方形の関心領域(ROI)を、DAPIチャンネルのみを用いて卵胞の周囲に描いた。卵胞開口部近傍の卵胞関連蛍光を捉えるために、卵胞開口部近傍に0.5W×2Wの第2のROIを描いた。直交処理ステップでは、FISH蛍光チャンネルの画像全体のピクセル強度を、非組織バックグラウンド、非プローブ自家蛍光、およびプローブベースのシグナルにほぼ対応する3成分ガウス混合モデル(GMM)を用いてフィッティングした。このGMMを各画像のバックグラウンドサブトラクション値として使用し、各ROIの平均強度を算出した。この閾値を用いて ROI 内と ROI 外の平均蛍光値を算出した。ROI内の平均蛍光値とROI外の平均蛍光値の比を、毛包および角層関連蛍光の定量に用いた(濃縮スコア)。有意性はMann-Whitney検定を用いて算出した。
定常期培養は以下の条件に従って増殖させた:
菌株増殖時間(時間)培地環境条件Cutibacterium acnes ATCC691972強化クロストリジウム培地(RCM)(Oxoid、CM0149)嫌気性、37°Corynebacterium striatum ATCC694024脳心筋注入(BHI)(BD、 237500)Aerobic, 30°CMicrococcus luteus ATCC469824Tryptic soy broth (TSB) (BD, 211825)Aerobic, 30°CStaphylococcus epidermidis EGM 2-0624Tryptic soy broth (TSB) (BD, 211825)Aerobic, 37°C
ヒト被験者のマイクロバイオームサンプル
詳細プロトコールを請求する
マイクロバイオームサンプルは、Princeton University IRB #13003に基づき 、Princeton University Department of Molecular Biologyで採取された。健康なボランティアは、案内チラシで募集した。ボランティアはサンプル採取前にインフォームド・コンセントを行った。参加者は健康なボランティアで、男女、白人、26~35歳、慢性皮膚疾患や自己免疫疾患の既往歴がなく、サンプル採取時およびサンプル採取1ヶ月前に抗生物質を使用していなかった。
健康なボランティアの皮膚マイクロバイオームは、滅菌1×DPBSであらかじめ湿らせた滅菌泡立て綿棒を用いて採取した。しばしば含まれるが、細菌の細胞膜に悪影響を及ぼし、生存率スコアが変化するのを避けるため、綿棒のバッファーには洗剤を使用しないことにした。図 2-図 1G は、0.1% Triton X-100 (Sigma)を使用したスワッビングと使用しな いスワッビングが、バイアビリティスコア、ddPCR 結果、CFU にどのような影響を与えるかを示している。関心領域は、滅菌 1×DPBS に再懸濁する前に 60 秒間サンプリングした。舌のマイクロバイオームサンプルは、滅菌泡立て綿棒を用いて採取した。毛幹サンプルは、毛を摘み取り、球根部分のみを用いて採取した。毛包内容物はBioré Deep Cleansing Pore Strips(Kao USA Inc, Cincinnati, OH)を用いて採取した。唾液は、健康なボランティアから滅菌した50mLコニカルに採取した。歯垢は、健康なボランティアの歯を滅菌つまようじで削り、滅菌1×DPBSに懸濁して採取した。C57BL/6マウスの糞便サンプルは解剖時に採取した。図2-図1 に示したヒト皮膚マイクロバイオームサンプルは、PMA添加前にCFU計算用にプレーティングした。サンプルは血液寒天培地(トリプシン大豆寒天培地中5%ヒツジ血、VWR International)にプレーティングし、好気的または嫌気的に24~48時間培養した。摂動回復実験では、実験期間中(48時間)およびベースラインサンプリングの24時間前から、ボランティアは顔にいかなる種類の製品も使用しないよう求められた。ボランティアの入浴/シャワーは許可されたが、洗顔は控えるよう求められた。皮膚表面の殺菌は、塩化ベンザルコニウム0.13%w/v(North by Honeywell、#150910)を含む消毒用ウェットティッシュでサンプル部位を60秒間こすることで行った。皮膚表面のサンプリング過程が細菌の再増殖動態に影響を与えないようにするため、各ボランティアの額を面積の等しい4つのセクションに分けた: T0(ベースライン、滅菌前)、T1(滅菌後3時間)、T2(滅菌後24時間)、T3(滅菌後48時間)。各セクションは、滅菌1×DPBSであらかじめ湿らせた滅菌泡立て綿棒を用いて60秒間サンプリングした。上記のように、各スワブを滅菌1×DPBSに懸濁した。
ヒト糞便サンプルはPrinceton University IRB #11606に基づきDonia labによって採取され、Gitai labに贈られた。このサンプルと採取方法はJavdanら、2020に記載されている。
加熱死滅大腸菌比
詳細なプロトコールを請求する
PMAの有効性を実証するために、大腸菌の生菌培養と加熱死菌培養を既知の比率で混合し、PMA処理に供した。まず、一晩培養した大腸菌NCM3722を新鮮なLB培地に1:1000の割合で逆希釈し、37℃で4時間培養してミッドログに到達させた。定常期の実験は一晩、18時間の培養で行った。培養液を3×洗浄し、滅菌PBSに懸濁した。培養液の半分を70℃で20分間インキュベートし、残りの半分を室温で静置した。加熱死滅させた大腸菌培養物を室温まで冷却した後、非加熱死滅の大腸菌培養物と合わせて50%(体積比)の加熱死滅混合物とした。0%加熱死菌では、加熱死菌は加えなかった。同様に、100%熱-キルドでは、熱-キルド菌のみを使用した。各条件をよく混合し、滅菌した1.5mL微量遠心チューブ2本に均等に分けた。
PMA処理とDNA単離
詳細プロトコールを請求する
採取後、サンプルを2本の滅菌済み1.5mL微量遠心チューブに均等に分注した。2本のチューブのうち1本にPMA(Biotium Inc)を最終濃度50μMになるように添加した。すべてのチューブを暗所、室温で10分間インキュベートした後、PMA-Lite LED Photolysis Device(Biotium Inc)を用いて光を照射し、PMA分子を架橋させた。その後、DNeasy PowerSoil Kit(QIAGEN)を用いるか、Meiselら、2016に概説されたプロトコールに従って、すべてのサンプルからDNAを単離した。Meiselらのプロトコールは、細胞溶解の目的でビーズ叩打の代わりに超音波処理(Branson Sonifier 250, VWR Scientific)を使用するように修正した。どちらのDNA単離法も同様に機能する(図2-図1H)。リゾスタフィン(Sigma-Aldrich)を使用する場合は、PMA活性化後、DNA単離前に最終濃度0.1 mg/mLになるように添加し、室温で30分間インキュベートした。
液滴デジタルPCR (ddPCR)
詳細プロトコルのリクエスト
Bio-Rad QX200 AutoDG Droplet Digital PCR Systemを使用して、マイクロバイオームサンプルおよび純粋な細菌培養から抽出したDNAを定量した。反応混合物には、2×QX200 ddPCR EvaGreen Supermixと10 nM濃度のuniversal 16S qPCRプライマーを総容量25 µLで含む。プライマーの配列はSupplementary file 1にある。反応混合物を滅菌ddPCR 96ウェルプレート(Bio-Rad #12001925 )に移し、QX200 Automated Droplet Generatorにセットした。液滴生成後、PX1 PCR Plate Sealer (Bio-Rad #1814000 )を用いてプレートをヒートシールし、C1000 Touch Thermal Cycler (Bio-Rad #1851197 )を用いて、95℃、5分間のプレステップ、60℃、1分間のエクステンション、12℃の最終ホールド温度で40ラウンドの増幅を行うPCRを行った。その後、サンプルをQX200 Droplet Readerにロードし、定量を行った。Quantasoftソフトウェアを用いて自動閾値処理を行い、その後Microsoft Excelにエクスポートして解析を行った。有意性はStudent's t-testを用いて計算した。PMA'と'+PMA'をマッチさせたサンプルの所定のペアに対する生存率スコアを計算するために、以下の計算を行った:
PMAを添加したコピースパー20μL PMAを添加しないコピースパー20μL
ddPCRおよびCFU標準曲線
詳細プロトコルを請求する
S. epidermidis EGM 2-06の培養液をトリプシン系大豆ブロス(TSB)で一晩培養し、翌朝TSBで1:1000に希釈し、最終ODが0.4になるまで4時間培養した。その後、S. epidermidis培養液の10倍希釈液を調製し、トリプティック大豆寒天培地(VWR International)中の5%ヒツジ血液上にCFUを測定するためにプレーティングし、2本の1.5mL微量遠心チューブに分注した。C.acnes、S.epidermidis、M.luteus、およびC.striatumの定常期培養を、上記の表に従って増殖させた。一方のチューブにPMAを最終濃度50μMとなるように添加し、もう一方のチューブは未処理のままとした。PMAの活性化とDNAの単離は、上記の方法に従って行った。
16S rRNA遺伝子アンプリコンシークエンシング
詳細なプロトコールを請求する
DNeasy PowerSoil Kit(QIAGEN)を用いて(図2-5)、あるいはMeiselら(図6)に概説されたプロトコールに従って、マイクロバイオームサンプル(PMAあり・なし)からDNAを単離した。16S遺伝子のV1-V3領域は、プライマー27F(5′- AGAGTTTGATCCTGGCTCAG)および534R(5′- ATTACCGCGGCTGCTGG)を用いて増幅した。イルミナシーケンスライブラリーは、以前に発表されたプライマーを用いて調製した(Caporaso et al.) ライブラリーを等モル比でプールし、ペアエンドリードとしてIllumina MiSeq Micro 500 nt(図3および4)またはMiSeq V3 600 nt(図6)でシーケンスした。リードは2×250 bpまたは2×300 bpで、平均深度は500 ntで~33,616リード、600 ntで245,488リードであった(補足ファイル2および3)。また、メーカーのプロトコールに従った8 bpインデックスリードも含まれる(Illumina、米国)。生シーケンスリードはIllumina HiSeq Control Softwareでフィルターし、さらなる解析のためにPass-Filterリードを生成した。インデックスリードはサンプルデマルチプレックスに使用した。次に、QIIME2バージョン2018.6内のDADA2プラグイン(Bolyenら、2018;Callahanら、2016)を使用して、アンプリコンシーケンスバリアント(ASV)を配列から推論した。リバースリードは、2×250 bpデータでは245 bpにトリミングされ、2×300 bpデータでは275 bpにトリミングされた。2×250 bpのデータでは、2種類のASV推論法を使用した。1つはフォワードリードのみを使用し(平均より長いStaphylococcusアンプリコンを捕捉するため)、もう1つはペアエンドリードの両方を使用した。2×300bpのデータについては、ペアエンドリードを両方用いてASV推論を行った。フォワードリードを用いたASV推論はDADA2のdenoise-single関数を用いて行い、両リードを用いたASV推論はdenoise-paired関数を用いて行った。分類学は、ASVによってスパンされた16S rRNA遺伝子の領域のみを使用してGreenGenesデータベースバージョンで訓練されたナイーブベイズ分類器を使用して、結果のASVに割り当てられた(Bokulichら、2018; McDonaldら、2012)。すべての下流解析は、ASVレベルであることが指定されていない限り、ファミリーレベルの分類学的割り当てを用いて実施した。500ntシーケンスデータにはライブラリー調製ステップでPBSコントロールサンプルを含め、600ntシーケンスデータには空気スワブDNA分離コントロールを含めた。コンタミネーション除去は、R用Decontamパッケージを使用して600 ntシーケンスデータで実施した(図6および図6-図2には、コンタミネーション除去なしのデータを示す;Davis et al.) 図6-図3は、従来のシーケンスとPMA-seqの間でシャノン多様性とリッチネスがどのように変化するかを示している(図3BとCに示したものと同様)。汚染物質の除去(0.1または0.2閾値のいずれかを使用)はデータにほとんど影響せず、空気スワブコントロールがないため500 ntシーケンスデータで汚染除去を実行できないが、これは図3および4に示す全体的な所見を変えないはずであることを示唆している。相対的存在量、豊かさ、シャノン多様性、Bray-Curtis非類似度、PMA-indexは、Vegan package for RまたはMicrosoft Excelを用いて評価し、R、Prism、MATLABを用いてプロットした(J. Oksanen, F. G. Blanchet, M. Friendly, R. Kindt, P. Legendre, D. Mcglinn, P. R. Minchin, R. B. O'Hara, G. L. Simpson, P. Solymos, M. H. H. Stevens, E. Szoecs, H, 2019)。PMA指数は相対存在量を用いて計算され、4サンプル未満に存在する細菌分類群については計算されなかった。
ADD A COMMENT +アノテーションを開く。このページの現在のアノテーション数は0です。
データの利用可能性
本研究で作成および解析したシーケンスデータセットは、NCBI Sequence Read Archive (SRA) (BioProject number PRINA918671 and PRINA918959)で入手可能。使用したオリゴヌクレオチド配列は表S1に含まれる。
以下のデータセットが作成された
Acosta E
(2023) NCBI BioProject
ID PRJNA918671。ヒト皮膚マイクロバイオーム摂動回復。
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA918671
アコスタE
(2023) NCBIバイオプロジェクト
id prjna918959. ヒト皮膚マイクロバイオーム。
https://www.ncbi.nlm.nih.gov/bioproject/PRJNA918959
参考文献
Amann RI
ビンダーBJ
オルソンRJ
チショルムSW
デヴリューR
Stahl DA
(1990) 16S rRNA標的オリゴヌクレオチドプローブとフローサイトメトリーとの組み合わせによる混合微生物集団の解析
応用・環境微生物学 56:1919-1925.
https://doi.org/10.1128/aem.56.6.1919-1925.1990
Google Scholar
ベルカイド Y
タムトゥヌール S
(2016) 皮膚免疫における皮膚微生物の影響
Nature Reviews. Immunology 16:353-366.
https://doi.org/10.1038/nri.2016.48
PubMed
グーグル奨学生
ビエールRD
ヒューガースLW
ブールンドF
セイファートM
ヨハンセンJD
エングストランドL
(2019) ヒト皮膚マイクロバイオーム調査におけるサンプリング戦略とDNA抽出の効果
Scientific Reports 9:17287.
https://doi.org/10.1038/s41598-019-53599-z
PubMed
グーグル奨学生
ボクリッチ NA
ケーラーBD
ライドアウトJR
ディロンM
ボリエンE
ナイト R
ハットリーGA
グレゴリー・カポラソJ
(2018) QIIME 2のQ2-feature-classifierプラグインによるマーカー遺伝子アンプリコン配列の分類学的分類の最適化
Microbiome 6:90.
https://doi.org/10.1186/s40168-018-0470-z
PubMed
Google Scholar
プレプリント
ボリエン E
ライドアウトJR
ディロンMR
ボクリッチNA
アブネットC
アル・ガリスGA
アレクサンダー H
アルムEJ
アルムガム M
アスニカー F
バイ Y
ビザンツJE
ビッティンガー K
ブレインロッド A
ブリスローンCJ
ブラウンCT
カラハンBJ
カラバロ・ロドリゲス AM
チェイス J
コープ E
ダ・シルバ R
ドーレスタインPC
ダグラス GM
デュラルDM
デュヴァレC
エドワードソンCF
エルンスト M
エスタキ M
フーキエJ
ゴーグリッツJM
ギブソンDL
ゴンザレス A
ゴーリック・K
グオ J
ヒルマンB
ホームズ S
ホルステ・H
ハッテンハワー C
ハットリー・G
ヤンセン S
ヤルムッシュAK
ジアン L
ケーラーB
カン KB
キーフCR
ケイム P
ケリーST
ナイツ D
コスター I
コジオレック T
クレップスJ
ランギールMG
リーJ
レイ・R
リュー Y-X
ロフトフィールド E
ロズポーン C
マハー M
マロッツ C
マーティンBD
マクドナルド D
マカイバーLJ
メルニクAV
メトカーフJL
モーガンSC
モートンJ
ナイミーAT
ナバス-モリーナJA
ノチアスLF
オルチャニアンSB
ピアソンT
ピープルズSL
ペトラスD
プレウスML
プルエッセ E
ラスムッセン LB
リバーズ A
ロベソンII MS
ローゼンタール P
セガタ・N
シェイファーM
シファー A
シンハ R
ソングSJ
スピアJR
スワフォードAD
トンプソンLR
トーレス PJ
トリンP
トリパティ A
ターンボーPJ
ウル・ハサン S
ファン・デル・フフトJJ
バルガス F
バスケス=バエサ Y
フォクトマンE
フォン・ヒッペル M
ウォルターズ W
ワン Y
ワン M
ウォーレン J
ウェーバー KC
ウィリアムソンCH
ウィリスAD
シュー ZZ
ザネベルトJR
チャン Y
朱Q
ナイト R
カポラソJG
(2018) qiime 2: 再現可能、インタラクティブ、スケーラブル、拡張可能なマイクロバイオームデータサイエンス
PeerJ Preprints。
https://doi.org/10.7287/peerj.preprints.27295
グーグル・スカラー
バード AL
ベルカイドY
セグレJA
(2018) ヒト皮膚マイクロバイオーム
Nature Reviews. Microbiology 16:143-155.
https://doi.org/10.1038/nrmicro.2017.157
PubMed
Google Scholar
キャラハンBJ
マクマーディPJ
ローゼンMJ
ハンAW
ジョンソンAJA
ホームズSP
(2016) Dada2:イルミナアンプリコンデータからの高分解能サンプル推論
Nature Methods 13:581-583.
https://doi.org/10.1038/nmeth.3869
PubMed
Google Scholar
カポラソ JG
ラウバーCL
ウォルターズWA
バーグ-ライオンズD
ハントレーJ
フィーラーN
オーエンスSM
ベトリーJ
フレイザー L
バウアー M
ゴームリー N
ギルバート JA
スミス G
Knight R
(2012) イルミナHiseqおよびMiseqプラットフォームにおける超高スループット微生物群集解析
ISME Journal 6:1621-1624.
https://doi.org/10.1038/ismej.2012.8
Google Scholar
コンウィル A
Kuan AC
Damerla R
ポレットAJ
ベイカーJS
トリップAD
アルムEJ
リーバーマンTD
(2022) 解剖学はヒト皮膚マイクロバイオームにおける菌株の中立的共存を促進する
Cell Host & Microbe 30:171-182.
https://doi.org/10.1016/j.chom.2021.12.007
PubMed
Google Scholar
デイビス NM
プロクターDiM
ホームズSP
レルマンDA
カラハン BJ
(2018) マーカー遺伝子とメタゲノミクスデータにおける汚染配列の簡単な統計的同定と除去
Microbiome 6:226.
https://doi.org/10.1186/s40168-018-0605-2
PubMed
Google Scholar
ガロ RL
(2017) ヒト皮膚は微生物と相互作用する最大の上皮表面である
The Journal of Investigative Dermatology 137:1213-1214.
https://doi.org/10.1016/j.jid.2016.11.045
PubMed
Google Scholar
グライスEA
セグレJA
(2011) 皮膚マイクロバイオーム
Nature Reviews. 微生物学 9:244-253.
https://doi.org/10.1038/nrmicro2537
PubMed
グーグル奨学生
ハッテンハワーC
ゲバースD
ナイトR
アブバッカー S
バッジャーJH
チンワラAT
クリーシーHH
アール AM
フィッツジェラルドMG
フルトンRS
ギリオMG
ホールズワース・ペピンK
ロボスEA
マドゥプ R
マグリーニ V
マーティンJC
ミトレバ M
ムズニーDM
ソダーグレンEJ
ヴェルサロヴィッチJ
ウォラム AM
ウォーリーKC
ワートマンJR
ヤングSK
ゼンQ
アーガードKM
アボルードOO
アレン・バーコー E
アルムEJ
アルバラド L
アンデルセンGL
アンダーソン S
アッペルバウム E
アラチHM
アーミテージ G
アーゼCA
アイバズT
ベーカーCC
ベッグL
ベラチューT
ボナギリ V
ビハン M
ブレイザーMJ
ブルーム T
ボナッツィ V
ブルックスJP
バック GA
ブヘイCJ
ブザムDA
キャンベルJL
キャノンSR
カンタレルBL
チェーンPSG
チェン IMA
チェン・L
チバ S
チュー K
チウラDM
クレメンテJC
クリフトンSW
コンラン S
クラブツリーJ
カッティングMA
ダビドビクスNJ
デイビスCC
デサンティスTZ
ディール C
デレハンティKD
デューハーストFE
デイチ E
ディン・Y
ドーリングDJ
デュガンSP
ダンWM
ダーキンAS
エドガーRC
アーリックRL
ファーマーCN
ファレルRM
ファウストK
フェルドガーデンM
フェリックスVM
フィッシャー S
フォドーAA
フォーニーLJ
フォスターL
フランチェスコVD
フリードマン J
フリードリッヒDC
フロニックCC
フルトンLL
ガオ・H
ガルシア・N
ジャンヌコスG
ギブリン C
ジョバンニ MY
ゴールドバーグJM
ゴル J
ゴンザレス A
グリッグス A
グジャ S
ハーケSK
ハース BJ
ハミルトン HA
ハリスEL
ヘップバーンTA
ヘルターB
ホフマンDE
ホルダー ME
ハワース C
ホアン KH
ヒューズSM
イザード J
ヤンソン JK
ジアン H
ジョーダン C
ジョシ V
カタンシックJA
カイテル WA
ケリーST
ケルズC
キングNB
ナイツD
コングHH
コレンO
コレンS
コタ KC
コバーCL
キルピデスNC
ローザ PSL
リーSL
レモン KP
レノンN
ルイスCM
ルイスL
レイRE
リー・K
リオリオK
リウ・B
リウ・Y
ローCC
ロズポーンCA
ランズフォードRD
マデン T
マフルカーAA
マンノンPJ
マーディスER
マーコウィッツVM
マブロマティスK
マコーリソンJM
マクドナルドD
マキュウエンJ
マクガイアAL
マクインズP
メータ T
ミヒンドゥクラスリヤ KA
ミラーJR
ミンクスPJ
ニューシャムI
ヌスバウムC
オグラウリン M
オービスJ
(2012) 健康なヒトのマイクロバイオームの構造、機能、多様性
Nature 486:207-214.
https://doi.org/10.1038/nature11234
Google Scholar
ジャヴダン B
ロペスJG
チャンカムジョンP
リー YCJ
ハル R
ウー Q
ワン X
チャタジー S
ドニアMS
(2020) ヒト腸内細菌叢による薬物代謝の個別化マッピング
Cell 181:1661-1679.
https://doi.org/10.1016/j.cell.2020.05.001
パブコメ
Google Scholar
ソフトウェア
J. オクサネン、F. G. ブランシェ、M. フレンドリー、R. キント、P. レジェンドル、D. マクグリン、P. R. ミンチン、R. B. オハラ、G. L. シンプソン、P. ソリモス、M. H. H. スティーブンス、E. スゾエックス、H.
(2019)
Wagner, Vegan: Community Ecology package, version Rパッケージバージョン2.4-2
Community Ecol. パッケージ
カラン LR
Meisel JS
Loesche MA
ホルウィンスキー J
ソアイタ I
チェン X
ウベロイ A
ガードナー SE
グライスEA
(2019) 糖尿病創傷のマイクロバイオームにおける株レベルおよび種レベルの変異は、臨床転帰および治療効果と関連する
Cell Host & Microbe 25:641-655.
https://doi.org/10.1016/j.chom.2019.03.006
PubMed
グーグル奨学生
キム JH
ヤン B
テデスコ A
リービッヒEGD
ルーガーPM
シュー・K
ボーンマン J
マーティンス・グリーン M
(2019) 糖尿病マウスにおける慢性創傷の開始と発症には、高レベルの酸化ストレスと皮膚マイクロバイオームが重要である。
Scientific Reports 9:1-16.
https://doi.org/10.1038/s41598-019-55644-3
Google Scholar
リウQ
リウQ
Meng H
Lv H
Liu Y
劉 J
Wang H
He L
秦J
Wang Y
ダイ・イー
オットー M
Li M
(2020)表皮ブドウ球菌は抗菌ペプチド産生を刺激することで鼻腔マイクロバイオームの健全な成熟に寄与する
Cell Host & Microbe 27:68-78.
https://doi.org/10.1016/j.chom.2019.11.003
PubMed
Google Scholar
ローシェ M
ガードナーSE
カランL
ホルウィンスキー J
鄭Q
ホドキンソンBP
ティルスリーAS
フランシスカスCL
ヒリスSL
メータS
マーゴリスDJ
グライス EA
(2017) 慢性創傷微生物叢の時間的安定性は治癒不良と関連する
The Journal of Investigative Dermatology 137:237-244.
https://doi.org/10.1016/j.jid.2016.08.009
PubMed
Google Scholar
ルサダ MB
ラクニットT
エデルカンプJ
ルイエT
アジュディック D
内田裕也
ディ・ナルド A
ボッシュTCG
パウスR
(2021) ヒト毛包マイクロバイオームの探索
The british journal of dermatology 184:802-815.
https://doi.org/10.1111/bjd.19461
PubMed
グーグル奨学生
前田紘一
(2017) ヒトにおける表皮ターンオーバーの新しい測定法
化粧品 4:47.
https://doi.org/10.3390/cosmetics4040047
Google Scholar
マクドナルド D
プライス MN
グッドリッチJ
ナウロッキEP
デサンティスTZ
プロブスト A
アンデルセンGL
ナイト R
ヒューゲンホルツ P
(2012) 細菌と古細菌の生態学的・進化学的解析のためのランクを明示した改良型Greengenes分類法
The ISME Journal 6:610-618.
https://doi.org/10.1038/ismej.2011.139
Google Scholar
マイゼル JS
ハニガンGD
ティルスリーAS
サンミゲルAJ
ホドキンソンBP
鄭Q
グライス EA
(2016) 皮膚マイクロバイオーム調査は実験デザインに強く影響される
The Journal of Investigative Dermatology 136:947-956.
https://doi.org/10.1016/j.jid.2016.01.016
PubMed
Google Scholar
マイゼルJS
ナスコDJ
ブルーバッハB
セペダ・エスピノサV
チョピクJ
コラーダ・ブラボH
フェダルコ M
グリエJ
ジャヴカー K
オルソンND
シャー N
アラードSM
バジネットAL
バーグマンNH
ブラウン A
カポラソJG
コンラン S
ディルッジェーロJ
フォリーSP
ハサンNA
クラリJ
ルエティPM
ミルトンDK
オンドフBD
プレハイム S
ラトナヤケ S
ロジャース SM
ロソヴィッツMJ
サコウスキー EG
シュリーブスNO
ソマーDD
ターナスKL
ウリツキG
チャン SX
ポップM
トレアンゲンTJ
(2018a) バイオインフォマティクスのバイオディフェンスおよび病原体検出への応用における現在の進展と将来の機会:冬の中部大西洋マイクロバイオームミーティングアップ(2018年1月10日、メリーランド州カレッジパーク)からの報告
Microbiome 6:197.
https://doi.org/10.1186/s40168-018-0582-5
PubMed
Google Scholar
マイゼルJS
スフィロエラG
バートウ-マッケニーC
ギンブレットC
ブガエフJ
ホルウィンスキー J
キム・B
ブレストフJR
ティルスリーAS
鄭Q
ホドキンソンBP
アーティスD
グライス EA
(2018b) 通常性微生物叢は皮膚における遺伝子発現を調節する
Microbiome 6:20.
https://doi.org/10.1186/s40168-018-0404-9
PubMed
Google Scholar
ニールセン MC
江 SC
(2019) 海洋水曝露後のヒト皮膚マイクロバイオームの変化
Marine Pollution Bulletin 145:595-603.
https://doi.org/10.1016/j.marpolbul.2019.06.047
PubMed
グーグル奨学生
ノッカー A
張 CY
キャンパー AK
(2006) 死細胞からのDNA選択的除去による生菌と死菌の鑑別におけるプロピジウムモノアジドとエチジウムモノアジドの比較
Journal of Microbiological Methods 67:310-320.
https://doi.org/10.1016/j.mimet.2006.04.015
PubMed
グーグル奨学生
ノッカー A
リヒターハイトマンT
モンティンR
シュレンF
Kort R
(2010) 454パイロシーケンスによる環境水サンプルの細菌群集における生細胞と死細胞の識別
International Microbiology 13:59-65.
https://doi.org/10.2436/20.1501.01.111
PubMed
Google Scholar
Oh J
バードAL
パク M
Kong HH
セグレJA
NISC比較シーケンスプログラム
(2016) ヒト皮膚マイクロバイオームの時間的安定性
Cell 165:854-866.
https://doi.org/10.1016/j.cell.2016.04.008
PubMed
Google Scholar
ポラック・ウィツカ K
ルドニツカL
ブルーム・ペイタヴィU
フォクト A
(2020) 頭皮毛包の生物学と疾患におけるマイクロバイオームの役割
Experimental Dermatology 29:286-294.
https://doi.org/10.1111/exd.13935
PubMed
Google Scholar
シャーシュミットTC
バスケスKS
Truong HA
ギアティSV
パウリML
ノスバウム A
グラッツIK
オットー M
ムーンJJ
リーゼJ
アッバス AK
フィッシュバッハMA
ローゼンブラムMD
(2015) 新生児皮膚への制御性T細胞の波は、通性微生物に対する寛容を媒介する
Immunity 43:1011-1021.
https://doi.org/10.1016/j.immuni.2015.10.016
PubMed
Google Scholar
ストッダード SF
スミスBJ
ハインR
ローラーBRK
シュミット TM
(2015) Schmidt, rrnDB: 細菌と古細菌のrRNA遺伝子量を解釈するツールの改良と今後の開発のための新たな基盤
Nucleic Acids Research 43:D593-D598.
https://doi.org/10.1093/nar/gku1201
Google Scholar
ファン・ト・ホフW
ヴィールマンECI
ニーウ・アメロンゲンAV
リッテンバーグAJM
(2014) 唾液中の抗菌防御システム
Monographs in Oral Science 24:40-51.
https://doi.org/10.1159/000358783
PubMed
グーグル奨学生
ヤン・ヤン
チェン D
Li Y
ユー L
ジン KYH
チェン JP
ラインハルト M
(2017) 生物学的に処理された廃水中の細菌群集シフトに対するモノクロラミンと過酸化水素の影響
Chemosphere 189:399-406.
https://doi.org/10.1016/j.chemosphere.2017.09.087
Google Scholar
Yu Y
ダナウェイ S
チャンパー J
キム J
アリカン A
(2020年)我々のマイクロバイオームを変える:皮膚科学におけるプロバイオティクス
英国皮膚科学会誌 182:39-46.
https://doi.org/10.1111/bjd.18088
PubMed
Google Scholar
査読
査読者1(公開査読):
本論文の著者は、現在のDNAベースの微生物ゲノム解析手法には固有の制約があることを示す強力な証拠を提示した。これらのアプローチでは、塩基配列が決定されたDNAの出所を検出することができず、塩基配列が決定されたDNAが生きた細菌や生存していない細菌から得られたものであることを証明することができない。さらに科学者たちは、生きたバクテリアはほとんどの場合、皮膚表面ではなく毛包内に留まっていることを人やマウスで証明した。全体として、この研究は非常に質の高いものであり、特定のテーマにとどまらない広い意味を持っている。
長所:
この研究はよくデザインされており、実験方法もよく説明されている。
結果は明確に示され、統計解析によって裏付けられている。
この研究で得られた知見は斬新であり、皮膚マイクロバイオームと皮膚の生物学を理解する上で重要な意味を持つ。
弱点:
RNAベースのNGSは、このDNAベースのマイクロバイオーム研究の結果を並行して研究することが可能である。バルクRNA-Seqは、各生存菌から数千の転写産物をシーケンスし、細菌ゲノムおよびトランスクリプトームリファレンスと照合することができる。RNA-Seqは、皮膚常在細菌の多様性を示すのに最適な確認方法のひとつである。
https://doi.org/10.7554/eLife.87192.2.sa1
査読者2(公開査読):
Acostaらの研究は、皮膚中の生菌と死菌のDNAを同定するためのシンプルで簡単な方法-PMA標識とFISHによる検証-を提示しており、非常に興味深い。この研究は、今後の皮膚マイクロバイオーム研究に有益な結論をいくつか提示している:
第一に、綿棒で採取した皮膚微生物サンプルの16s rRNA遺伝子配列決定には、多数の死菌のDNAが含まれる可能性があり、その結果、皮膚細菌が過剰に解析に反映されることになる。
第二に、検出された細菌DNAから予測されるよりも、皮膚表面には生きた細菌が少なく、皮膚細菌のほとんどは毛包に生息していることがわかった。この結論は、毛包上皮が湿潤で栄養豊富、低紫外線、免疫に恵まれた環境を作り出し、微生物の増殖、コロニー形成、発育を助長しているという皮膚の生理学的特性と一致する。
最後に著者らは、皮膚表面の細菌は毛包常在菌の増殖と補充に由来するものであり、これが皮膚マイクロバイオームの短期的不安定性と長期的安定性の一因である可能性を提唱している。
全体として、本研究は皮膚細菌の組成と分布に関する貴重な洞察を提供し、皮膚マイクロバイオーム研究において生きた細菌を同定するために適切な方法を用いることの重要性を強調している。
https://doi.org/10.7554/eLife.87192.2.sa2
論文および著者情報
著者詳細
エレン・M・アコスタ
プリンストン大学分子生物学科、プリンストン、米国
貢献
概念化, ソフトウェア, 形式分析, 調査, 視覚化, 方法論, 原稿執筆, 原稿執筆-校閲・編集
競合利益
このORCID iDは、この論文の著者を特定するものである。
キャサリン・A・リトル
プリンストン大学分子生物学科、プリンストン、米国
貢献
調査、方法論
競合利益
このORCID iDは、この論文の著者を特定するものです: "0000-0001-6993-5312
ベンジャミン・P・ブラットン
プリンストン大学分子生物学科、プリンストン、アメリカ合衆国
プリンストン大学ルイス・シグラー統合ゲノミクス研究所、プリンストン、米国
バンダービルト大学医療センター病理学・免疫学・微生物学教室(米国・ナッシュビル
バンダービルト感染・免疫・炎症研究所(米国・ナッシュビル
貢献
ソフトウェア, 形式的解析, 可視化, 執筆 - 査読と編集
競合利益
このORCID iDは、この論文の著者を特定するものです。
ハイメ・G・ロペス
ルイス・シグラー統合ゲノミクス研究所、プリンストン大学、プリンストン、米国
貢献
ソフトウェア, 形式分析, 執筆 - 査読と編集
競合利益
競合利益なし
毛 暁明
米国フィラデルフィア、ペンシルバニア大学皮膚科
貢献
調査
競合利益
競合利益なし
エイミー・S・ペイン
米国フィラデルフィア、ペンシルバニア大学皮膚科
貢献
リソース、方法論、執筆 - 査読と編集
競合利益
このORCID iDは、この論文の著者を特定するものである。
モハメド・ドニア
プリンストン大学分子生物学科、プリンストン、米国
貢献
概念化、方法論、執筆 - 査読と編集
競合利益
競合利益なし
ダネル・デヴェンポート
プリンストン大学分子生物学科、プリンストン、アメリカ合衆国
貢献
概念化, 資料, 方法論, 執筆 - 査読と編集
競合利益
Reviewing editor, eLife "This ORCID iD identifies the author of this article: "0000-0002-5464-259X
ゼマー・ギタイ
プリンストン大学分子生物学科, プリンストン, アメリカ合衆国
貢献
概念化、リソース、監督、資金獲得、方法論、執筆-原案、執筆-校閲・編集
コレスポンデンス
zgitai@princeton.edu
競合利益
このORCID iDは、この論文の著者を特定するものです:0000-0002-3280-6178」。
資金提供
米国国立アレルギー感染症研究所(DP1AI124669)
エレン・M・アコスタ
ベンジャミン・ブラットン
ゼマー・ギタイ
国立総合医療科学院 (GM007388)
エレン・M・アコスタ
アメリカ国立科学財団 (PHY-1734030)
ベンジャミン・ブラットン
全米トランスレーショナル・サイエンス推進センター(TL1TR003019)
エレン・M・アコスタ
ペンシルバニア大学皮膚生物学・疾患リソースベースセンター(P30-AR069589)
Aimee S Payne
資金提供者は、研究デザイン、データ収集、解釈、論文投稿の決定に関与していない。
謝辞
Gitai研究室のすべてのメンバーの洞察とコメントに感謝する。また、Gary Laevsky博士とNikon Center of ExcellenceであるPrinceton Molecular Biology Microscopy Coreの顕微鏡サポートに感謝する。Wei Wang博士とプリンストン大学Lewis Sigler Institute for Integrative GenomicsのGenomics Core Facilityには16S rRNA遺伝子アンプリコンシーケンスのサポートを、Matthew Cahnには16S rRNA遺伝子アンプリコンシーケンスデータの処理を、Bahar Javdanにはヒト糞便サンプルの処理を、Elizabeth Griceにはサポートとフィードバックを、Laurice FlowersとElizabeth Griceには使用した表皮ブドウ球菌株を提供していただいた。資金の一部はNIHから提供された(ZG、EMA、BPBにはDP1AI124669、EMAにはT32 GM007388)。また、全米科学財団(NSF PHY-1734030、BPB)からも資金提供を受けた。本書で報告された研究は、米国国立衛生研究所(National Institutes of Health)のNational Center for Advancing Translational Sciences(賞番号TL1TR003019、EMA)およびSchmidt Transformative Technology Fund(EMA)からも助成を受けた。本研究は、Penn Skin Biology and Diseases Resource-based Center (P30-AR068589)およびペンシルバニア大学ペレルマン医学部から一部支援を受けた。内容はあくまで著者の責任であり、必ずしも各資金提供機関の公式見解を表すものではない。
倫理
ヒトを対象とした研究 ヒトを対象とした研究は、Princeton University IRB #13003のもと 、Princeton University Department of Molecular Biologyで実施された。健康なボランティアは、案内チラシで募集した。ボランティアはサンプル採取前にインフォームド・コンセントを行った。
すべてのマウスは公認動物施設で飼育・管理され、すべての実験はUSA Public Health Service Policy of Humane Care and Use of Laboratory Animalsに従って行われた。すべてのプロトコールは、Institutional Animal Care and Use Committeeのプロトコル#1867(Princeton University)により承認された。
編集委員
Bavesh D Kana、ウィットウォーターズランド大学、南アフリカ共和国
バージョン履歴
査読者へ送付: 2023年3月15日
プレプリント掲載 2023年5月3日(プレプリントを見る)
プレプリント掲載: 2023年5月19日(プレプリントを見る)
記録版発行 2023年6月30日(バージョン1)
著作権
© 2023, Acosta et al.
この記事はクリエイティブ・コモンズ 表示ライセンスの条件の下で配布されています。このライセンスは、原著者および出典がクレジットされていることを条件に、無制限の使用と再配布を許可するものです。
メトリクス
396
ページビュー
0
引用
(Monthly)Page Views010020030040006/2307/23
トグルチャート
毎日
月間
以下のソースの中で最も引用数が多いものをポーリングして作成した論文引用数: Cross, PubMed Central, Scopus.
リンクのダウンロード
カテゴリーとタグ
研究論文
微生物学と感染症
マイクロバイオーム
細菌生存率
表皮ブドウ球菌
アクネ菌
研究対象生物
大腸菌
ヒト
参考文献
計算生物学とシステム生物学
微生物学と感染症
HIVの未熟な格子の構造が、出芽したビリオンの中で格子のリモデリングを可能にする
シカオ・グオ, イプシータ・サハ... マーガレット・E・ジョンソン
研究論文 2023年7月21日更新
疫学・グローバルヘルス
微生物学・感染症学
近距離接触パターンとSARS-CoV-2との関連:家庭内伝播研究
ジャッキー・クラインハンス、ロレンツォ・ダラミーコ ... SA-S-HTSグループ
研究論文 2023年7月20日更新
微生物学・感染症学
HIV-1感染後のCARD8インフラマソーム活性化を促進するヒト特異的モチーフ
ジェシー・クルスプトラクル, エリザベス・A・ターコット... パトリック S ミッチェル
ショートレポート 2023年7月20日更新
eLifeの新しい記事をいち早く読む
アラートメールに登録する
プライバシーポリシー
eLifeについて
求人情報
提携企業
アラート
連絡先
契約条件
プライバシーポリシー
インサイド・ライフ
月刊アーカイブ
プレス向け
リソース
XMLとデータ
githubで検索
eLifeは、研究資金提供者に触発され、科学者が率いる非営利団体です。eLife Sciences Publications, Ltdは、米国デラウェア州で設立された有限責任非営利非株式会社で、会社番号は5030732、英国では会社番号FC030576、支店番号BR015634で登録されています:
イーライフサイエンスパブリケーションズ
ウェストブルックセンター、ミルトン・ロード
ケンブリッジ CB4 1YG
英国
© 2023 eLife Sciences Publications Ltd. 特に断りのない限り、クリエイティブ・コモンズ表示ライセンスに従う。ISBN: 2050-084x
