
あなたは誰?コンタミDNA物語
多くの問題点が指摘されていながら緊急承認ということで導入された遺伝子ワクチンを接種してから我が国の超過死亡が異常に増え続けており、ワクチン接種に反対する声が高まる一方、東京都は高齢者と医療従事者に6回目のワクチン接種を開始しました$${^{1)}}$$。
そんな中、ワクチンの危険性を訴え続けてきた荒川央氏がモデルナやファイザーのワクチンにDNAが混入している報告を紹介しました$${^{2)}}$$。オリジナルはアメリカのKevin McKernan氏の一連のブログです$${^{3-6)}}$$。
McKernan氏はシュードウリジンを含む遺伝子ワクチンでは製造過程で変異が高頻度で生じる可能性があり、その品質をチェックする目的でワクチンのRNAを逆転写酵素でDNAに変換しDeep Sequencingという手法で配列を調べていました$${^{4)}}$$。
Deep SequencingというのはDNAをバラバラにして個々のDNA配列を決定して、あとからコンピューターでつなぎあわせて配列を決める手法です。1本のDNAの配列を端から決めて行く従来の手法に比べて、同時進行で大量の配列を決めることができるので配列決定速度が飛躍的に速くなりました$${^{7)}}$$。
配列を調べること自体、製薬会社の特許保護のための法律にふれるそうで、McKernan氏はかなりの覚悟で公表に踏み切ったものと推察されます。
その過程でワクチンに含まれるmRNAからできるcDNAのほかに発現ベクターのDNAそのものが検出されたのです(図1)。

これらのプラスミドの配列はGene Bankに登録されています$${^{8,9)}}$$。ファイザーワクチンに見つかったプラスミドは7,810bp、モデルナワクチンに見つかったものは6,777bpです(図2)。

McKernan氏の検出したファイザーのコンタミプラスミド中にはスパイクタンパク質の1,268アミノ酸をコードする塩基配列が配備されています(表1)。

オミクロンはアミノ酸は受容体結合ドメインとN末に多くの変異がありオミクロンに対応した2価ワクチンに含まれるリルトジナメラン$${^{10)}}$$(図3, Pfizer (DB))はこれを模してしますが全て一致するわけでなくBA.1.1.529と比較するとアミノ酸レベルでは98%のホモロジーしかありません(表1)。

Mckernan氏の検出したプラスミドのスパイクタンパク質もオミクロン型でN末と受容体結合ドメインの変異を模していますがリルトジナメランと全て一致するわけではなく、アミノ酸も2つ足りません。塩基配列は武漢型のトジナメランやリルトジナメランと同様にGC含有量が高く57%で、ワクチンに特徴的なC末のアミノ酸の入れ替えも保持しています(図3, 表1)。
しかし、mRNAはワクチンにDNAが含まれることは専門家には周知の事実だったようです。mRNAワクチンはDNAを鋳型にしてウィルス由来のT7 RNAポリメラーゼを使った酵素反応で作りますから、反応生成物には鋳型に使ったDNAが存在するのです。
鋳型DNAは大腸菌にプラスミドを形質転換して作ります。大腸菌で大量に作られたプラスミドは精製したのち特異的な配列を認識して切断する制限酵素で直鎖DNAにしてから鋳型に使うのです。もちろん生成物にDNAが入っては困るので、これをDNase Iという酵素でバラバラに壊して取り除くのです。
ファイザーのプラスミドには発がんウィルスのSV40のプロモーターまで含まれていました。SV40プロモーターは動物細胞に遺伝子を発現させるための配列です。鋳型配列を大腸菌で作るためには全く不要の配列であるばかりでなく、ヒトの細胞に導入された場合には、たとえプラスミドの断片であっても発がんにつながる恐れもあるのです$${^{2,11)}}$$。
政府はこれまでRNAワクチンはDNAワクチンと異なり、ゲノムに組み込まれることはないから安全だと喧伝してきました。しかし、ヒトの細胞にはRNAを逆転写できる酵素が存在するのです。この酵素で作られたワクチン由来のDNAがヒトのゲノムに組み込まれる危険性が指摘されてきました$${^{12)}}$$。
この確率は極めて低いことが予想され、ワクチンの安全性を覆すことにはならないとの意見もあります。今回見つかったプラスミドDNAはスパイクタンパク質そのものをコードしているのです。逆転写酵素などなくてもプラスミド由来のDNAがゲノムに組み込まれる可能性が出てきたのです。
EMA (Europe Medicines Agency: 欧州医薬品庁)やFDA (Food and Drug Administration: 米国食品医薬品局)はワクチンに含まれるDNA混入の基準値として10ng/dose、200bp以下と定めています。ファイザーのワクチンは30μg/dose、モデルナは50μg/doseですから、重量比でそれぞれ0.03%, 0.02%以下にすることが義務づけられています$${^{13)}}$$。
McKernan氏はワクチンがこの基準を満たしているかDNAの定量をしています。一つはQubit 3という吸光度を利用した定量です。核酸は260nm近辺の紫外線を吸収するのでその吸光度で定量することができます。しかし、RNAもDNAも同じ核酸ですからこの方法では二つを測り分けることはできません。
Qubit 3は特別な色素を使ってDNAとRNAの吸光度を測り分けることができる優れものです$${^{14)}}$$。McKernan氏はこの測定法でワクチンを測定し、ファイザーのワクチンの核酸含有量13μg/dose、DNAが0.64μg/dose、DNA含有率5.7%、RNA/DNA=23と算出しました。モデルナワクチンは核酸含有量18μg/dose、DNAが0.93μg/dose、DNA含有率6.5%、RNA/DNA=28でした$${^{4,6)}}$$。
いずれも基準値を大幅に上回る値です。しかし、Qubit 3はDNAとRNAを測り分けることができると言ってもDNAとRNAを同時に測定するとRNAによる吸光度がDNAの吸光度に10%程度入り込んできます。RNAが20倍も存在する状況ではDNAの吸光度の半分はRNAのものですから、Qubit3の測定によるDNA量は過剰定量である可能性があります。
McKernan氏はAgilent Tape StationでもDNAとRNAを測り分けています。これはDeep Sequencing用の機械で核酸フラグメントの分布をみることができます。RNAとDNAは色素の違いで区別するところはQubit 3と同様です$${^{14)}}$$。
その結果、ファイザーのワクチンの核酸含有量は10μg/dose、DNAが2.6μg/dose、DNA含有率30%、RNA/DNA=3.1、モデルナワクチンは核酸含有量25μg/dose、DNAが4.9μg/dose、DNA含有率17%、RNA/DNA=4.6というとんでもない値でした(図4)$${^{4,6)}}$$。

この場合にもQuit 3同様にDNAの過剰評価の可能性がありますが、Tapeのフラグメント分布をみてみると4,300bのRNAが大量に存在するはずの2,200bp付近にはDNAのピークはありませんからこのシステムではDNAをきちんと測り分けているように見えます。
代わりに9,347bpにシングルピークが出ています。これはMcKernan氏が見いだしたプラスミド (図1)$${^{3)}}$$のピークであることが想定されます。
このことを確かめるためにMcKernan氏はワクチンを大腸菌に形質転換し、大腸菌内でこれらのプラスミドが増えるか確かめています。その結果、モデルナ、ファイザーいずれのワクチンからもプラスミドに由来するカナマイシン耐性形質をもったコロニーが現れ、そのDNAを解析すると大腸菌ゲノムDNAの他にプラスミドと考えられるバンドが検出できました(図5)$${^{4)}}$$。

DNase Iで処理してDNAをフラグメントにした場合のフラグメントの存在確率は理論的にはポアソン分布に従うはずです。DNAを平均mbpで切断したときにnbpのフラグメントに一つの切れ目もできない確率p、つまりintactである確率は次の式で表せます(図6)。
$${p=exp(-n/m)}$$
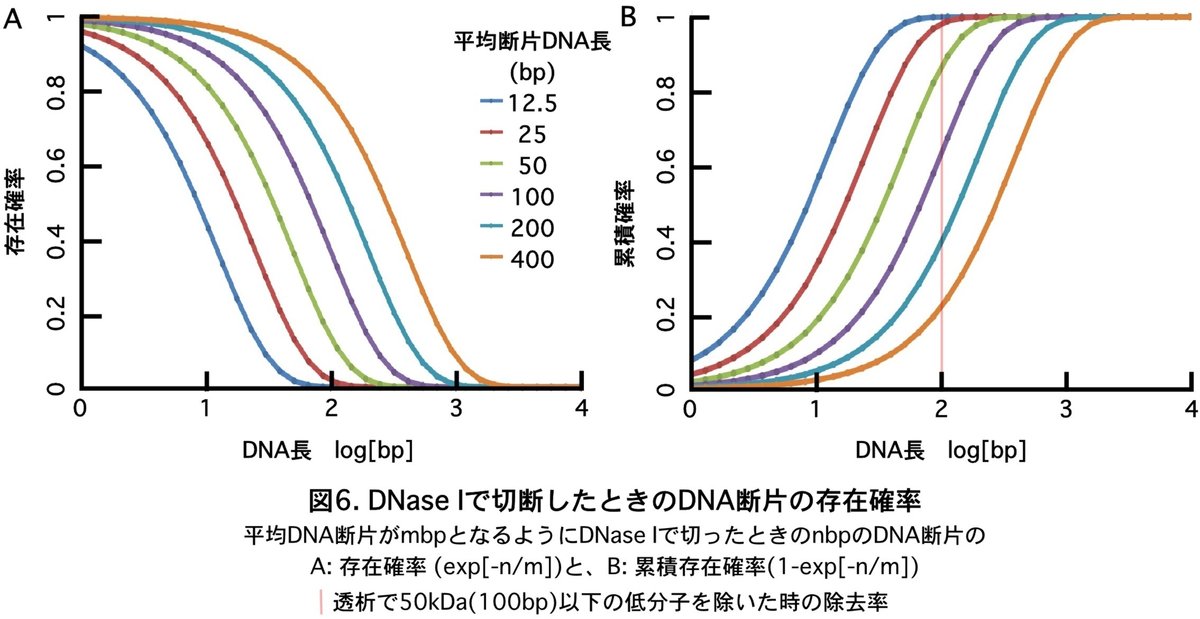
もし、製薬会社が規定通り平均200bp以下になるようにDNase処理をしているなら7,810bpのプラスミドDNAに傷がつかずにintactである確率は1.1x10$${^{-17}}$$になります。もし、ワクチンにAligent Tapeの示すように10μg/doseのような大量のDNAが入っていたとしても、3,866kDaのプラスミドの分子数は、
1x10$${^{-5}}$$g/3866,512x6.02x10$${^{23}}$$=1.56x10$${^{12}}$$個
(6.02x10$${^{23}}$$: アボガドロ数)
であり、intactなプラスミドがでてくる確率は
1.1x10$${^{-17}}$$x1.56x10$${^{12}}$$=1.7x10$${^{-5}}$$
にしかなりません。図5のような形質転換実験でコロニーがでてくる確率などほとんどゼロに近いしintactなプラスミドバンドがでてくることなどあり得ないのです。東京大学医学部の新田剛氏はMckernan氏と同じ形質転換実験をモデルナワクチンについて行っていますが、コロニーは得たもののその中にプラスミドは検出できませんでした。検出できなくて当たり前なのです。
新田氏の得たコロニーは薬剤耐性遺伝子だけを組み込んだ大腸菌のコロニーなのではないかと思います。新田氏の使ったカナマイシン耐性遺伝子は795bpですから規定通り10ngの平均200bpで断片化したDNAが混入したとしても3000万分子/doseほど残っていることになります。大腸菌ゲノムに入り込んで発現する効率を考えてもコロニーが出てきてもおかしくはないと思います。
このことは断片化したDNAが大腸菌のゲノムに入り込むことが可能であることも示唆しています。
発がん因子として懸念されているSV40プロモーターはカナマイシン耐性遺伝子より小さな358bp程度のDNAです。自己免疫疾患をひきおこすことが懸念されている抗原ペプチドはアミノ酸20個程度のオリゴペプチドですから60bp程度です。これらがintactである確率は17%、74%であり1回の注射で数10億という大量の分子が体内の細胞に供給されることになるのです。
大腸菌のゲノムに組み込まれてタンパク質を発現するなら同じことがヒトの細胞でおきても不思議ではありません。intact DNAが入り込まなくてもDNAの断片がはいることでも様々な健康影響があらわれることが予想されます。このことは荒川氏が詳しく解説しています$${^{2,11)}}$$。
3つめの定量法としてMcKernan氏は定量的PCR (Quantitative Polymerase Chain Reaction: QPCR)を行っています。PCRでは目的とする標的DNAが1サイクル行うごとに2倍に増えます。ですからある一定の値になるまでに何サイクル (Ct)を要するか測定することでもともとあったDNAの量が測定できるのです。
例えばもともと10$${^8}$$分子あるDNAが10サイクル(10Ct)で基準値に達したとします。この時あるサンプルを同じ条件でPCRを行い、15サイクル (15Ct)で基準値に達したとします。
サンプルは基準値に達するまでに5サイクル増やす必要があったわけですから、中には2$${^{-5}}$$だけ少ないDNAが存在していたことになり、10$${^8}$$x2$${^{-5}}$$分子の標的DNAがあることが定量できます。
これをQPCR (Quantitative Polymerase Chain Reaction)の絶対定量といいます。
McKernan氏はプラスミド内の標的遺伝子として複製開始点 (origin) にあたる場所(ori: 106bp)とスパイクタンパク質をコードしている場所 (spike: 114bp)を選んで、8本のファイザーワクチンの絶対定量をしています$${^{5)}}$$。彼の示した検量線からワクチンが含むDNAの量を計算してみました(図7)。

その結果、ファイザーのワクチンに含まれるDNAの平均値 (g/μl)はorigin targetを使うと7.34x10$${^{-13}}$$、spike targetを使うと4.66x10$${^{-13}}$$という値が得られます。
この数字は標的DNAを定量した数字です。spike target (114bp)を使用した場合、7,810bpのプラスミド全体では7,810/114を掛けて1μl当たりのDNA量が計算できます。ファイザーの場合1doseは0.3mlです。平均200bpに切ったときの114bp targetの存在確率は0.57ですから、1μlあたりのDNA量に1 doseの液量を掛けて存在確率で割ると1 dose あたりのDNA量は
4.66x10$${^{-13}}$$x7810/114x300/0.57=1.70x10$${^{-8 }}$$g/dose=17ng/dose
となります。これはQubit 3とAgilent Tapeで測定した値の1/38および1/289で、基準値の10ng/doseはクリアしていません。
しかし、QPCRのDNAの定量はintactなtarget DNAの存在確率によって大きく変化しますからあてにならないのです。
平均4bpに分解することをDNase Iによる完全分解と定義します$${^{15)}}$$。もし完全分解しているならspike targetの存在確率はexp(-114/4)=4.2x10$${^{-13}}$$となり、DNA量は23kg/doseというあり得ない数字になります。
反対に、Agilent Tapeの結果が正しく、2.6μg/doseだと言うのならDNase I処理は平均20bp程度に切っていることを想定すれば辻褄のあう結果であるとも言えます。
QPCRの定量はintactなtarget DNAがどの程度存在しているかわからないと正確な値はわからないのです。
DNA断片の分布は様々な要因でポアソン分布の予想からずれてきます。ワクチン制作過程では低分子や高分子を除く操作をしていますから当然分布は大きく変化します。McKernan氏は合成したmRNAが鋳型DNAのマイナス鎖と強固に結合してDNase Iによる切断を阻害していることも予想しています$${^{16,17)}}$$。
したがって、プラスミドDNAをQPCRで定量するとspike targetを使った場合にはori targetを用いた場合に比べDNase Iに耐性を示します(図8)。事前にRNase AでくっついているmRNAを除いてやるとori target同様の感受性になります(図8A)。McKernan氏はモデルナワクチンではori targetでQPCRを行うとspike targetに比べCt値が多くなることも示しています(図9)$${^{4)}}$$。このことはモデルナワクチンでは共存するmRNAが鋳型DNAのDNase Iによる分解を部分的に阻害していたことを示唆します。

Agilent Tapeの結果(図4B)でもintact plasmidのピークと思しき9,340bpのピークは7,810bpのプラスミドのピークより明らかに高く4,278塩基のワクチンRNAとintact plasmidが会合したものであることを予想しています 。

DNase Iで処理し、精製して多くの断片が除去されているDNAと違い断片化の憂き目にあっていないmRNAはQPCRで正確に定量できるように思いますが、実はここにも大きな問題があります。mRNAをQPCRで定量するには一度mRNAを逆転写酵素でDNAにする必要があるのですが、逆転写酵素の効率は必ずしも高くないことが知られています。QPCRを逆転写してからspike targetで定量したMcKernan氏のデータ$${^{5,6)}}$$から計算するとファイザーワクチンのmRNA量は2μg/doseにしかなりません。mRNA量は30μg/ doseですから、逆転写効率は6.6%しかないことが推定されます。
要するに、DNAが断片化して均一な分子でない場合QPCRで正確な定量を行うのは不可能だと言うことです。ですからMcKernan氏が定量化の方法としてQubit 3やAligent Tape Stationを利用した$${^{4)}}$$のはそれぞれの欠点はあるものの妥当な選択であるとも言えます。
それにしても30%もDNAが混入することなどあり得るのでしょうか?なぜ鋳型DNA作製には必要のないSV40プロモーターの入った動物細胞用の発現ベクターを使っているのでしょうか?
ファイザーの申請書には鋳型DNAを作るためのプラスミドはSV40プロモーターなどない大腸菌用のプラスミドが記載されています(図10)$${^{18)}}$$。

大腸菌で複製するために必要なori、薬剤選択のためのカナマイシン耐性遺伝子、RNA合成に必要なT7プロモーター、Kozak配列がスパイク遺伝子の上流に配置され、下流にはmRNAを安定化させるためのFIエレメント、polyA tailが配備、1本鎖DNAに切るための制限酵素Eam1104Iの認識配列$${^{19)}}$$が配備されています(引用文献18, p.24)。
McKernan氏が示したプラスミドがコードする鋳型DNAもこれとほぼ同等の構造を持っています(図2,表2)。

それにしても、なぜ鋳型作成には必要ではない発がんリスクが推定されるSV40のプロモーターがあるのでしょうか?このプラスミドはopen reading frame (ORF: タンパク質を発現できる配列、ORFファインダー$${^{20)}}$$で探せます)を39個もコードしています。SV40 プロモーターの下流にはカナマイシン耐性遺伝子などいくつかのORFがありますが (図1B)、これらを細胞で発現させることが真の目的かもしれません。
この申請書$${^{18)}}$$はMcKernan氏が基準値を示すための引用文献として紹介していたものですが、鋳型mRNAワクチンの作製法についての記述もあります。
それによると、1)大腸菌にpST4-1525を入れてプラスミドDNAを作り、2)制限酵素Eam1104Iを使って1本鎖にし、3)RNAポリメラーゼでmRNAを作り、3)鋳型DNAをDNAase Iでこわし、4)プロテアーゼで処理し、5)硫安分画を行い、6) 限外濾過により低分子を除き、7)精製したRNAをLNPにつつみ、8)製品化する、といった手順を踏むようです。
限外濾過は300kDa MWCOですが、この透析膜を使うと分子量50,000(100bp)以下の低分子DNAはほぼ完全に除去できます$${^{21)}}$$。もしDNase処理が不完全で平均DNA断片が200bpなら100bp以下の断片を除いてもDNAは9%程度しか除けませんが、しっかり反応させて平均4bpにまでしてやれば除去率は99.9999996%となります$${^{22)}}$$。
この場合、たとえ鋳型DNAを生成RNAの1/3の10μg/doseで使用していたとしても製品化したワクチンには4fg/dose (4x10$${^{-15}}$$g/dose)のDNAしか入っていないことになり、基準値は十分にクリアできるはずです。RNAの合成には最終生成物の0.1-1%の鋳型DNAがあれば事足りるようですから、きちんと精製されていればDNAが30%も入っていることありえないように思います。
もし入っていたとしたら、mRNAとの結合によりDNase Iが効かなかったか、精製後に誰かが何らかの理由で投入したか?
これらの可能性はMcKernan氏が行ったようなAgilent Tapeでの分析で見えてくるでしょう。Agilent Tapeのような高級品を使わなくても10%ものDNAが壊れずに入っていたら普通にアガロースのゲルで見えるでしょう。またDNase Iで切れていないのなら、大腸菌に形質転換すれは山のようにコロニーがでてくるはずです。でてきたコロニーのDNAを精製して配列を決めるのも難しい技術ではありません。
McKernan氏が見つけたSV40発現ベクターを検出するならpST4-1525には存在しない、SV40 プロモーターのユニークな配列を標的としてPCRをやればよいのです。アガロース電気泳動による分析、大腸菌での形質転換、SV40 targetを使ったPCR、はいずれも次世代シークエンサーのような高い機械がなくても普通の町医者でもできる安上がりのテストです。
国は第6回目のワクチン接種を始めましたが、ワクチンを推奨しこれを接種するなら国や医者は患者にこれらの情報をしっかり伝えるインフォームドコンセントの義務を担っているのです。ワクチン接種の前にこれら3つの試験ぐらいは行って安全性を確認する必要があるのでないでしょうか?
引用文献
1)産経新聞 (2023/5/11)
2)荒川央、note (2023/4/5)
3)Anandamide, Substack (2023/2/16)
4)Anandamide, Substack (2023/3/9)
5)Anandamide, Substack (2023/3/30)
6) Anandamide, Substack (2023/4/11)
7)荒川央、note (2023/4/23)
8)McKernan KJ et al, GenBank, OR134577 (2023)
9)McKernan KJ et al, GenBank, OR134578 (2023/6/20)
10)コミナティー医薬品インタビューフォーム 13版 (2023)
11)荒川央、note (2023/4/14)
12)荒川央、note (2022/3/6)
13)Yang H, PDA J Pharm Sci Tech, 67:155 (2013)
14)荒川央、note (2023/5/2)
15)New England Biolab, 製品カタログ
16)Anandamide, Substack (2023/4/15)
17)荒川央、note (2023/5/10)
18)Rapporteur Rolling Review critical assessment report (2020/11/19)
19)Thermo Fisher Scientific 製品カタログ
20)NCBI, Open Reading Frame Finder
21)日本ポール限外濾過解説
22) mbi, note (2023/6/15)
