
HADを使ってみた:10.平均値の差の検定…の前に
平均値の差の検定,つまりt検定や分散分析は,統計分析の定番のひとつですが,それについて書く前に,フィルタ設定とHAD2Rコードのちょっとした注意点について触れておきましょう。ざっくりまとめると,フィルタ設定を使うときは,出力されるフィルタ設定情報に常に注意,となります。そうか,だからこその赤字なのかもしれませんね。納得です。(note では赤字にすることができない…のですよね?)
HADにはt検定がない?
HADの分析メニューで,「平均値の推定」の次にあるのは「平均値の差の検定」です。これはいわゆる「t検定」などに相当するのですが,分析メニューには「t検定」という文字は出てきません。面白いですね。統計法の教科書などでは,たいてい,2群の比較(男性と女性とか)ならt検定,3群以上の比較(1年生から3年生とか,30代から60代まで4段階とか)なら分散分析とか,分けて説明されています。手で計算するときには,やっている計算がずいぶん違う感じもするのですが,実際には,どちらの検定も「すべての水準の平均値に差はない」を帰無仮説とした検定ですので,同じ目的の分析をしているともいえるわけですね。
なので,たとえば性別の差を検討しようとして,男性と女性の2水準だからt検定が行われるという期待をもってOKクリックしたのに,なぜか3水準で分散分析の結果が出力されたりすると,一瞬パニックになったりします。性別で「その他」を選択した人がいたのをうっかりするとこうなりますね。性別だから2水準だ,という「思い込み」がパニックの原因だったりするわけです。心理学を学ぶものがそんなんでいいのか? などというつっこみは,今は脇に置いておくとして,性別で「その他」を選んだ人のこと,あなたは忘れてたでしょ,と静かに指摘してくれるHADはえらい,ということにしておきましょう。
とりあえず検定してみる
何はともあれ,やってみましょう。t検定も分散分析も,もう何度もやってみてはいるのですが,「あれこれ試してみる」モードでやっていると,いろいろ見つかることもあるのですね。
データは iris を使います。手順は次の通りです。
使用変数を設定(従属変数が前,独立変数が後)
⇒【分析】
⇒「平均値の差の検定」
⇒「対応なし」を選択(デフォルト)
従属変数は複数指定できる(!)のですが,その話はしばらくおいておいて,今回は従属変数を「Sepal.L(がく長)」,独立変数を「Species(種)」としました。結果はこんな感じです。iris データで「種」は1~3の3種類ありますから,当然,3水準の分散分析が実行されますね。

HADの分散分析シートは,かなり多くの情報がどわっ!と出力されますから,見慣れるのに少々手間取った記憶があるのですが,それはまたのちの機会に書くとして,一番うれしかったのは,このプロットですね。これ。
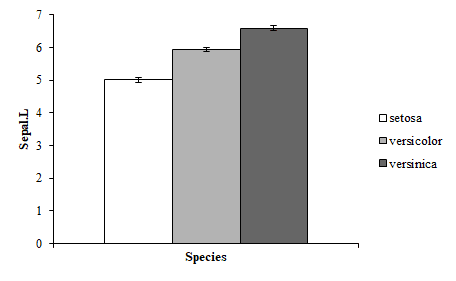
自動的にプロットを描いてくれてエラーバーまでつけてくれる。親切じゃありませんか。わたしが「HADっていいなあ」と思ったのは,ほぼ100%,このプロットのおかげです。ありがとう,HAD。これ,自分でエクセルで描こうとするとけっこう手間だし,卒業研究やっているころは,Rのプロットにエラーバーとかつける方法がわからなかった。
ちなみに,上のプロットのエラーバーは「標準誤差」を示しています。えー,95%CIじゃないのかよお,と思った場合は,次の手順で変更できます。
【HADの設定】⇒【グラフ設定】⇒「グラフの表示設定」または
【分析】⇒【グラフ設定】⇒「棒グラフ」
出力してしまった後でも,HADのプロットは要するにエクセルのグラフですから,「誤差範囲」の書式設定をいじってやれば好きなように変えられますね。
むりやりt検定してみる
さて,同じデータを使ってt検定をむりやり(?)やってみましょう。考え方としては,3種類ある「種」のうちの2種類だけを使って検定すればいい。そこで,「フィルタ」機能を使います。「Species」の行の【フィルタ】の列に,半角数字で「3」と入力します。これで,「Species が 3 のデータを使わないで分析する」という指定になります。
わたしは覚えられないので【フィルタ】ボタンをクリックしてGUIを使います。こんなGUIですよ。これでOKすると,上の画面と同じように設定されます。
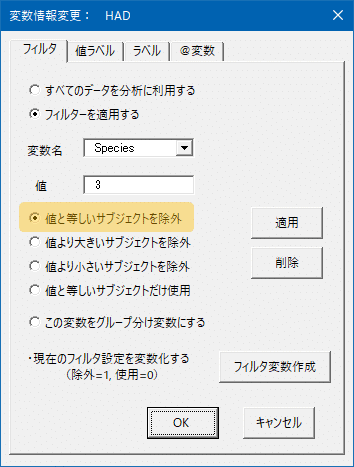
この状態で,さっきと同じ分析をします。
使用変数を設定(従属変数「Sepal.L」が前,独立変数「Species」が後)
⇒【分析】
⇒「平均値の差の検定」
⇒「対応なし」を選択(デフォルト)
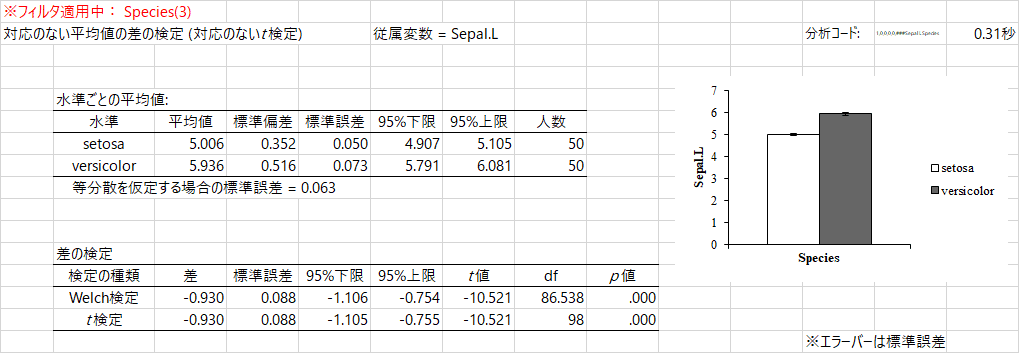
結果は上のようになりますね。同じメニューを実行しているのに,まったく別の出力。ここでもやはり,プロットが光りますね。たいそうありがたい。もう一つありがたいというか,そうだよな,と思ってしまうのは,Welch 検定とt検定を両方とも実行してくれていることです。両者の違いについて,専門的なことを論じるほど詳しくありませんが,2群の分散が等しいと仮定するのがt検定,そのような仮定をしないのがWelch検定ですね。
統計法の教科書の中には,まず分散が等しいかどうかを検定して,その結果をもとに,t検定かWelch検定か,適切な方を用いなさいという説明が,以前はされておりました。わたしがはじめて勉強した教科書でもそのような方向の説明がありましたが,そのような手順は不要で,常にWelch検定をするべきで,清水先生のブログでも,次のように書かれています。
よく用いられる方法に,等分散性の検定を行って,もし等分散が成り立っていれば普通のt検定,成り立っていなければWelch検定という手続きがあります。しかし,これについても青木先生のサイトのシミュレーションが明らかにしているように,Welchの検定は等分散が成り立っていても成り立っていなくても常に正しい結果を出すので,常にWelchの検定を行うべきです。また,等分散の検定→t検定は,検定の二重性の問題があり,有意水準を維持できなくなる問題があります。なので,二群の平均値の差の検定については,常にWelchの検定を利用しましょう。(https://norimune.net/1585)
言及されている青木先生のサイトはここです。
だったらどうしてt検定の結果も出しているのだ? という疑問も芽生えますが,まあ,いろいろと,事情もあるのかもしれません。
フィルタに注意
ところで,さきほどの出力結果の冒頭には,赤い文字でこんな表示がありました。
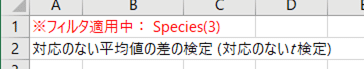
フィルタの設定をそのまま表示していますね。Species変数に「3」というフィルタが設定されている。つまり,Species=3 のデータが分析から除外されていることについて,注意喚起しています。しかし,このフィルタの設定には注意すべきことがあります。ここでは2つの場合に限ってみてみましょう。1つは,分析履歴の利用という面から。もう1つは,HAD2Rの利用という面から。
分析履歴はフィルタを記録しない
HADの出力には,必ず「分析コード」がついてきます。シートの右上の方に小さく出力されている,これですね。目立ってほしいので字を大きくして貼り付けてみましたよ。

数字で分析の設定が記録されていて,このコードをもとに,同じ分析を再現できるのです。HAD12.21から実装された機能です。使い方は,
分析コードが出力されたセルを右クリック
⇒メニューから「分析コードを実行」
または⇒「分析コードをHADに反映」をクリック
前者「分析コードを実行」を選択すると,同じ分析を黙って実行してくれます。このとき,モデリングフォームが現在どのように設定されているかに影響されず,また,同名のシートも上書きされません。これは便利。ただし,フィルタ設定は分析コードに含まれていません。ここ重要です。つまり,
フィルタ「 Species(3) 」で「散布図」分析を実行する⇒
「Scatter」シート《フィルタなし分析》が出力される⇒
「モデリング」シートでフィルタ設定を「 Species(1) 」に変更する⇒
先ほどの「Scatter」シートの「分析コード」《さっきフィルタなしで実行したよね?》を実行する⇒
フィルタ「 Species(1) 」で「散布図」分析が実行される
なので,「あのフィルタ設定をもう一度」という分析は,ただ「分析コード」を実行するだけではだめ。「別のフィルタ設定でもう一度」は,フィルタ設定を変更してから「分析コード」実行でOK。ちょいとややこしいね。くせを理解して,便利に使えば便利です。きっと。(当たり前か)
また,後者「分析コードをHADに反映」をクリックすると,使用変数の設定を分析コードに合わせて直してくれますが,実行はされず,モデリングシートに戻るだけ。なので,同じ変数を使って別のフィルタ,とか,別の分析,というときにはこっちが便利でしょう。
いずれにしても,このフィルタ設定が今後も重要,と考えるならば,データの不要な行を削除して,必要な行のデータを新たに作ってから(もとのデータを失わないように注意),分析を続行するのが賢いと思われます。
フィルタをデータに反映させる
ついでなので,HADを使ってそれを実行する方法を整理しておきます。いま,iris のデータから,Species=3 のデータだけを除外して分析を続けたいとします。「 Species(3) 」というフィルタを使い続けるよりも,Species=3 の行を除外した新しいデータを作るほうが,その後の分析での単純ミスを減らせます。その手順は,
使用変数に,今後必要な変数をすべて設定
⇒フィルタ「Species=3」を設定
⇒【データセット】
⇒【データセットの出力】
⇒一番上「選択した変数を出力する」にチェックしてOK
すると,「DataOutput」というシートにこんなデータが出力されます。
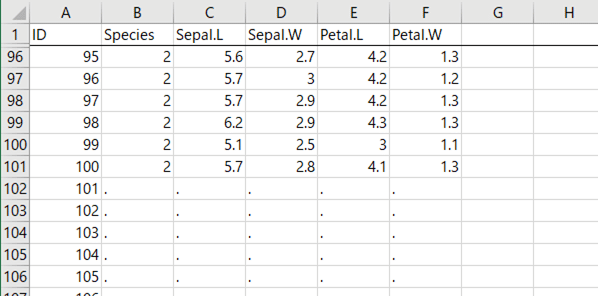
上の図では2~95行を非表示にしています。もとのデータでは,101行目からSpecies=3 のデータが並んでいるのですが,その部分がすべて欠損値になっていますね。これを新たなデータセットとして使うことにします。手順の続き。
出力された変数のすべての列を選択
⇒選択範囲で右クリック
⇒「メインデータセットとして利用」をクリック
すると,「データ」シートに,新たに作成した変数が挿入されます。以前のデータは右側に残っています。この例では,H列が空白になっていますので,それより右は,分析対象のデータとみなされません。
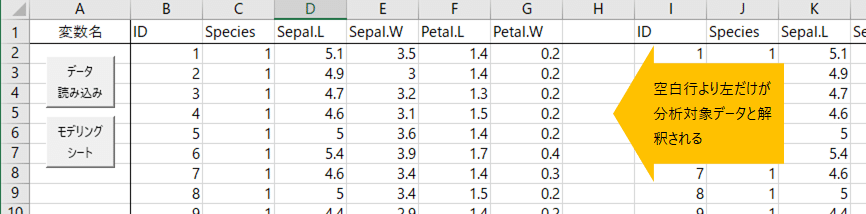
【データ読み込み】をクリックして,分析を続けることができます。この状態で,さきほどと同じ分析をすると,フィルタの設定をしなくても,t検定が実行されます。上記の手順で,Species=3 のデータがすべて欠損値になったため,Species が2水準になったからですね。めでたし。
ほかにも,さきほどのフィルタ設定のGUIで,「フィルタ変数作成」をクリックし,出力された変数をデータに追加して,その変数に「1」のフィルタを設定するという方法もあります。いずれにしても,どこまでの対象者に対して分析をしているかに,常に注意を払う必要がありますね。
HAD2Rもフィルタを記録しない
さいごに,HAD2Rについてです。平均値の差の検定をすると,次のようなコードが出力されます。2水準の場合と3水準の場合にはコードが当然違うのですが,これは2水準のとき,つまりt検定のときのコードです。
dat <- read.csv(".../csvdata.csv")
t.test(Sepal.L~Species, dat)
で,最初に小さい声で(?)書いておきますと,上記の出力で「~」(チルダ演算子)が全角になっていますが,これはエラーです。半角の「~」を使わないと正しく分析できません(小さい声でといいながら太字にしてしまいました。大事なので)。それから,すでに明らかなのですが,Species はもともと3水準の変数ですから,上記のコードでは,t検定はできません。フィルタ設定がコードに反映されていないため,Species は3水準のままだからです(フィルタを反映させた新しいデータセットならOKですが)。

フィルタ設定(Species=3 を除外)を反映されるためには,たとえば次のように書く必要があります。チルダ演算子も半角に直しておくと,
subdat <- dat[dat$Species!=3, ]
t.test(Sepal.L ~ Species, subdat)
また,Rのt検定は,t.test という名前ですが,デフォルトでWelch検定を行います。通常のt検定を行いたいときには,var.equal = TRUE というオプション設定が必要です。まあ,実質,Welch検定だけで用が足りると言っているので不要と言えば不要ですが,どうしても使いたい場合は,オプションの設定が必要です。
ということで,3水準の Species を独立変数にしてむりやりにt検定をやろうとした結果,フィルタの設定についていろいろと分かったことがあって長くなってしまったので,今回はここまで。t検定と分散分析については別に書くことにしましょう。
