SARS-CoV-2の変異を修復するnsp14
---
※ この記事は個人の見解であり、所属機関を代表するものではありません。
※ この記事に特定の個人や団体を貶める意図はありません。
※ 文責は、全て翡翠個人にあります。
---
SARS-CoV-2の持つ『nsp14(その機能から「3′→5′ exoribonuclease」とも呼ばれる)』について。

(画像:https://www.nature.com/articles/s41586-020-2286-9/figures/1)
10月13〜16日に開催された日本人類遺伝学会において、国立遺伝学研究所の井ノ上逸朗教授らの研究チームは、SARS-CoV-2の「第5波」の急激な収束には、デルタ株でウイルスのRNAゲノムの変異を修復する(exoribonuclease活性を持つ)nsp14が変化し、働きが落ちたことが影響した可能性があるとの研究結果を発表した。

(画像:https://www.sankeibiz.jp/econome/photos/211010/ecb2110100928002-p1.htm)
大会公式プログラムより(Oral English Session)。↓

(画像:https://www.jshg-jsgdt2021.org/files/program/program8.pdf)
大手メディアによる報道。↓(10月30, 31日)

(画像:https://www.chunichi.co.jp/article_photo/list?article_id=357277&pid=1648317)
井ノ上逸朗教授は「修復が追いつかず死滅していったのではないか」と指摘する。
https://www.sankei.com/article/20211030-WBVHCYJCZVNXXMIHON7AN6MBDI/
「エラー・カタストロフ」仮説。(9月29日)
◆どうして急速に感染者が減少してきたのか。いろいろな説がある。2回のワクチン接種者が60%程度となり、感染が落ち着いてきたという説。集団免疫ができあがってきたという説。もう一つ面白いのは「エラーカタストロフ」仮説というのがある
◆ウイルス学者には常識だろうが、一般の人たちには聞き慣れない言葉である。ある報道番組で、東大の児玉達彦名誉教授が唱えていた。新型コロナウイルスは一本鎖のRNAウイルス。RNAウイルスは複製が早いと、複製エラーが多数発生し、ウイルスが生存できなくなり、自壊が開始されるというもの
https://www.yakuji.co.jp/entry89668.html
私は、11月16〜18日に開催された日本ウイルス学会に参加した。
プログラムは非公開(閲覧するためにはパスワードが必要)であるが、この議論において重要であることから、これを一部公開する。
この学会で「nsp14のアミノ酸置換(=校正機能の不全)」に関する発表は2つ。井ノ上逸朗教授らの発表はなかった。
口頭発表:O6-20 ↓

シンポジウム:S4-07 ↓

(画像:https://web.apollon.nta.co.jp/jsv68/pdf_abstracts.html)
【基礎知識】nsp14を持つコロナウイルスの変異速度は遅い。
世界各地のSARS-CoV-2のゲノム配列が解読され,2020 年 10 月 20 日現在で 109,794 患者の SARS-CoV-2ゲノム配列(ゲノム分子疫学に適正な完全長配列 ; complete, high coverage) が GISAID に登録されている 1).SARS-CoV-2の変異速度は現在のところ25.6塩基変異/ゲノム/年(つまり,1 年間で平均25.6箇所の変異が見込まれる)と推定されており(図1: https://nextstrain.org/ncov/gisaid/global?l=clock),これはゲノム情報が収集され始めた2020年3月時点の数値から大きく変動はない.コロナウイルス全般でNsp-14 エキソヌクレアーゼ活性によるproof-reading 校正機能をもったRNA依存性RNAポリメラーゼ(RdRp)を有しており 2),他のRNAウイルスと比べても変異(進化)速度は遅いほうに分類される.
http://jsv.umin.jp/journal/v70-2pdf/virus70-2_147-154.pdf
12月28日現在、23.2塩基変異/ゲノム/年(図中黒色の線)。

(画像:https://nextstrain.org/ncov/gisaid/global?l=clock)
SARS-CoV-2のゲノムRNAの全長約3万塩基中の約23塩基。
【疑問】nsp14のアミノ酸置換(P203L:203番目のプロリン(P)がロイシン(L)に変異したという意)により、変異速度はどう変化するか?
>YP_009725309.1 3'-to-5' exonuclease [Severe acute respiratory syndrome coronavirus 2]
AENVTGLFKDCSKVITGLHPTQAPTHLSVDTKFKTEGLCVDIPGIPKDMTYRRLISMMGFKMNYQVNGYPNMFITREEAIRHVRAWIGFDVEGCHATREAVGTNLPLQLGFSTGVNLVAVPTGYVDTPNNTDFSRVSAKPPPGDQFKHLIPLMYKGLPWNVVRIKIVQMLSDTLKNLSDRVVFVLWAHGFELTSMKYFVKIG|P《L》ERTCCLCDRRATCFSTASDTYACWHHSIGFDYVYNPFMIDVQQWGFTGNLQSNHDLYCQVHGNAHVASCDAIMTRCLAVHECFVKRVDWTIEYPIIGDELKINAACRKVQHMVVKAALLADKFPVLHDIGNPKAIKCVPQADVEWKFYDAQPCSDKAYKIEELFYSYATHSDKFTDGVCLFWNCNVDRYPANSIVCRFDTRVLSNLNLPGCDGGSLYVNKHAFHTPAFDKSAFVNLKQLPFFYYSDSPCESHGKQVVSDIDYVPLKSATCITRCNLGGAVCRHHANEYRLYLDAYNMMISAGFSLWVYKQFDTYNLWNTFTRLQ
https://www.ncbi.nlm.nih.gov/protein/YP_009725309.1
nsp14はnsp10(赤色)と結合し、協調的に働く。

(画像:https://www.pnas.org/content/112/30/9436.long)
日本ウイルス学会で発表を行った研究チームの研究結果は、昨年末にプレプリントサーバー(bioRxiv)で公開されているため、これを紹介・解説する。
Genomic diversity of SARS-CoV-2 can be accelerated by a mutation in the nsp14 gene
Kosuke Takada, Mahoko Takahashi Ueda, Tokiko Watanabe, So Nakagawa
doi: https://doi.org/10.1101/2020.12.23.424231
Posted December 26, 2020.(投稿日)
※ 日本ウイルス学会での発表内容とほぼ同じであった。
nsp14(ExoN)のうち、変異の修復に関わるアミノ酸(90D/92E、191E、273D)。↓

(画像:Figure 2B)
203Pは変異の修復に関わるアミノ酸ではないが、nsp10と直接結合する部位(200K, 201I)に近く、P203Lは間接的にnsp10との結合を減少させる。

(画像:Supplementary figure 6B)
変異速度は、203Pでは19.8塩基/年であったが、P203Lでは35.9塩基/年となった。↓

(画像:Table 2)

(画像:Figure 3)
結論:nsp14のP203Lなどのアミノ酸置換は、SARS-CoV-2の変異速度を増大させる可能性がある。
---
個人的な見解:SARS-CoV-2のゲノムの長さは約3万塩基である。約20塩基/年の変異速度が2倍になったところで「自滅」するのだろうか?
---
井ノ上逸朗教授らの解析結果。↓
nsp14の遺伝子が変化したウイルスでは、ゲノムの変異が通常の10〜20倍あった。
https://www.chunichi.co.jp/article/357277
【疑問】変異速度が10〜20倍になると「自滅」するのだろうか?
2010年のSARS-CoVの研究を紹介する。
PLoS Pathog. 2010 May 6;6(5):e1000896. doi: 10.1371/journal.ppat.1000896.
Infidelity of SARS-CoV Nsp14-exonuclease mutant virus replication is revealed by complete genome sequencing
https://journals.plos.org/plospathogens/article?id=10.1371/journal.ppat.1000896
nsp14(ExoN)の変異の修復に関わるアミノ酸のうち、90D(アスパラギン酸)、92E(グルタミン酸)をアラニン(A)に置換し、D90A, E92Aの2つの変異を持つウイルスを「S-ExoN1」と名付けた。

(画像:Figure 1)
これらのアミノ酸はコロナウイルスで高度に保存されている。

(画像:https://www.biorxiv.org/content/10.1101/2020.12.23.424231v1)
S-ExoN1では、変異頻度が20.7倍に増加した。

(画像:Figure 4B)
SARS-CoVとS-ExoN1の増殖能を比較するため、それぞれのウイルスをVero細胞に感染させた。感染から20時間後までの培養液中に産生されるウイルス量は同じであったが、30〜36時間後のピークでは、S-ExoN1のウイルス量は通常のSARS-CoVの4分の1に減少した。

(画像:Figure 3A)
結論:変異の修復に関わる90D/92EはSARS-CoVの増殖に必須ではないが、このアミノ酸置換により変異頻度は増大し、ウイルスの増殖が損なわれた。
---
個人的な見解:P203Lとは異なり、D90A, E92AでExoN活性を直接的に失わせ、変異頻度が20倍以上に増加しても、ピーク時のウイルス量の減少は4分の1程度か、という印象。
---
東京大学の黒木登志夫名誉教授の解説。(京都大学の山中伸弥教授の「山中伸弥による新型コロナウイルス情報発信」にまとめがある。)
日本で流行していた第5波のデルタ株は、デルタ株の亜株 AY.29 でした。この株は日本の 95%以上を占め、日本独自のウイルス株です。AY.29 には、ウイルスゲノムの修復遺伝子 nsp14 に変異が入っているため、ゲノム上の変異を修復できず、変異が蓄積し、ウイルス が自滅したものと思われます。
https://shard.toriaez.jp/q1541/015.pdf
Genomic Surveillance in Japan of AY.29—A New Sub-lineage of SARS-CoV-2 Delta Variant with C5239T and T5514C Mutations
Takashi Abe, Masanori Arita
doi: https://doi.org/10.1101/2021.09.20.21263869
Posted October 08, 2021.
AY.29は、nsp14に「A394V」という変異を持つ。
● AY.29: A394V 変異。
https://shard.toriaez.jp/q1541/015.pdf
>YP_009725309.1 3'-to-5' exonuclease [Severe acute respiratory syndrome coronavirus 2]
AENVTGLFKDCSKVITGLHPTQAPTHLSVDTKFKTEGLCVDIPGIPKDMTYRRLISMMGFKMNYQVNGYPNMFITREEAIRHVRAWIGFDVEGCHATREAVGTNLPLQLGFSTGVNLVAVPTGYVDTPNNTDFSRVSAKPPPGDQFKHLIPLMYKGLPWNVVRIKIVQMLSDTLKNLSDRVVFVLWAHGFELTSMKYFVKIGPERTCCLCDRRATCFSTASDTYACWHHSIGFDYVYNPFMIDVQQWGFTGNLQSNHDLYCQVHGNAHVASCDAIMTRCLAVHECFVKRVDWTIEYPIIGDELKINAACRKVQHMVVKAALLADKFPVLHDIGNPKAIKCVPQADVEWKFYDAQPCSDKAYKIEELFYSYATHSDKFTDGVCLFWNCNVDRYP|A《V》NSIVCRFDTRVLSNLNLPGCDGGSLYVNKHAFHTPAFDKSAFVNLKQLPFFYYSDSPCESHGKQVVSDIDYVPLKSATCITRCNLGGAVCRHHANEYRLYLDAYNMMISAGFSLWVYKQFDTYNLWNTFTRLQ
https://www.ncbi.nlm.nih.gov/protein/YP_009725309.1
394A(図中赤囲み)は、変異を修復するexoribonuclease(ExoN)の働きを持つ部位ではない。
nsp14の後半部分は、グアニン-N7-メチルトランスフェラーゼ(N7-MTase)活性と呼ばれる別の機能を持つ。それぞれの機能は独立している。

(画像:https://journals.asm.org/doi/10.1128/JVI.01246-20)
N7-MTase活性部位の周辺。↓
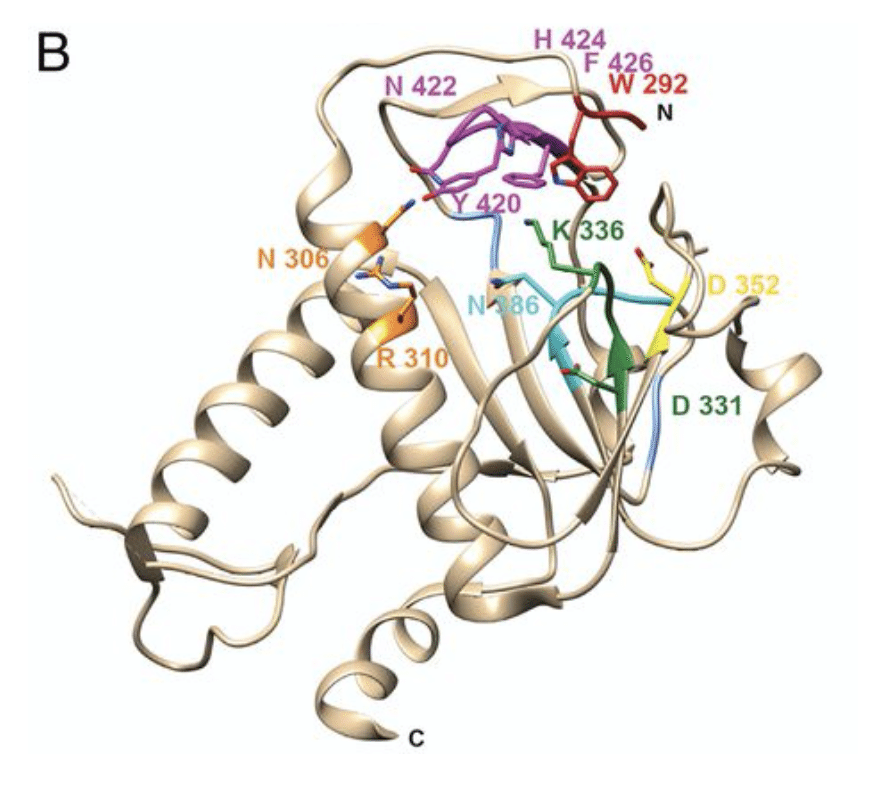
(画像:https://www.pnas.org/content/118/49/e2108709118)
---
個人的な見解:「394A」の位置を考えると、A394Vは、nsp14の変異の修復とは別の機能(=N7-MTase活性。ウイルスRNAのキャップ構造のグアニンN7位をメチル化し、真核細胞のmRNAに共通した構造を形成することにより、宿主細胞の翻訳機構を「ハイジャック」する)を低下させる可能性がある。

(画像:https://febs.onlinelibrary.wiley.com/doi/10.1111/febs.15815)
---
まとめ。
個人的な見解:この「エラー・カタストロフ」仮説は、かなり情報が錯綜しているように思う。その原因の一つが「変異の位置の誤解」であると思う。
日本独自の「AY.29」における変異は「ORF1ab:V1750A」である。
Therefore, mutations C5239T and T5514C (ORF1ab: V1750A) are unique to Japan.
https://www.medrxiv.org/content/10.1101/2021.09.20.21263869v3.full
「ORF1ab:V1750A」は「nsp3:932V」に相当し、「nsp14:394A」とは全く異なる位置にある。
AY.29に限らず、同じデルタ系統から派生した「AY.1」,「AY.3」,「AY.4」なども「nsp14:A394V」を持つ。↓

(画像:https://covdb.stanford.edu/page/mutation-viewer/#delta)
「AY.4」は、イギリスにおける流行株であった。

(画像:http://www.tokyo-eiken.go.jp/lb_virus/worldmutation/)
黒木登志夫名誉教授と井ノ上逸朗教授は、「nsp14:A394V」がAY.29の日本独自の変異であると誤解している可能性がある。
井ノ上逸朗によると、nsp14 の変異は次の3つの変異株に見られるという。
● B1.1.214(第3波):P43L 変異。
● R.1: P412H 変異.(R.1 は、図2の第4波の最初に見られる⻩色の小さなピーク。
B.1.1.316 の亜株。日本が70%を占めている。)
● AY.29: A394V 変異。
https://shard.toriaez.jp/q1541/015.pdf

【疑問】A394Vは日本における第5波の急激な収束を説明できるのか?
フランスAFMBのウイルス学者、Bruno Canard博士のツイートを紹介する。↓
Sorry: it is EXTREMELY UNLIKELY that A394V mutation in nsp14 has any responsibility in a potential 'error catastrophe extinction' in Delta/Japan. A394A is in the N7-MTase, remote from ExoN. There must be another explanation @hervenirom @SFV_France. https://t.co/sOtbfn9mtK
— BrunoCanard Virology (@canardbruno) December 9, 2021
Sorry: it is EXTREMELY UNLIKELY that A394V mutation in nsp14 has any responsibility in a potential 'error catastrophe extinction' in Delta/Japan. A394A is in the N7-MTase, remote from ExoN. There must be another explanation.
訳)申し訳ありませんが、nsp14のA394V変異が日本におけるデルタ株の「エラー・カタストロフによる死滅」に関与している可能性は極めて低いと思われます。A394VはN7-MTaseにあり、ExoNから離れた場所にあります。他の説明があるはずです。
https://twitter.com/canardbruno/status/1469047107239792641?s=20
全くその通りだと思う。
奇しくも「N7-MTase活性部位の周辺」の図で引用した論文の共著者(監修担当)であった。
Proc Natl Acad Sci U S A. 2021 Dec 7;118(49):e2108709118. doi: 10.1073/pnas.2108709118.
Structure-function analysis of the nsp14 N7-guanine methyltransferase reveals an essential role in Betacoronavirus replication
Natacha S Ogando, Priscila El Kazzi, Jessika C Zevenhoven-Dobbe, Brenda W Bontes, Alice Decombe, Clara C Posthuma, Volker Thiel, Bruno Canard, François Ferron, Etienne Decroly, Eric J Snijder
https://www.pnas.org/content/118/49/e2108709118
米国科学アカデミー紀要(PNAS)に掲載されたnsp14 N7-MTaseに関する論文の監修を担当した研究者の意見ほど、信頼できるものはないだろう。
児玉達彦名誉教授は、第5波の急激な収束の主な理由について、「2回のワクチン接種者が60%程度となり、感染が落ち着いてきたという説」、「集団免疫ができあがってきたという説」というヒト側の要因と、「エラー・カタストロフ仮説」というウイルス側の要因を提示した。
◆どうして急速に感染者が減少してきたのか。いろいろな説がある。2回のワクチン接種者が60%程度となり、感染が落ち着いてきたという説。集団免疫ができあがってきたという説。もう一つ面白いのは「エラーカタストロフ」仮説というのがある
https://www.yakuji.co.jp/entry89668.html
イギリスICLの小野昌弘准教授の言うように、「エラー・カタストロフ」仮説の根拠が薄弱であるならば、「ヒト側の要因」に重きを置くべきではないだろうか。
しかしながらデルタの死滅・自壊説は自己矛盾しており、このようなものが存在するとは考えられない。またこのような根拠薄弱の言説は、コロナへの対策のために重要な事実を見えないようにしてしまうがゆえに社会に有害であると考える。
https://news.yahoo.co.jp/byline/onomasahiro/20211111-00267460
適切な理解に基づいた対策が行われることを心から願っている。
