
第2章「血液型と性格には関連がある?」のベイズモデリングをPyMC Ver.5 で
この記事は、テキスト「たのしいベイズモデリング」の第2章「血液型と性格には関連がある?」のベイズモデルを用いて、PyMC Ver.5で「実験的」に実装する様子を描いた統計ドキュメンタリーです。
今回は初学者に優しい入門編的なモデルです。
果たして血液型と性格には関連があるのでしょうか?
PyMCでモデリングして、ベイズ推論を楽しみましょう!
テキストの紹介、引用表記、シリーズまえがき、PyMC等のバージョン情報は、このリンクの記事をご参照ください。
テキストで使用するデータは、R・Stan等のサンプルスクリプトとともに、出版社サイトからダウンロードして取得できます。
サマリー
テキストの概要
執筆者 : 下司忠大 先生
モデル難易度: ★・・・・ (やさしい)
自己評価
評点
$$
\begin{array}{c:c:c}
実装精度 & ★★★★★& GoooD!\\
結果再現度 & ★★★★・& あと1歩\\
楽しさ & ★★★★★ & 楽しい! \\
\end{array}
$$

評価ポイント
「血液型と性格の関係」という興味津々のデータ分析なので、モデリングから結果の分析まで楽しく取り組めました。
テキストにモデルの数式が詳しく掲載されているので、容易にPyMCのモデリングができました。
テキストの推定値と結果が異なっていますが、原因が分からず悩んでいます。

工夫・喜び・反省
「たのしいベイズモデリング」の実践で初めて納得できるPyMCモデルになりました。(第1章がラスボスだったこともあり)
モデルの概要
テキストの調査・実験の概要
血液型と性格の間に統計学的に有意な関連を示した論文(Tsuchimine, Saruwatari, Kaneda, & Yasui-Furukori (2015))のデータを用いて、血液型による性格「持続」の差が実質的に意味のある差かどうかをベイズ推論で調べます。

【ちょっぴり補足】
元となった論文では、「持続」の性格について、A型は他の血液型に比べて平均値が有意に高かった、としているそうです。
これはざっくり「A型は他の血液型に比べて持続的な性格である」ということのようです。
経験的で何となくのイメージですが、A型は我慢強さがありそうなので、持続の性格が強い印象があります(あくまで個人の感想です)。
テキストの調査概要に戻ります。
性格「持続」に関する質問数が8つあり、はいの回答が1点、いいえの回答が0点でカウントされ、データには、回答者(データ1行)ごとに血液型と得点(満点8)が記録されています。
8つの質問の内容はテキストに掲載されています。
また、こちらの論文のpdfファイルからも確認できます。
テキストのモデリング
目的変数$${y_{ji}}$$は「持続」の得点であり、独立に正規分布に従うと仮定しています。
正規分布のパラメータは、平均$${\mu_j}$$が血液型ごとの平均、標準偏差が$${\sigma_e}$$です。
関心があるパラメータは、この血液型ごとの平均パラメータ$${\mu_i}$$です。
$$
y_{ji} \sim \text{Normal}(\mu_j,\ \sigma_e) \\
\mu_{j} \sim \text{Uniform}(0, 100) \\
\sigma_e \sim \text{Uniform}(0, 100) \\
$$
添字$${j}$$は水準のラベルであり、血液型$${\{A, B, O, AB\}}$$です。
添字$${i}$$は血液型ごとのデータ番号です。
例えばA型の5番目の得点は$${y_{A5}}$$です。
パラメータ$${\mu_{j},\ \sigma_e}$$の事前分布は十分に広い範囲の一様分布を仮定しています。
■分析・分析結果
「持続」の性格について、A型とO型、A型とAB型で差があるかどうかを、事後分布からサンプリングした平均パラメータ$${\boldsymbol{\mu_i}}$$~血液型別の持続得点の平均データを用いて分析します。
効果量$${\delta}$$、非重複度$${U_3}$$、優越率$${\pi_d}$$という概念が出現します!
分析結果はテキストに記載のとおりです。
PyMCモデリングでの分析結果は、「PyMC実装」章の【分析】の部分をご覧ください。

PyMC実装
Let's enjoy PyMC & Python !
準備・データ確認
1.インポート
### インポート
# ユーティリティ
import pickle
# 数値・確率計算
import pandas as pd
import numpy as np
import scipy.stats as stats
# PyMC
import pymc as pm
import pytensor.tensor as pt
import arviz as az
# 描画
import matplotlib.pyplot as plt
import seaborn as sns
plt.rcParams['font.family'] = 'Meiryo'
# ワーニング表示の抑制
import warnings
warnings.simplefilter('ignore')2.データの読み込み
csvファイルをpandasのデータフレームに読み込みます。
### データの読み込み
data = pd.read_csv('Blood.csv')
# カラム名を読み替え
data.columns = ['血液型', '血液型コード', '新奇性追求', '損害回避', '報酬依存',
'持続', '自己志向', '協調', '自己超越']
display(data)【実行結果】
血液型ごとに、「新奇性追求」から「自己超越」までの7つの性格得点が記録されています。
このモデリングでは性格「持続」の得点を目的変数に用いて、血液型との関係を調べます。

3.データの外観・統計量(テキストの表2.1、表2.2に対応)
まず、血液型ごとのデータ数を確認します。
### 血液型ごとの人数のカウント
count_data = data['血液型'].value_counts().to_frame()
count_data['比率'] = count_data['count'] / count_data['count'].sum()
display(count_data.round(2))【実行結果】
血液型ごとのデータ数の比率は、ほぼ4:3:2:1です。

要約統計量を確認しましょう。テキストの表2.1を代替します。
### 記述統計量の表示 ★表2.1に対応
data.iloc[:, 2:].describe().T.round(2)【実行結果】
性格ごとの質問数は、新奇性追求40、損害回避35、報酬依存32、持続8、自己志向44、協調42、自己超越33です。
「持続」は最小得点0から最大得点8まで幅広い区間があります。
ちなみに「報酬依存」の最大値22は質問数32からかなり乖離しているようです。
また「自己超越」には欠損値がありますが、「持続」の分析に影響しないので、削除や加工をしないで、そのまま置いておきます。

血液型ごとの性格別得点を確認しましょう。テキストの表2.2を代替します。
### 血液型別の性格得点 ★表2.2に対応
data.groupby(['血液型'])[data.columns[2:]].agg(['mean', 'std']).T.round(2)【実行結果】
みなさんが想像する血液型と性格の関係と比べて、この数値はいかがでしょう?

性格「持続」の血液型別ヒストグラムを確認しましょう。
### 性格「持続」の血液型別ヒストグラムの描画
sns.histplot(data=data, x='持続', hue='血液型', bins=9, stat='density',
common_norm=True, element='step')
plt.title('「持続」の血液型別得点のヒストグラム')
plt.xlabel('得点')
plt.ylabel('確率密度');【実行結果】
血液型ごとに差があるのか無いのか、よく分かりません・・・。


モデル構築
モデルの数式表現
目指したいPyMCのモデルの雰囲気を混ぜた「なんちゃって数式」表記です。
テキストの数式とほぼ同じです(テキストの詳細記載に感謝です!)。
$$
\begin{align*}
\mu_j &\sim \text{Uniform}(0, 100) \\
\sigma_e &\sim \text{Uniform}(0, 100) \\
likelihood &\sim \text{Normal}(\text{mu}=\mu_j,\ \text{sigma}=\sigma_e) \\
\end{align*}
$$
1.血液型インデックスの作成
blood_idxはカテゴリ変数「血液型」の要素$${\{A,\ B,\ O,\ AB\}}$$を次の整数値に置き換えたものです。
0:A型、1:B型、2:O型、3:AB型
PyMCのConstantDataに定義して利用します。
### 血液型のインデックスの作成
# 血液型を0:A, 1:B, 2:O, 3:ABの順番に変換して、blood_idxに格納
# PyMCのConstantDataで利用
# 血液型をidxに仮格納
blood_idx = data['血液型'].values
# A:0、B:1、O:2、AB:3に変換
blood_idx = np.where(blood_idx=='A', 0,
np.where(blood_idx=='B', 1,
np.where(blood_idx=='O', 2,
np.where(blood_idx=='AB', 3, 9))))
blood_idx = blood_idx.astype(int)
# idxの表示
blood_idx【実行結果】

2.モデルの定義
coord、data、パラメータの事前分布、尤度、後から利用する計算値をそれぞれ指定します。
### モデルの定義
with pm.Model() as model:
## データ関連定義
# coordの定義
model.add_coord('data', values=data.index, mutable=True)
model.add_coord('blood', values=['A', 'B', 'O', 'AB'], mutable=True)
# dataの定義
y = pm.ConstantData('y', value=data['持続'], dims='data')
bloodIdx = pm.ConstantData('bloodIdx', value=blood_idx, dims='data')
## パラメータの事前分布
mu = pm.Uniform('mu', lower=0, upper=100, dims='blood')
sigmaE = pm.Uniform('sigmaE', lower=0, upper=100)
## 尤度
likelihood = pm.Normal('likelihood', mu=mu[bloodIdx], sigma=sigmaE,
observed=y, dims='data')
## 計算値
# 全平均 muAll
muAll = pm.Deterministic('muAll', pt.mean(mu))
# 要因の効果の標準偏差 sigmaA
sigmaA = pm.Deterministic('sigmaA', pt.std(mu))
# 分散説明率 eta2
eta2 = pm.Deterministic('eta2',
pt.sqr(sigmaA) / (pt.sqr(sigmaA) + pt.sqr(sigmaE)))【モデル注釈】
coordの定義
座標に名前を付けたり、その座標が取りうる値を設定できます。
今回は次の2つを設定しました。
・行の座標:名前「data」、値「行インデックス」
・血液型の座標:名前「blood」、値「A、B、O、AB」dataの定義
目的変数$${y}$$と血液型インデックス$${bloodIdx}$$を設定しました。パラメータの事前分布
muの次元は血液型であり、coordで指定した「blood」を指定します。尤度
muはdataで定義した血液型インデックスを利用して「mu[bloodIdx]」とします。計算値
テキストの表2.4で用いる全平均$${\mu}$$:muAll、要因の効果の標準偏差$${\sigma_a}$$:sigmaA、分散説明率$${\eta^2}$$:eta2を計算します。分散説明率$${\eta^2=\cfrac{\sigma_a^2}{\sigma_a^2 + \sigma_e^2}}$$です。
pytensor.tensorの関数を利用します。pm.mathよりもコードを短く書けます。
pt.mean()は平均の計算。pt.mean($${\mu}$$)は$${\mu}$$の全平均です。
pt.std()は標準偏差の計算。pt.std($${\mu}$$)は$${\mu}$$の標準偏差です。
pt.sqr()はニ乗の計算。pt.sqr($${\sigma_a}$$)=$${\sigma_a^2}$$です。
3.モデルの外観の確認
### モデルの表示
model【実行結果】

### モデルの可視化
pm.model_to_graphviz(model)【実行結果】


MCMCの実行
1.事後分布からのサンプリング
乱数生成数(draws, tune, chains)はテキストどおりです。
nuts_sampler='numpyro'とすることで、numpyroをNUTSサンプラーに利用できます。
処理時間は20秒でした。
### 事後分布からのサンプリング ※NUTSサンプラーにnumpyroを使用 20秒
with model:
idata = pm.sample(draws=20000, tune=1000, chains=5, target_accept=0.95,
nuts_sampler='numpyro', random_seed=1234)【実行結果】省略
2.サンプリングデータの確認
$${\hat{R}}$$、事後分布の要約統計量、トレースプロットを確認します。
事後分布の収束確認はテキストにならって$${\hat{R} \leq 1.1}$$としています。
### r_hat>1.1の確認
rhat_idata = az.rhat(idata)
(rhat_idata>1.1).sum()【実行結果】
$${\hat{R} > 1.1}$$のパラメータは「0」件です。
全てのパラメータが$${\hat{R} \leq 1.1}$$であることを確認できました。
事後分布の要約統計量です。
テキストの表2.4、表2.5の代替です。
### 推論データの要約統計情報の表示 ★表2.4、2.5に対応
var_names = ['muAll', 'sigmaA', 'eta2', 'mu', 'sigmaE']
pm.summary(idata, var_names=var_names, hdi_prob=0.95)【実行結果】
統計量は上から、$${\mu}$$、$${\sigma_a}$$、$${\eta^2}$$、$${\mu_A}$$、$${\mu_B}$$、$${\mu_O}$$、$${\mu_{AB}}$$、$${\sigma_e}$$です。
EAP(mean)、post.sd(sd)、2.5%(hdi_2.5%)、97.5%(hdi_97.5%)の値はテキストとほぼ同じです!(やったー!)
なお、テキストはの95%区間は信用区間ですが、summary関数の95%区間はHDI区間です。

【分析】
分散説明率$${\eta^2}$$は、血液型が性格「持続」の得点をどの程度説明しているかを表す指標です。
$${\eta^2}$$のEAP=点推定値は$${0.7\%}$$とかなり小さな値です。
血液型が持続得点の$${\boldsymbol{0.7\%}}$$しか説明していないということは、血液型で性格「持続」が分かるとは言い難い、ということのようです。
トレースプロットです。
### トレースプロットの描画
pm.plot_trace(idata, var_names=var_names, compact=False)
plt.tight_layout();【実行結果】
きれいなトレースプロットです。収束している感触を満喫しました。


分析
1.平均値差に関連する推定量の計算
テキストの表2.6、表2.7の推定量を計算してpandasのデータフレームに格納します。
### 「持続」の平均値差について、A型とO型、A型とAB型の比較数値を計算
# 事後分布からサンプリングした 100000個のデータを用いて推定する
## numpy配列のEAP, std, 2.5%/97.5%quantileを返す関数の定義
def calc_estimator(dat):
return (np.mean(dat), np.std(dat), np.quantile(dat, 0.025),
np.quantile(dat, 0.975))
## 推論データの変数の取り出し
# 尤度の血液型別平均データ shape=(100000, 4)
# A型:mu[:, 0], B型:mu[:, 1], O型:mu[:, 2], AB型:mu[:, 3]
mu = idata.posterior.mu.data.reshape(100000, 4)
# 尤度の標準偏差データ shape=(100000, )
sigma_E = idata.posterior.sigmaE.data.flatten()
## A型とO型の平均値差に関する推定値の計算
# 平均値差μA-μO, 効果量δ, 非重複度U3, 優越率πd
mu_diff_A_O = mu[:, 0] - mu[:, 2]
delta_A_O = mu_diff_A_O / sigma_E
U3_A_O = stats.norm.cdf(mu[:, 0], mu[:, 2], sigma_E)
pi_A_O = stats.norm.cdf(delta_A_O / np.sqrt(2), 0, 1)
# 推定値のデータフレーム化
df_A_O = pd.DataFrame({'μA-μO': calc_estimator(mu_diff_A_O),
'効果量_δ': calc_estimator(delta_A_O),
'非重複度_U3': calc_estimator(U3_A_O),
'優越率_πd': calc_estimator(pi_A_O)},
index=['EAP', 'post.sd', '2.5%', '97.5%']).T
## A型とAB型の平均値差に関する推定値の計算
# 平均値差μA-μAB, 効果量δ, 非重複度U3, 優越率πd
mu_diff_A_AB = mu[:, 0] - mu[:, 3]
delta_A_AB = mu_diff_A_AB / sigma_E
U3_A_AB = stats.norm.cdf(mu[:, 0], mu[:, 3], sigma_E)
pi_A_AB = stats.norm.cdf(delta_A_AB / np.sqrt(2), 0, 1)
# 推定値のデータフレーム化
df_A_AB = pd.DataFrame({'μA-μAB': calc_estimator(mu_diff_A_AB),
'効果量_δ': calc_estimator(delta_A_AB),
'非重複度_U3': calc_estimator(U3_A_AB),
'優越率_πd': calc_estimator(pi_A_AB)},
index=['EAP', 'post.sd', '2.5%', '97.5%']).T【実行結果】なし
【計算式】
統計量に関するテキストの説明とRコードの計算式を引用させていただき、A型とO型の差を例にして書きます。
■ 効果量:$${\delta=\cfrac{\mu_A - \mu_O}{\sigma_e}}$$
効果量$${\delta}$$の値を10倍すると偏差値のように平均値差を解釈できるそうです。
■ 非重複度:$${U_3=F(x=\mu_A \mid \text{mu}=\mu_O,\ \text{sigma}=\sigma_e)}$$
$${F}$$は正規分布の累積分布関数です。Norm.CDFです。
A型の平均値$${\mu_A}$$がO型の分布の何%点に位置するかを表すそうです。
50%点に位置する場合、A型とO型は完全に重複していると解釈されます。
■ 優越率:$${\pi_d=F \left(x=\delta / \sqrt{2} \mid \text{mu}=0,\ \text{sigma}=1 \right)}$$
標準正規分布の累積分布関数です。
A型からランダムに選ばれた測定値が、O型からランダムに選ばれた測定値より大きい確率を表すそうです。
では、計算した推定値を表示します。
### 平均値差に関する推定値の表示 ★表2.6、2.7に対応 ★AとABの値がテキストと異なる
print('【A型とO型の平均値差に関する推定値】')
display(df_A_O.round(3))
print('\n【A型とAB型の平均値差に関する推定値】')
display(df_A_AB.round(3))【実行結果】
A型とO型の推定値はテキストとほぼ同じ値です!(やったー!)
しかし、A型とAB型の推定値はテキストと大きく異なります(がーん)。
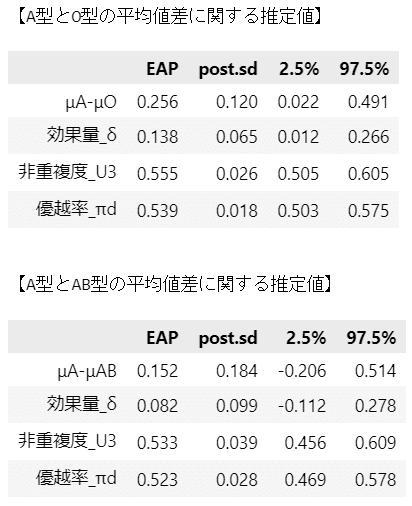
【推定値の評価】
事後分布の要約統計量の値を用いると$${\mu_A}$$の平均$${4.588-\mu_{AB}}$$の平均$${4.436=0.152}$$となり、この表の$${\mu_A - \mu_{AB}}$$のEAP$${0.152}$$と一致するのです。
なので、この推定値が適切であると仮定して、分析を進めます!
【分析】
テキストの分析の仕方を利用させていただき、書きます。
概して、血液型による性格「持続」の差があるとしても些細な差であり、実質的に意味のある差とは考えにくい、ということのようです。
■ A型とO型の平均値差
効果量$${\delta}$$(偏差値換算)はA型のほうがO型よりも$${1.38}$$ほど「持続」の得点が高い。
→偏差値的に差はほとんど無いと言えるかも?
非重複度$${U_3}$$はA型の「持続」の得点の平均値はO型の$${55.5\%}$$あたりに位置する。A型の方が$${5.5\%}$$分だけ「持続」の高い人が多いことを示す。
→$${5.5\%}$$という違いは小さいと言えるかも?
優越率$${\pi_d}$$はランダムにA型の人とO型の人を選んで比較すると$${53.9\%}$$の確率でA型の人の「持続」の得点が高いことを示す。
→$${53.9\%}$$程度の確率の高さは差が小さいと言えるのかも?
■ A型とAB型の平均値差
効果量$${\delta}$$(偏差値換算)はA型のほうがAB型よりも$${0.82}$$ほど「持続」の得点が高い。
→偏差値的に差はほとんど無いと言えるかも?
非重複度$${U_3}$$はA型の「持続」の得点の平均値はAB型の$${53.3\%}$$あたりに位置する。A型の方が$${3.3\%}$$分だけ「持続」の高い人が多いことを示す。
→$${3.3\%}$$という違いは小さいと言えるかも?
優越率$${\pi_d}$$はランダムにA型の人とAB型の人を選んで比較すると$${52.3\%}$$の確率でA型の人の「持続」の得点が高いことを示す。
→$${52.3\%}$$程度の確率の高さは差が小さいと言えるのかも?

最後にこれらの推定値をヒストグラムに描画しましょう。
### A型とO型の平均値差に関する推定値のヒストグラムの描画
plt.figure(figsize=(8, 4))
# ヒストグラムの描画(平均値差)
plt.hist(mu_diff_A_O, bins=50, density=True, histtype='step',
label=r'平均値差 $\mu_A-\mu_O$')
# ヒストグラムの描画(効果量)
plt.hist(delta_A_O, bins=50, density=True, histtype='step',
label=r'効果量 $\delta$')
# ヒストグラムの描画(非重複度)
plt.hist(U3_A_O, bins=50, density=True, histtype='step',
label=r'非重複度 $U_3$')
# ヒストグラムの描画(優越率)
plt.hist(pi_A_O, bins=50, density=True, histtype='step',
label=r'優越率 $\pi_d$')
# 修飾
plt.xticks(np.arange(-0.3, 0.9, 0.1))
plt.title('A型とO型の平均値差に関する推定値のヒストグラム')
plt.xlabel('推定値')
plt.ylabel('確率密度')
plt.legend(loc='upper left')
plt.show()【実行結果】

### A型とAB型の平均値差に関する推定値のヒストグラムの描画
plt.figure(figsize=(8, 4))
# ヒストグラムの描画(平均値差)
plt.hist(mu_diff_A_AB, bins=50, density=True, histtype='step',
label=r'平均値差 $\mu_A-\mu_{AB}$')
# ヒストグラムの描画(効果量)
plt.hist(delta_A_AB, bins=50, density=True, histtype='step',
label=r'効果量 $\delta$')
# ヒストグラムの描画(非重複度)
plt.hist(U3_A_AB, bins=50, density=True, histtype='step',
label=r'非重複度 $U^3$')
# ヒストグラムの描画(優越率)
plt.hist(pi_A_AB, bins=50, density=True, histtype='step',
label=r'優越率 $\pi_d$')
# 修飾
plt.xticks(np.arange(-0.6, 1.1, 0.1))
plt.title('A型とAB型の平均値差に関する推定値のヒストグラム')
plt.xlabel('推定値')
plt.ylabel('確率密度')
plt.legend(loc='upper left')
plt.show()【実行結果】

2.推論データ(idata)の保存
推論データを再利用する場合に備えてファイルに保存しましょう。
idataをpickleで保存します。
### idataの保存 pickle
file = r'idata_ch02.pkl'
with open(file, 'wb') as f:
pickle.dump(idata, f)読み込みコードは次のとおりです。
### idataの読み込み pickle
file = r'idata_ch02.pkl'
with open(file, 'rb') as f:
idata_load = pickle.load(f)3.おまけ
A型とB型の平均値差の推定値も計算しましょう。
### A型とB型の平均値差に関する推定値の計算
# 平均値差μA-μB, 効果量δ, 非重複度U3, 優越率πd
mu_diff_A_B = mu[:, 0] - mu[:, 1]
delta_A_B = mu_diff_A_B / sigma_E
U3_A_B = stats.norm.cdf(mu[:, 0], mu[:, 1], sigma_E)
pi_A_B = stats.norm.cdf(delta_A_B / np.sqrt(2), 0, 1)
# 推定値のデータフレーム化
df_A_B = pd.DataFrame({'μA-μB': calc_estimator(mu_diff_A_B),
'効果量_δ': calc_estimator(delta_A_B),
'非重複度_U3': calc_estimator(U3_A_B),
'優越率_πd': calc_estimator(pi_A_B)},
index=['EAP', 'post.sd', '2.5%', '97.5%']).T
print('\n【A型とB型の平均値差に関する推定値】')
display(df_A_B.round(3))【実行結果】
実は、A型とB型の差がもっとも大きく、効果量・非重複度・優越率の全てが他の血液型よりも高い値になっています。
高いと入っても、おそらく、O型・AB型の分析と同様に、実質的に意味のある差では無かろう、という結論になりそうです。
なお、テキストはB型を取り上げていませんが、RスクリプトはB型を取り上げています(RスクリプトではA型とO型、A型とB型を比較)。

恒例のヒストグラムを描画してみます。
### A型とB型の平均値差に関する推定値のヒストグラムの描画
plt.figure(figsize=(8, 4))
# ヒストグラムの描画(平均値差)
plt.hist(mu_diff_A_B, bins=50, density=True, histtype='step',
label=r'平均値差 $\mu_A-\mu_B$')
# ヒストグラムの描画(効果量)
plt.hist(delta_A_B, bins=50, density=True, histtype='step',
label=r'効果量 $\delta$')
# ヒストグラムの描画(非重複度)
plt.hist(U3_A_B, bins=50, density=True, histtype='step',
label=r'非重複度 $U^3$')
# ヒストグラムの描画(優越率)
plt.hist(pi_A_B, bins=50, density=True, histtype='step',
label=r'優越率 $\pi_d$')
# 修飾
plt.xticks(np.arange(-0.3, 1.0, 0.1))
plt.title('A型とB型の平均値差に関する推定値のヒストグラム')
plt.xlabel('推定値')
plt.ylabel('確率密度')
plt.legend(loc='upper left')
plt.show()【実行結果】

以上で第2章は終了です。

おわりに
血液型と性格の関係、優しいモデル
「血液型による性格診断の信ぴょう性」
長きにわたる論争は決着できるのでしょうか?
個人的には、人々が血液型性格診断に触れることによって、あたかも洗脳の如く、性格診断内容に合った振る舞いをするようになる、こんな気がします。
なので、血液型性格診断が流行している間は、血液型と性格に関係があるように感じ続けるのだと思います。

ところで、今回は初学者にとって優しいモデルでした。
尤度に正規分布、パラメータ$${\mu,\ \sigma}$$の事前分布に一様分布を用いたシンプルな構成です。
$${\mu}$$は「血液型」の要素ごとに設定しました。
ベイズモデリングの基本を学べる「やさしい」テーマです。
また、血液型間の差について、「差の検定」などの伝統的な統計的仮説検定との比較で、事後分布からのサンプルデータに基づくベイズ流の分析は斬新に感じます。
シンプルで力強いこの分析をしっかり身につけたいと思います。
シリーズの記事
次の記事
前の記事
目次
ブログの紹介
note で4つのシリーズ記事を書いています。
ぜひ覗いていってくださいね!
1.のんびり統計
統計検定2級の問題集を手がかりにして、確率・統計をざっくり掘り下げるブログです。
雑談感覚で大丈夫です。ぜひ覗いていってくださいね。
統計検定2級公式問題集CBT対応版に対応しています。
2.RとStanではじめる心理学のための時系列分析入門 を PythonとPyMC Ver.5 で
書籍「RとStanではじめる心理学のための時系列分析入門」の時系列分析トピックを PythonとPyMC Ver.5で取り組みます。
豊富なテーマ(お題)を実践することによって、PythonとPyMCの基礎体力づくりにつながる、そう信じています。
日々、Web検索に勤しみ、時系列モデルの理解、Pythonパッケージの把握、R・Stanコードの翻訳に励んでいます!
このシリーズがPython時系列分析の入門者の参考になれば幸いです🍀
3.Python機械学習プログラミング実践記
書籍「Python機械学習プログラミング PyTorch & scikit-learn編」を学んだときのさまざまな思いを記事にしました。
この書籍は、scikit-learn と PyTorch の教科書です。
よかったらぜひ、お試しくださいませ。
4.データサイエンスっぽいことを綴る
統計、データ分析、AI、機械学習、Python のコラムを不定期に綴っています。
「統計」「Python」「数学とPython」「R」のシリーズが生まれています。
ベイズ書籍の実践記録も掲載中です。
最後までお読みいただきまして、ありがとうございました。
