
slspecファイルの作り方
概要
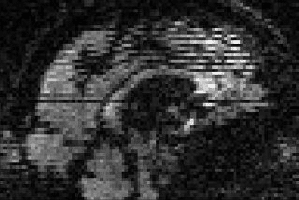
MRIの撮影法に「interleaved」という方法がある。1枚おきにタイミングを変えて撮影する方法で、拡散強調像は主にこの方法で取得される。この方法で撮影中に体動が激しいと、一つのvolume内でも動きが出てスライスの繋がりがおかしくなってしまい、矢状断、冠状断で見たときにこんなジグザグパターンの画像が出来上がる。FSL(FMRIB Software Library)のツールを用いてこれを補正する方法についてまとめた。
追記(2023年10月5日)
以下の内容をほぼそのままやってくれるプログラムをpython(jupyter-notebook)で作ってみました。pandasのライブラリが必要です。結果だけ欲しい、かつgithubやjupyter-notebookに抵抗のない方は以下のリンクからお試しください。日本語READMEもあります。
https://github.com/Kikubernetes/slspec
slspecファイルとは
FSLのeddyというツールはその名の通りeddy currentを補正するのに使われているが、同時に被験者の体動についても補正してくれる。通常はボリューム間の体動補正のみだが、--mporderのオプションをつけるとボリューム内体動も補正してくれる。(現状、FSLのUserguideにも書かれているとおりeddy_cuda(eddyのGPUバージョン)でしか使えない)
これを使うためには--slspec=slspec.txtという形でslspecファイルを与える必要がある。slspecファイルとは、スライス番号(volume内の位置)を撮影した順番に並べたものである。マルチバンドを使っている場合は同時に撮影されたものを横にならべて書く。スライスの位置番号はpython等のインデックスと同じで0番目から数える。例えば以下の場合は全部で15枚の画像があり、マルチバンドは3、一番最初に1枚目、6枚目、11枚目が撮影されている。

これは現時点では自動的に作ってはくれないので自分で作る必要がある。なかなか面倒だが撮影方法を変えない限りは使い回せる。
作り方はhttps://fsl.fmrib.ox.ac.uk/fsl/fslwiki/eddy/UsersGuide#A--slspec:~:text=s2v_interp%20is%20set.-,%2D%2Dslspec,-Specifies%20a%20textに記載されているとおりなのだが、いまいち分かりづらい&英語なのでエクセルとターミナル(もしくはテキストエディタ)を使って比較的簡単に作る方法をまとめてみた。
スライスタイミングの取得
ここではz軸方向に74枚のスライスがある、interleaved、マルチバンド2の画像を使った。
まずはdcm2niixを使ってDICOM画像をniftiファイルに変換する。
そうすると自動的にjsonファイルができる。この中で"SliceTiming": という項目を探す。
"SliceTiming": [
0,
4.72432,
0.248649,
4.97297,
0.497297,
5.22162,
0.745946,
5.47027,
0.994595,
5.71892,
1.24324,
5.96757,
1.49189,
6.21622,
1.74054,
6.46486,
・・・以下略・・・
こんな感じで書かれている。
数字の部分だけをコピーしてテキストエディタに貼り付ける。(temp.txtとする)

そうするとタブがたくさん入ってしまうので、コマンドを使って消す。(エディタの「検索と置換」でもできるはず)
cat temp.txt | sed 's/\t\t//g' >> order.csv エクセルで加工
できあがったcsvファイルをエクセルやlibreofficeで開くとこんな感じになる。

この列の左側に、スライス位置を表す番号として0から始まる連番の列を挿入する。(連番を挿入するには0を入れてリターンしてから右下の十字マークをひっぱる)

ここでB列を昇順にソート→データ全体のソートとすると次のようになる。

このときA列は0から始まるスライス位置、B列が撮影時間を表している。たとえば上の表なら、0枚目(=ほんとは1枚目)と37枚目(=38枚目)が同時に時刻0に撮影され、次に2枚目と39枚目が時刻0.248649に撮影され・・・ということである。
ここで復習だがslspecファイルには撮影したスライスを順番に並べる(同時のものを横に書く)必要がある。スライスの順番は0番目から数える。ということで一番最初は

となる。同じようにならべていくと、

これもエクセルの機能で0から始まって2ごとの連番、37から始まって2ごとの連番をそれぞれ作れる。(最初の2個を入れてから上記と同様にする)
この画像はスライス方向の枚数が74枚なので、73枚目(=74枚目)で切り替わるため、(36、73)の次は(1、38)のペアになる。

同様に1,3,5・・・の連番、38、40、42・・・の連番を作る。
最終形がこちら。

これをそのままテキストエディタに貼り付けてslspec.txtという名前にする。タブ区切りのテキストファイルが出来上がる。明示されていないのだがFSLのサイトにある図だと区切りがスペースっぽいので一応「検索と置換」を使って区切りをスペースにしておいたほうがいいかもしれない。
eddyで使うには
使い方については上記のFSLのサイトにもあるが、コマンドの一例を貼っておく。
eddy_cuda10.2 --imain=eddy_in.nii --mask=eddy_mask.nii --acqp=eddy_config.txt --index=eddy_indices.txt --bvecs=bvecs --bvals=bvals --topup=my_topup_file --slm=linear --repol --out=dwi_post_eddy --verbose --mporder=6 --slspec=slspec.txt --s2v_niter=5 --s2v_lambda=1 --s2v_interp=trilinear--mporder=6 --slspec=slspec.txt --s2v_niter=5 --s2v_lambda=1 --s2v_interp=trilinearの部分がボリューム内の動き補正に関連するコマンド。個々のオプションの詳細については上記サイトを参照。
MRtrix3のdwifslpreprocを使っている場合は-nocleanupのオプションをつけて実行するとdwifslpreproc-tmp-XXXXXXというディレクトリの中にeddyに必要なファイルとeddyコマンドの記載されたcommand.txtを吐き出してくれる。このeddyコマンドの後に--mporder=6以下の部分を追加してこのディレクトリ内で実行すればお手軽に補正前後を比較できる。
左が動き補正前、右が補正後。完璧とはいかないが、かなりジグザグパターンが消えている。


おまけ
最近までGEのMRIではdcm2niixでスライスタイミングを取得できなかったが、最新版version v1.0.20230411では取得できるようになりました。
