
書記のBio/Chem-Info日誌#17 AutoDock Vinaで分子ドッキングをする
AutoDock Vinaで分子ドッキングをする方法にはいろいろあるが,細かいことを気にしないならUCSF Chimeraと組み合わせるのが楽でいいと思う。
前回の続き:
準備
AutoDock Vinaは以下よりダウンロードできる。
https://vina.scripps.edu/downloads/
ダウンロードすると,「autodock_vina_1_1_2_mac_catalina_64 bit」というファイルが作られるので,適当なところに置いて,binフォルダにあるvinaファイルを実行すれば,準備完了である。
構造の前処理
まずはTools→Surface/Binding Analysis→Dock Prepより,前処理を行う。ここでは,溶媒分子やalternative coordinatesの削除などの処理,水素分子の付加などが行われる。とりあえずデフォルトのままOKを押す。

ここでもデフォルトでOK。

さて,ドッキングで鋳型とするのはレセプター構造である。前回作った構造から,レセプターとリガンドを分離するには,Favorites→ Command Lineでコマンドラインを表示して,下に表示されるCommand:に「split ligand」と入力しエンター (リターン) キーを押す。

分離できると,以下のようなメッセージが出る。

最後に,File→Open...からドッキングをしたい分子を選択する。
今回は,β2受容体に対して,アドレナリン(内因性リガンド,CID:5816),サルブタモール(β2アゴニスト,CID:2083),プロプラノロール(β2アンタゴニスト,CID_4946)をドッキングしてみる。また,3NS6構造に元々ついていた分子名がよく分からないリガンド(CID:49837874)を基準とする。
ドッキング本番,基準の分子から
まずは基準となるCID:49837874分子をドッキングする。というのも,ドッキングで得られた分子構造が,X線構造と比較してズレが少ないのが望ましい。
Tools→Surface/Binding Analysis→AutoDock Vinaから以下のパネルを表示する。設定事項は以下の通り:
Output file:ファイルの保存場所を指定し,ファイル名をつける
Recdptor:split ligandすると作られるレセプター構造を選択する(多分一番上の構造)
Ligand:ドッキングしたい構造を選択する,CID番号で表示される
Receptor search volume options:毎回必要な設定。リガンドと周辺残基が収まるようにグリッドを調整する。
Executable location:Localを選択し,先ほどダウンロードしたvivaファイルを指定。
他はそのままとした。

こんな感じで収まっていれば問題ない(多分)。

Applyを押すと計算をするようになる。今回は1分ほどかかった。
結果がこのように表示される。左のScoreが重要で,今回は「-11.1」が基準となる。

一番上の構造がこちら。元のX線構造と同じ構造をとっていることがわかり,ドッキングの妥当性が一応は示せた。
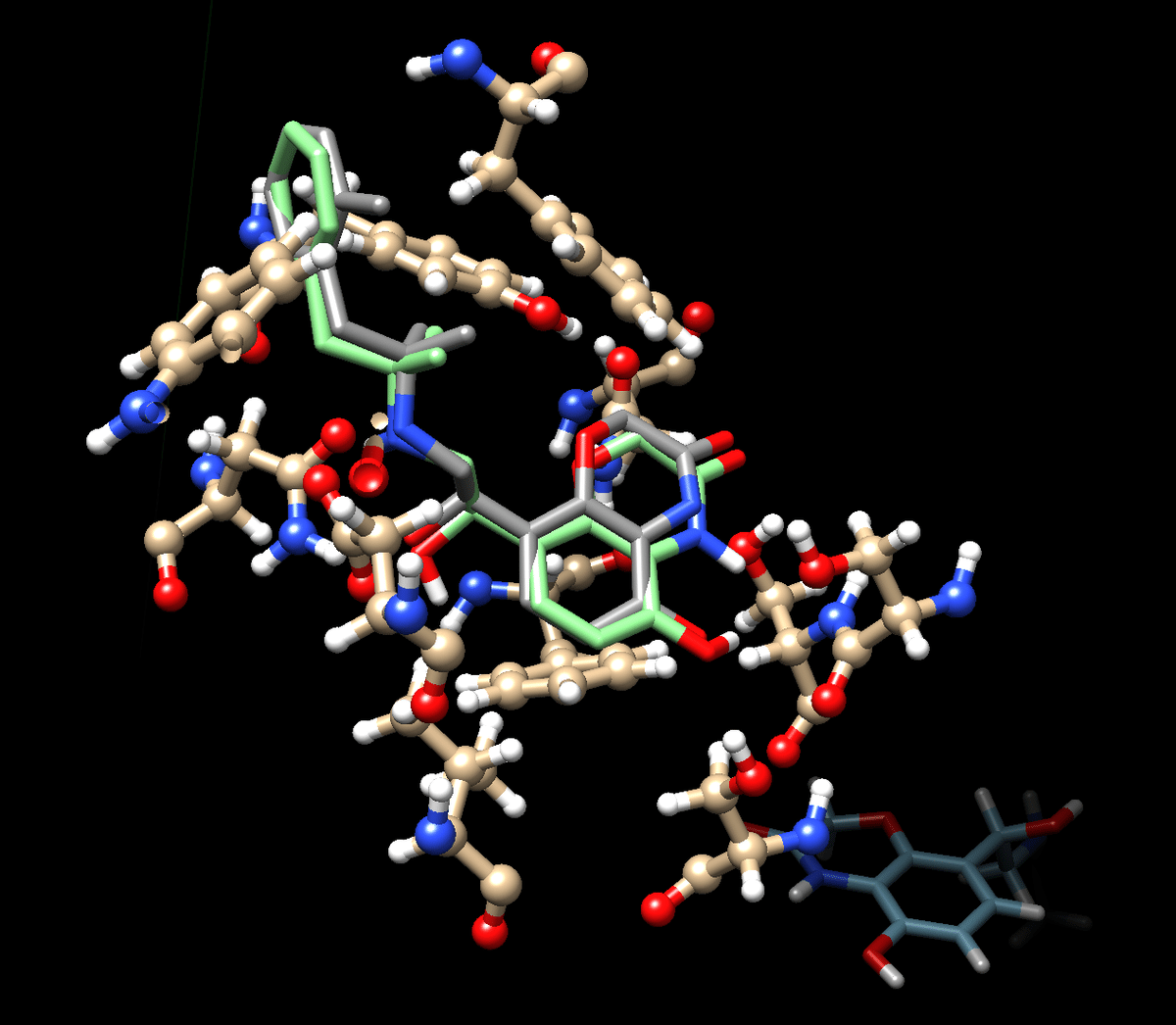
他の分子の結果
アドレナリン(内因性リガンド,CID:5816)
元の構造の一部分を埋めている形をとっており,その分結合する場所が少なくなり,相互作用が弱まることが予想できる。


サルブタモール(β2アゴニスト,CID:2083)
アドレナリンの誘導体であり,多少位置のズレのある結合をする,Scoreより相互作用はより強いものと予測できる。


プロプラノロール(β2アンタゴニスト,CID_4946)
CID:49837874と構造が似ており,同じような構造を示すかと思ったが,実際には大きくズレていた。あまり適切なドッキングではないかもしれない。


参考:
本記事のもくじはこちら:

