
書記が数学やるだけ#850 実際のネットワークの比較
ネットワーク科学の具体例としてタンパク質間相互作用(PPI)を扱ってみる。
問題

説明
タンパク質間相互作用(PPI)について,近年になって薬剤は生体のネットワークを変調させるものとして捉えるネットワーク薬理学が注目されている。

PPIのネットワークを得る手順は以下記事を参照;
解答
今回は風邪(cold)に関連するタンパク質について,GeneCardsというデータベースから取得する。

今回はスコア上位1000のタンパク質からネットワークを作る。
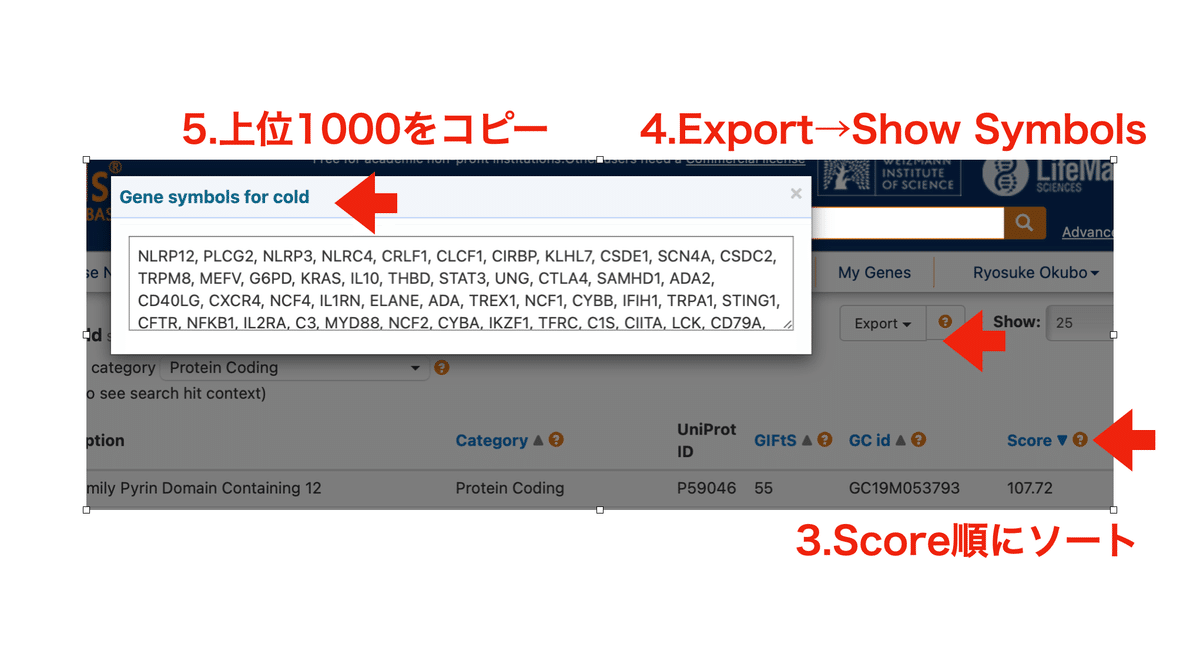
これをstringに入力した結果を以下に示す。このPPIネットワークについて,tsvでダウンロードして以後の分析に用いる。

まずはSTRINGからダウンロードしたTSVファイルを開き,「#node1」と「node2」のデータからエッジを追加していく。可視化は適当で良い。
import pandas as pd
import networkx as nx
import matplotlib.pyplot as plt
# STRINGからダウンロードしたTSVファイルのパス(適宜変える)
file_path = '/content/drive/MyDrive/string_interactions_short.tsv'
# データをpandasで読み込む
df = pd.read_csv(file_path, sep='\t')
# ネットワークを作成
G = nx.Graph()
# データフレームからエッジを追加(タンパク質1、タンパク質2を使用)
for index, row in df.iterrows():
protein1 = row['#node1'] # カラム名は実際のデータに合わせて変更
protein2 = row['node2'] # カラム名は実際のデータに合わせて変更
G.add_edge(protein1, protein2)
# ノードの配置を設定(ここではspringレイアウトを使用)
pos = nx.spring_layout(G)
# ネットワークの描画
plt.figure(figsize=(10, 8))
nx.draw_networkx_nodes(G, pos, node_size=500, node_color='skyblue')
nx.draw_networkx_edges(G, pos)
nx.draw_networkx_labels(G, pos, font_size=12, font_family='sans-serif')
plt.title("PPI Network Visualization from STRING Data (Without Scores)")
plt.show()
次数分布の可視化に必要な情報を計算しておく。
# 次数の合計
total_degree = sum(dict(G.degree()).values())
# 平均次数
average_degree = total_degree / G.number_of_nodes()
print("平均次数:", average_degree)
#平均次数: 48.094358974358975
# 各ノードの次数を取得
degree_dict = dict(G.degree())
# 次数の最大値を求める
max_degree = max(degree_dict.values())
print("次数の最大値:", max_degree)
#次数の最大値: 332
# ノード数を取得
number_of_nodes = G.number_of_nodes()
print("ノード数:", number_of_nodes)
ノード数: 975次数分布を可視化する。この段階でハブが多いことがわかる。
degree_dist = [i/number_of_nodes for i in nx.degree_histogram(G)]
plt.rcParams['font.size'] = 20
plt.bar(range(max_degree+1), height = degree_dist)
plt.xlabel('$k$')
plt.ylabel('$p(k)$')
plt.ylim(0,0.03)
まずはランダムネットワークかどうかを判断する,平均次数λのポアソン分布と比較するとランダムネットワークでは説明できないハブが目立つ。
from scipy.stats import poisson
import numpy as np
# ポアソン分布のパラメータλを計算
average_degree = sum(dict(G.degree()).values()) / number_of_nodes
# ポアソン分布を計算
k_values = np.arange(len(degree_dist))
poisson_dist = poisson.pmf(k_values, average_degree)
# グラフを描画
plt.figure(figsize=(10, 6))
plt.rcParams['font.size'] = 20
# 次数分布のプロット
plt.loglog(k_values, degree_dist, marker='o', linestyle='None', label='Degree Distribution')
# ポアソン分布のプロット
plt.loglog(k_values, poisson_dist, marker='x', linestyle='-', label='Poisson Distribution')
plt.xlabel('$k$')
plt.ylabel('$p(k)$')
plt.ylim(1e-4, 1) # Y軸の範囲を調整
plt.grid(True, which="both", ls="--")
plt.legend()
plt.show()
次にスケールフリーネットワークかどうか,γを計算して確かめる。近似の粗さもあるが,γが2~3にないことから,スケールフリーネットワークだけでは説明できない要素もあることが言える。
# 次数を取得
degrees = [d for n, d in G.degree()]
# ビンの境界を対数スケールで設定
bins = np.logspace(np.log10(min(degrees)), np.log10(max(degrees)), num=20)
# 各ビンに含まれるノードの数を計算
hist, bin_edges = np.histogram(degrees, bins=bins, density=True)
# ビンの中心を計算
bin_centers = (bin_edges[:-1] + bin_edges[1:]) / 2
# 対数スケールで線形回帰を行うために、データの対数を取る
log_bin_centers = np.log10(bin_centers)
log_hist = np.log10(hist)
# 有効なデータのみを使う(ゼロでないデータ)
valid_indices = ~np.isinf(log_hist) # 無限大を持たないインデックス
log_bin_centers = log_bin_centers[valid_indices]
log_hist = log_hist[valid_indices]
# 近似直線をフィッティング(1次の多項式)
slope, intercept = np.polyfit(log_bin_centers, log_hist, 1)
# フィッティング結果の表示
print(f"近似直線の傾き: {slope:.4f}")
# グラフを描画
plt.figure(figsize=(10, 6))
plt.rcParams['font.size'] = 20
# ドットプロットをプロット
plt.plot(bin_centers, hist, 'o', label='Data')
# 有効なデータの範囲でのみプロット
plt.plot(bin_centers[valid_indices], 10**(slope * log_bin_centers + intercept), '--', label=f'Fit: slope = {slope:.4f}')
plt.xscale('log')
plt.yscale('log')
plt.xlabel('$k$')
plt.ylabel('$p(k)$')
plt.ylim(1e-4, 1) # Y軸の範囲を調整
plt.grid(True, which="both", ls="--")
plt.legend()
plt.show()
最後に次数相関を計算,結果は正の値でありハブ同士の繋がりが示唆される。
# 次数相関を計算
assortativity_coefficient = nx.degree_assortativity_coefficient(G)
# 結果を表示
print("次数相関係数:", assortativity_coefficient)
#次数相関係数: 0.11596069421355934ハブ(次数200以上のノード)のネットワークを可視化してみる。
# ハブの閾値を設定
hub_threshold = 200
# ハブノードのみを抽出
hub_nodes = [node for node, degree in dict(G.degree()).items() if degree >= hub_threshold]
# ハブノードを含むサブグラフを作成
H = G.subgraph(hub_nodes)
# ノードのサイズに基づいてサイズを設定(スケールを調整)
node_size = [H.degree(v) * 300 for v in H]
# ハブノードのみのネットワークを可視化
plt.figure(figsize=(12, 12))
pos = nx.spring_layout(H, seed=42) # スプリングレイアウトでノードを配置
# ノードを描画 (色を黄色に統一)
nx.draw_networkx_nodes(H, pos, node_size=node_size, node_color='yellow', alpha=0.9)
# エッジを描画
nx.draw_networkx_edges(H, pos, alpha=0.5)
nx.draw_networkx_labels(H, pos, font_size=10, font_family='sans-serif')
plt.title('Hub Nodes Network Visualization')
plt.show()
本記事のもくじはこちら:
学習に必要な本を買います。一覧→ https://www.amazon.co.jp/hz/wishlist/ls/1XI8RCAQIKR94?ref_=wl_share
