
ChatGPTを使ってRの勉強をしながらグラフを描画する
以前、Rでグラフを描く記事を書きました。
Rを使ってグラフを描くと、エクセルと違ってログが残るので、どのデータを使ってどのようにして描いたかが一目でわかるという利点があります。
しかし、普段wetの実験が中心で、たまにしか使わないとRの関数を忘れてしまい、もう一度教科書を見たり、ググったりしたり、過去のノートを見返したりして、勉強し直すという面倒なことになります。
そこで役に立つのが、ChatGPTという便利なツールです。
https://chatgpt.com
ChatGPTの出力結果を100%信用するのは考えものですが、ChatGPTと会話しながらグラフを描いていけば、Rでグラフを描くのも簡単です。
では、やってみましょう。
例として使うデータは、以下の2つのcsvファイルです。
longday_control.csv
longday_low_temperature.csv
それぞれ、シロイヌナズナの野生株とtfl1変異体を22℃と16℃の長日条件下で栽培し、花茎の伸長(花成)が始まった時点でのロゼット葉の枚数をカウントしたものです。
* 元データは、Hanano et al. (2011) Plant Cell 23, 3172-3184 から抜粋
まず、ChatGPTのサイト(https://chatgpt.com)にアクセスし、ダイアログにcsvファイルをアップして、「次の2つのファイルを読み込んで、遺伝子型ごとにcontrolとlow_temperatureを比較するグラフを作成したい。そのためのRスクリプトを書いてもらえますか?」と入力してみます。

すると以下のコードが返ってきました。
さてどうでしょう?
# Load necessary libraries
library(ggplot2)
library(dplyr)
# File paths
control_file <- "/mnt/data/longday_control.csv"
low_temp_file <- "/mnt/data/longday_low_temperature.csv"
# Read the CSV files
control_data <- read.csv(control_file)
low_temp_data <- read.csv(low_temp_file)
# Add a column to identify the condition in each dataset
control_data$Condition <- 'Control'
low_temp_data$Condition <- 'Low Temperature'
# Combine the two datasets into one
combined_data <- rbind(control_data, low_temp_data)
# Ensure the 'Genotype' and 'Condition' columns are factors for proper plotting
combined_data$Genotype <- as.factor(combined_data$Genotype)
combined_data$Condition <- as.factor(combined_data$Condition)
# Plot: Comparing control and low temperature conditions by genotype
ggplot(combined_data, aes(x = Genotype, y = Value, fill = Condition)) +
geom_bar(stat = "identity", position = "dodge") +
labs(title = "Comparison of Control vs Low Temperature by Genotype",
x = "Genotype",
y = "Measured Value") +
theme_minimal() +
scale_fill_manual(values = c("Control" = "blue", "Low Temperature" = "red")) +
theme(axis.text.x = element_text(angle = 45, hjust = 1))
# If the "Value" column is named differently, adjust accordingly
# Replace `Value` with the correct column name (e.g., `Measurement`).まず、グラフ描画ツールggplot2、データフレーム操作ツールdplyrの2つのライブラリを使っていることがわかります。
私は、作業ディレクトリをデスクトップにして、csvファイルもデスクトップに置いているので、File pathsの指定はスキップします。
control_data <- read.csv(control_file)
low_temp_data <- read.csv(low_temp_file)の部分は、
control_data <- read.csv("longday_control.csv")
low_temp_data <- read.csv("longday_low_temperature.csv")に変更しましょう。
combined_data$Genotype <- as.factor(combined_data$Genotype)が動かないですね。
combined_dataの中身を見てやると、Genotypeはありません。
> control_data$Condition <- 'Control'
> low_temp_data$Condition <- 'Low Temperature'
> combined_data <- rbind(control_data, low_temp_data)
> combined_data
Col tfl1.1 tfl1.11 tfl1.13 tfl1.14 tfl1.17 Condition
1 6 5 5 5 5 7 Control
2 6 5 5 5 5 7 Control
3 7 5 5 5 5 7 Control
4 7 5 5 5 5 6 Control
5 7 5 5 5 5 7 Control
6 7 5 5 5 5 7 Control
7 7 5 5 6 5 7 Control
8 7 5 6 6 5 6 Control
9 8 5 6 6 6 NA Control
10 8 5 NA 6 6 NA Control
11 8 6 NA 6 6 NA Control
12 8 6 NA 6 6 NA Control
13 8 6 NA 6 6 NA Control
14 8 NA NA 6 6 NA Control
15 9 NA NA 6 6 NA Control
16 9 NA NA 7 NA NA Control
17 NA NA NA 7 NA NA Control
18 NA NA NA NA NA NA Control
19 18 6 13 6 7 8 Low Temperature
20 21 9 11 7 9 8 Low Temperature
21 22 9 11 8 8 8 Low Temperature
22 20 8 13 12 8 9 Low Temperature
23 17 8 13 12 7 8 Low Temperature
24 17 7 12 12 8 8 Low Temperature
25 16 8 14 13 8 7 Low Temperature
26 18 7 13 10 9 8 Low Temperature
27 28 8 12 12 8 8 Low Temperature
28 28 6 NA 12 8 8 Low Temperature
29 30 13 NA 9 7 7 Low Temperature
30 28 10 NA 11 8 6 Low Temperature
31 26 10 NA 12 8 8 Low Temperature
32 28 12 NA 12 7 7 Low Temperature
33 30 13 NA 12 8 NA Low Temperature
34 32 11 NA 13 9 NA Low Temperature
35 34 12 NA NA 9 NA Low Temperature
36 32 9 NA NA 9 NA Low TemperatureGenotypeを「Col tfl1.1 tfl1.11 tfl1.13 tfl1.14 tfl1.17」にしてもらうように、ChatGPTにお願いしてみましょう。

以下のコードが返ってきました。
# Load necessary libraries
library(ggplot2)
library(dplyr)
library(tidyr)
# File paths
control_file <- "/mnt/data/longday_control.csv"
low_temp_file <- "/mnt/data/longday_low_temperature.csv"
# Read the CSV files
control_data <- read.csv(control_file)
low_temp_data <- read.csv(low_temp_file)
# Add a column to identify the condition in each dataset
control_data$Condition <- 'Control'
low_temp_data$Condition <- 'Low Temperature'
# Combine the two datasets into one
combined_data <- rbind(control_data, low_temp_data)
# Convert from wide to long format
# Specifying that "Col", "tfl1.1", "tfl1.11", etc. are the genotypes
combined_data_long <- combined_data %>%
pivot_longer(cols = c("Col", "tfl1.1", "tfl1.11", "tfl1.13", "tfl1.14", "tfl1.17"),
names_to = "Genotype", values_to = "Value")
# Ensure the 'Genotype' and 'Condition' columns are factors for proper plotting
combined_data_long$Genotype <- as.factor(combined_data_long$Genotype)
combined_data_long$Condition <- as.factor(combined_data_long$Condition)
# Plot: Comparing control and low temperature conditions by genotype
ggplot(combined_data_long, aes(x = Genotype, y = Value, fill = Condition)) +
geom_bar(stat = "identity", position = "dodge") +
labs(title = "Comparison of Control vs Low Temperature by Genotype",
x = "Genotype",
y = "Measured Value") +
theme_minimal() +
scale_fill_manual(values = c("Control" = "blue", "Low Temperature" = "red")) +
theme(axis.text.x = element_text(angle = 45, hjust = 1))さらに、データ変形ツールのtidyrライブラリも読み込んでいます。
そして、以下のコマンドで、先ほどのデータからCondition, Genotype, Valueのデータを抜き出して、combined_data_longという縦長のファイルに変換してくれているのがわかります。
# Specifying that "Col", "tfl1.1", "tfl1.11", etc. are the genotypes
combined_data_long <- combined_data %>%
pivot_longer(cols = c("Col", "tfl1.1", "tfl1.11", "tfl1.13", "tfl1.14", "tfl1.17"),
names_to = "Genotype", values_to = "Value")combined_data_longの中身を見てみましょう。
> combined_data_long
# A tibble: 216 × 3
Condition Genotype Value
<chr> <chr> <int>
1 Control Col 6
2 Control tfl1.1 5
3 Control tfl1.11 5
4 Control tfl1.13 5
5 Control tfl1.14 5
6 Control tfl1.17 7
7 Control Col 6
8 Control tfl1.1 5
9 Control tfl1.11 5
10 Control tfl1.13 5
# ℹ 206 more rows
# ℹ Use `print(n = ...)` to see more rows動きそうですね。やってみましょう。
> library(ggplot2)
> library(dplyr)
> library(tidyr)
>
> control_data <- read.csv("longday_control.csv")
> low_temp_data <- read.csv("longday_low_temperature.csv")
>
> control_data$Condition <- 'Control'
> low_temp_data$Condition <- 'Low Temperature'
>
> combined_data <- rbind(control_data, low_temp_data)
>
> combined_data_long <- combined_data %>%
+ pivot_longer(cols = c("Col", "tfl1.1", "tfl1.11", "tfl1.13", "tfl1.14", "tfl1.17"),
+ names_to = "Genotype", values_to = "Value")
>
> combined_data_long$Genotype <- as.factor(combined_data_long$Genotype)
> combined_data_long$Condition <- as.factor(combined_data_long$Condition)
>
> ggplot(combined_data_long, aes(x = Genotype, y = Value, fill = Condition)) +
+ geom_bar(stat = "identity", position = "dodge") +
+ labs(title = "Comparison of Control vs Low Temperature by Genotype",
+ x = "Genotype",
+ y = "Measured Value") +
+ theme_minimal() +
+ scale_fill_manual(values = c("Control" = "blue", "Low Temperature" = "red")) +
+ theme(axis.text.x = element_text(angle = 45, hjust = 1))
Warning message:
Removed 45 rows containing missing values or values outside the scale range (`geom_bar()`). Genotypeによってサンプル数が違うので、NAが45行あるとWarningが出てきましたが、図が描けました。

よく見ると、平均値ではなく、最大値でグラフを描いています。Valueを上書きして描いているようです。
こういう初見で気づかない適当な回答をしてくるのが、ChatGPTの怖いところです!(そもそも平均値でグラフを描くように指定していないのですが…)
ChatGPTは便利なツールですが、鵜呑みにしてしまうとこういうところがあるので、必ず一つ一つの命令文を確認して何をやっているのか理解しながら使っていきましょう。
では、(平均値でグラフを描画して)エラーバーも足してもらいましょう。

と入力すると、以下のコードが返ってきます。
# Load necessary libraries
library(ggplot2)
library(dplyr)
library(tidyr)
# File paths
control_file <- "/mnt/data/longday_control.csv"
low_temp_file <- "/mnt/data/longday_low_temperature.csv"
# Read the CSV files
control_data <- read.csv(control_file)
low_temp_data <- read.csv(low_temp_file)
# Add a column to identify the condition in each dataset
control_data$Condition <- 'Control'
low_temp_data$Condition <- 'Low Temperature'
# Combine the two datasets into one
combined_data <- rbind(control_data, low_temp_data)
# Convert from wide to long format
combined_data_long <- combined_data %>%
pivot_longer(cols = c("Col", "tfl1.1", "tfl1.11", "tfl1.13", "tfl1.14", "tfl1.17"),
names_to = "Genotype", values_to = "Value")
# Ensure the 'Genotype' and 'Condition' columns are factors
combined_data_long$Genotype <- as.factor(combined_data_long$Genotype)
combined_data_long$Condition <- as.factor(combined_data_long$Condition)
# Calculate the mean and standard deviation for each genotype and condition
summary_data <- combined_data_long %>%
group_by(Genotype, Condition) %>%
summarise(
mean_value = mean(Value, na.rm = TRUE),
sd_value = sd(Value, na.rm = TRUE)
)
# Plot with error bars
ggplot(summary_data, aes(x = Genotype, y = mean_value, fill = Condition)) +
geom_bar(stat = "identity", position = "dodge") +
geom_errorbar(aes(ymin = mean_value - sd_value, ymax = mean_value + sd_value),
position = position_dodge(0.9), width = 0.25) +
labs(title = "Comparison of Control vs Low Temperature by Genotype",
x = "Genotype",
y = "Mean Value with SD Error Bars") +
theme_minimal() +
scale_fill_manual(values = c("Control" = "blue", "Low Temperature" = "red")) +
theme(axis.text.x = element_text(angle = 45, hjust = 1))必要な部分は平均値とS.D.の計算、および、それを足してグラフを再描画する後半部分です。
# Calculate the mean and standard deviation for each genotype and condition
summary_data <- combined_data_long %>%
group_by(Genotype, Condition) %>%
summarise(
mean_value = mean(Value, na.rm = TRUE),
sd_value = sd(Value, na.rm = TRUE)
)
# Plot with error bars
ggplot(summary_data, aes(x = Genotype, y = mean_value, fill = Condition)) +
geom_bar(stat = "identity", position = "dodge") +
geom_errorbar(aes(ymin = mean_value - sd_value, ymax = mean_value + sd_value),
position = position_dodge(0.9), width = 0.25) +
labs(title = "Comparison of Control vs Low Temperature by Genotype",
x = "Genotype",
y = "Mean Value with SD Error Bars") +
theme_minimal() +
scale_fill_manual(values = c("Control" = "blue", "Low Temperature" = "red")) +
theme(axis.text.x = element_text(angle = 45, hjust = 1))エラーバー付きのグラフが描けました。Boxの高さも平均値になっています。
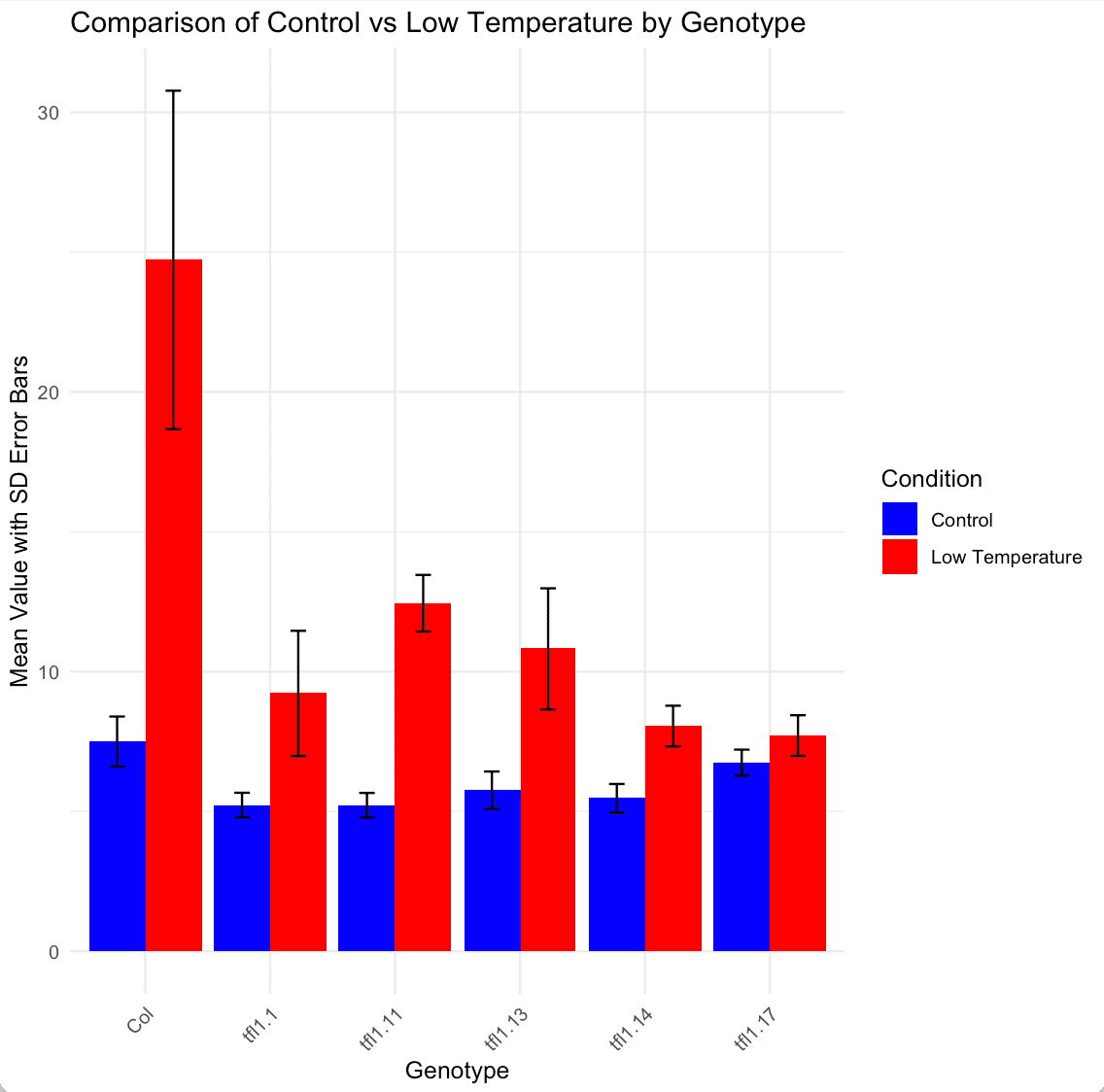
エラーバーがS.E.ではなく、S.D.でエラーバーがついていることに注意してください。
S.E.の場合には、S.E. = S.D./√n で計算させましょう。
次は箱ヒゲグラフにしてもらいましょう。

# Load necessary libraries
library(ggplot2)
library(dplyr)
library(tidyr)
# File paths
control_file <- "/mnt/data/longday_control.csv"
low_temp_file <- "/mnt/data/longday_low_temperature.csv"
# Read the CSV files
control_data <- read.csv(control_file)
low_temp_data <- read.csv(low_temp_file)
# Add a column to identify the condition in each dataset
control_data$Condition <- 'Control'
low_temp_data$Condition <- 'Low Temperature'
# Combine the two datasets into one
combined_data <- rbind(control_data, low_temp_data)
# Convert from wide to long format
combined_data_long <- combined_data %>%
pivot_longer(cols = c("Col", "tfl1.1", "tfl1.11", "tfl1.13", "tfl1.14", "tfl1.17"),
names_to = "Genotype", values_to = "Value")
# Ensure the 'Genotype' and 'Condition' columns are factors for proper plotting
combined_data_long$Genotype <- as.factor(combined_data_long$Genotype)
combined_data_long$Condition <- as.factor(combined_data_long$Condition)
# Plot: Boxplot comparing control and low temperature conditions by genotype
ggplot(combined_data_long, aes(x = Genotype, y = Value, fill = Condition)) +
geom_boxplot(position = position_dodge(0.9)) +
labs(title = "Boxplot: Control vs Low Temperature by Genotype",
x = "Genotype",
y = "Value") +
theme_minimal() +
scale_fill_manual(values = c("Control" = "blue", "Low Temperature" = "red")) +
theme(axis.text.x = element_text(angle = 45, hjust = 1))geom_boxplot( )を使っています。
これも必要なコードは最後の部分だけですね。
# Plot: Boxplot comparing control and low temperature conditions by genotype
ggplot(combined_data_long, aes(x = Genotype, y = Value, fill = Condition)) +
geom_boxplot(position = position_dodge(0.9)) +
labs(title = "Boxplot: Control vs Low Temperature by Genotype",
x = "Genotype",
y = "Value") +
theme_minimal() +
scale_fill_manual(values = c("Control" = "blue", "Low Temperature" = "red")) +
theme(axis.text.x = element_text(angle = 45, hjust = 1))
ヴァイオリンプロットにもしてもらいましょう。

# Load necessary libraries
library(ggplot2)
library(dplyr)
library(tidyr)
# File paths
control_file <- "/mnt/data/longday_control.csv"
low_temp_file <- "/mnt/data/longday_low_temperature.csv"
# Read the CSV files
control_data <- read.csv(control_file)
low_temp_data <- read.csv(low_temp_file)
# Add a column to identify the condition in each dataset
control_data$Condition <- 'Control'
low_temp_data$Condition <- 'Low Temperature'
# Combine the two datasets into one
combined_data <- rbind(control_data, low_temp_data)
# Convert from wide to long format
combined_data_long <- combined_data %>%
pivot_longer(cols = c("Col", "tfl1.1", "tfl1.11", "tfl1.13", "tfl1.14", "tfl1.17"),
names_to = "Genotype", values_to = "Value")
# Ensure the 'Genotype' and 'Condition' columns are factors for proper plotting
combined_data_long$Genotype <- as.factor(combined_data_long$Genotype)
combined_data_long$Condition <- as.factor(combined_data_long$Condition)
# Plot: Violin plot comparing control and low temperature conditions by genotype
ggplot(combined_data_long, aes(x = Genotype, y = Value, fill = Condition)) +
geom_violin(position = position_dodge(0.9), trim = FALSE) +
labs(title = "Violin Plot: Control vs Low Temperature by Genotype",
x = "Genotype",
y = "Value") +
theme_minimal() +
scale_fill_manual(values = c("Control" = "blue", "Low Temperature" = "red")) +
theme(axis.text.x = element_text(angle = 45, hjust = 1))
controlの22℃が寒色系の青で、low temperature (16℃) が暖色系の赤なのが気になります。
c("Control" = "blue", "Low Temperature" = "red")の部分を
c("Control" = "pink", "Low Temperature" = "light blue")に書き換えましょう。
> ggplot(combined_data_long, aes(x = Genotype, y = Value, fill = Condition)) +
+ geom_violin(position = position_dodge(0.9), trim = FALSE) +
+ labs(title = "Violin Plot: Control vs Low Temperature by Genotype",
+ x = "Genotype",
+ y = "Value") +
+ theme_minimal() +
+ scale_fill_manual(values = c("Control" = "pink", "Low Temperature" = "light blue")) +
+ theme(axis.text.x = element_text(angle = 45, hjust = 1))
という具合に、Rの関数を忘れていても、ChatGPTと相談しながら、スムーズにグラフを描くことができました。
P. S.
試しに、違う尋ね方をすると、異なる方法が返ってきたりするので面白いですね。


帰ってきた回答は以下のコードです。
# Load necessary libraries
library(ggplot2)
library(reshape2)
# Data for 22℃ (Flowering leaf counts for different genotypes)
data_22C <- data.frame(
Col = c(6, 6, 7, 7, 7, 7, 7, 7, 8, 8, 8, 8, 8, 8, 9, 9),
tfl1_1 = c(5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 6, 6, 6, 6, 6, 7),
tfl1_11 = c(5, 5, 5, 5, 5, 5, 6, 6, 6, NA, NA, NA, NA, NA, NA, NA),
tfl1_13 = c(5, 5, 5, 5, 5, 6, 6, 6, 6, 6, 6, 6, 6, 6, 6, 7),
tfl1_14 = c(5, 5, 5, 5, 5, 5, 5, 5, 6, 6, 6, 6, 6, 6, NA, NA),
tfl1_17 = c(7, 7, 7, 6, 7, 7, 7, 6, NA, NA, NA, NA, NA, NA, NA, NA)
)
# Data for 16℃ (Flowering leaf counts for different genotypes)
data_16C <- data.frame(
Col = c(18, 21, 22, 20, 17, 17, 16, 18, 28, 28, 30, 28, 26, 28, 30, 32, 34, 32),
tfl1_1 = c(6, 9, 9, 8, 8, 7, 8, 7, 8, 6, 13, 10, 10, 12, 13, 11, 12, 9),
tfl1_11 = c(13, 11, 11, 13, 13, 12, 14, 13, 12, NA, NA, NA, NA, NA, NA, NA, NA, NA),
tfl1_13 = c(6, 7, 8, 12, 12, 12, 13, 10, 12, 12, 9, 11, 12, 12, 12, 13, NA, NA),
tfl1_14 = c(7, 9, 8, 8, 7, 8, 8, 9, 8, 8, 7, 8, 8, 7, 8, 9, 9, 9),
tfl1_17 = c(8, 8, 8, 9, 8, 8, 7, 8, 8, 8, 7, 6, 8, 7, NA, NA, NA, NA)
)
# Add a column for temperature condition and melt the data for plotting
data_22C$Temp <- '22C'
data_16C$Temp <- '16C'
# Combine both datasets
data_combined <- rbind(melt(data_22C, id.vars = 'Temp'), melt(data_16C, id.vars = 'Temp'))
# Rename columns for clarity
colnames(data_combined) <- c('Temperature', 'Genotype', 'LeafCount')
# Plot the data using ggplot2
ggplot(data_combined, aes(x = Genotype, y = LeafCount, color = Temperature, group = interaction(Genotype, Temperature))) +
geom_line(aes(linetype = Temperature), size = 1) +
geom_point(size = 3) +
labs(title = 'Comparison of Flowering Leaf Count at 16°C and 22°C',
x = 'Genotype', y = 'Number of Leaves at Flowering') +
theme_minimal() +
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
scale_color_manual(values = c('22C' = 'blue', '16C' = 'red')) +
theme(legend.position = "bottom")
先ほどは、まず最初に棒グラフが作成されましたが、今回はドットと線で出力されてきました。

# ライブラリの読み込み
library(ggplot2)
library(reshape2)
# データを定義 (22℃)
data_22C <- data.frame(
Col = c(6, 6, 7, 7, 7, 7, 7, 7, 8, 8, 8, 8, 8, 8, 9, 9),
tfl1_1 = c(5, 5, 5, 5, 5, 5, 5, 5, 5, 5, 6, 6, 6, 6, 6, 7),
tfl1_11 = c(5, 5, 5, 5, 5, 5, 6, 6, 6, NA, NA, NA, NA, NA, NA, NA),
tfl1_13 = c(5, 5, 5, 5, 5, 6, 6, 6, 6, 6, 6, 6, 6, 6, 6, 7),
tfl1_14 = c(5, 5, 5, 5, 5, 5, 5, 5, 6, 6, 6, 6, 6, 6, NA, NA),
tfl1_17 = c(7, 7, 7, 6, 7, 7, 7, 6, NA, NA, NA, NA, NA, NA, NA, NA)
)
# データを定義 (16℃)
data_16C <- data.frame(
Col = c(18, 21, 22, 20, 17, 17, 16, 18, 28, 28, 30, 28, 26, 28, 30, 32, 34, 32),
tfl1_1 = c(6, 9, 9, 8, 8, 7, 8, 7, 8, 6, 13, 10, 10, 12, 13, 11, 12, 9),
tfl1_11 = c(13, 11, 11, 13, 13, 12, 14, 13, 12, NA, NA, NA, NA, NA, NA, NA, NA, NA),
tfl1_13 = c(6, 7, 8, 12, 12, 12, 13, 10, 12, 12, 9, 11, 12, 12, 12, 13, NA, NA),
tfl1_14 = c(7, 9, 8, 8, 7, 8, 8, 9, 8, 8, 7, 8, 8, 7, 8, 9, 9, 9),
tfl1_17 = c(8, 8, 8, 9, 8, 8, 7, 8, 8, 8, 7, 6, 8, 7, NA, NA, NA, NA)
)
# 温度条件の列を追加してデータを「縦長」形式に変換
data_22C$Temp <- ‘longday_control’
data_16C$Temp <- ‘longday_low_temperature’
# データを結合
data_combined <- rbind(melt(data_22C, id.vars = 'Temp'), melt(data_16C, id.vars = 'Temp'))
# 列名を変更
colnames(data_combined) <- c('Temperature', 'Genotype', 'LeafCount')
# ggplot2を使ってヴァイオリンプロットを作成
ggplot(data_combined, aes(x = Genotype, y = LeafCount, fill = Temperature)) +
geom_violin(trim = FALSE) +
geom_boxplot(width = 0.1, position = position_dodge(0.9)) +
labs(title = 'Comparison of Flowering Leaf Count at 16°C and 22°C',
x = 'Genotype', y = 'Number of Leaves at Flowering') +
theme_minimal() +
theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
scale_fill_manual(values = c('longday_control' = 'lightblue', 'longday_low_temperature' = 'pink')) +
theme(legend.position = "bottom")回答のうち、ggplot以下の部分で再描画してみます。
よくみると、ヴァイオリンプロットにboxplotも重ねていますね。
> ggplot(data_combined, aes(x = Genotype, y = LeafCount, fill = Temperature)) +
+ geom_violin(trim = FALSE) +
+ geom_boxplot(width = 0.1, position = position_dodge(0.9)) +
+ labs(title = 'Comparison of Flowering Leaf Count at 16°C and 22°C',
+ x = 'Genotype', y = 'Number of Leaves at Flowering') +
+ theme_minimal() +
+ theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
+ scale_fill_manual(values = c('16C' = 'lightblue', '22C' = 'pink')) +
+ theme(legend.position = "bottom")
geom_boxplot(width = 0.1, position = position_dodge(0.9)) +の部分を取り除いて再描画してみます。
> ggplot(data_combined, aes(x = Genotype, y = LeafCount, fill = Temperature)) +
+ geom_violin(trim = FALSE) +
+ labs(title = 'Comparison of Flowering Leaf Count at 16°C and 22°C',
+ x = 'Genotype', y = 'Number of Leaves at Flowering') +
+ theme_minimal() +
+ theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
+ scale_fill_manual(values = c('16C' = 'lightblue', '22C' = 'pink')) +
+ theme(legend.position = "bottom")
逆に、boxplotだけにもできます。
> ggplot(data_combined, aes(x = Genotype, y = LeafCount, fill = Temperature)) +
+ geom_boxplot(width = 0.1, position = position_dodge(0.9)) +
+ labs(title = 'Comparison of Flowering Leaf Count at 16°C and 22°C',
+ x = 'Genotype', y = 'Number of Leaves at Flowering') +
+ theme_minimal() +
+ theme(axis.text.x = element_text(angle = 45, hjust = 1)) +
+ scale_fill_manual(values = c('16C' = 'lightblue', '22C' = 'pink')) +
+ theme(legend.position = "bottom")
ChatGPTの出力結果を100%鵜呑みにするのは危険ですが、回答のコードを眺めながらトライアンドエラーで描画してみたり、Rの関数を調べたりすることで、勉強できて便利な世の中になったものです。
