
マウスの糞便中のウイルス群集に対する抗生物質による摂動の影響
マウスの糞便中のウイルス群集に対する抗生物質による摂動の影響
Jacqueline Moltzau Anderson, Tim Lachnit, Simone Lipinski, Maren Falk-Paulsen, Philip Rosenstiel 著者ノート
G3 Genes|Genomes|Genetics, jkac293, https://doi.org/10.1093/g3journal/jkac293
掲載:2022年11月22日 記事履歴

pdfPDF
閲覧数
引用
アクセス許可 アイコン アクセス許可
共有
要旨
ウイルスおよびバクテリオファージは、腸管バリア機能および常在細菌群の構成と機能特性に強い影響を及ぼす。糞便中のウイルス群の変動は、炎症性腸疾患(IBD)を含むヒトの疾病に関与している可能性がある。NOD2(nucleotide-binding oligomerization domain-containing protein 2)遺伝子の機能欠損変異は、慢性IBDのサブタイプであるクローン病の発症リスク上昇と関連しており、そこでも糞便中のウイルス群集の特異的変化が記述されている。腸管ウイルスの動態に関する理解を深めるため、野生型C57BL/6JマウスとNOD2ノックアウトマウスの糞便サンプルについて、抗生物質による擾乱に対するウイルス群の縦断的な特徴を調べた。ウイルス様粒子の配列決定により、真核生物ウイルスとバクテリオファージからなるマウス糞便ウイルス群の高い多様性と個体間の高い変動が示された。抗生物質は糞便中のウイルス群に大きな影響を与えた。ウイルス群集の組成は,遺伝子型に関係なく観察期間(抗生物質投与停止後10週間)中に部分的にしか回復しなかった.しかし、ウイルスとバクテリオームの組成のシフトは高い相関があり、特定のファージの喪失が細菌群集組成の長期的な調節異常に寄与している可能性が示唆された。このことから、糞便中のビロームに対する治療的干渉は、微生物叢を標的とした治療法における新たなアプローチとなる可能性があることが示唆された。
ビローム、腸内バクテリオファージ、抗生物質擾乱、NOD2、レジリエンス
課題区分 調査研究
編集者:Bアンドリュース
はじめに
ウイルスは、腸内細菌叢の重要な構成要素である(Reyes et al.2010、Minot et al.2011)。ほとんどがバクテリオファージで構成されるこのコミュニティは、疾患がない限り、時間的に安定しています(Reyes et al.2010、Minot et al.2011)。腸内細菌叢の変化は、ヒトの再発性慢性炎症性疾患である炎症性腸疾患(IBD)と関連していますが、その正確な原因はいまだ解明されていません。欧米の工業化されたライフスタイルを含む環境要因が、微生物叢のシフトを引き起こし、遺伝的に感受性の高い宿主における腸の炎症反応の異常に寄与している可能性を示唆する証拠があります(Chang 2020)。最初のクローン病感受性遺伝子であるnucleotide-binding and oligomerization domain 2(NOD2)は、この概念の礎となるものである。生理的には、NOD2はペプチドグリカン断片であるムラミルジペプチドの細胞内センサーである(Girardin et al.2003; Inohara et al.2003)。NOD2は、腸内細菌叢の監視と制御に関与しており、ディフェンシン、DMBT1、活性酸素種などのバリア保護分子の発現を制御している (Wehkamp et al. 2004; Kobayashi et al. 2005; Rosenstiel et al. 2007; Lipinski et al. 2009)。いくつかの研究は、NOD2がウイルス感染の認識にも関与している可能性を示唆している(Sabbahら、2009年;Lupferら、2013年)。NOD2のセンサードメインにおける遺伝的機能喪失変異は、クローン病に対する感受性の増加と関連している(Hugotら、2001;Oguraら、2001)。
以前、我々は、NOD2が腸内細菌叢(バクテリオームおよびマイコバイオーム)の回復力を制御する役割を担っている証拠を示し、それによって、NOD2欠損マウスにおける抗生物質摂動後の細菌叢の回復動態の障害が、腸粘膜の炎症を起こしやすい状態に寄与していることを示した(Moltzau Anderson et al 2020)。このような生理的平衡の回復能力(すなわち、健康なヒトの生理機能を支える長期的な群集の安定性と安定した生態系サービス)の変化は、慢性炎症性疾患やその他の腸疾患の病因に関与していると考えられる(Moltzau Anderson et al.2020)。この仮説を支持するものとして、IBD患者の腸内細菌叢の多様性と機能的特性が健常者と比較して高い時間変動を示し、宿主の恒常性が失われる可能性を示すことが、いくつかのヒトコホート研究で示されている(Ott et al.2004、Manichanh et al.2006、Frank et al.2007、Willing et al.2010)。
そこで我々は、NOD2の機能欠損が腸内ビロームに直接的な影響を及ぼす可能性があると仮定した。バクテリオームとは対照的に、外部事象が短期間(すなわち抗生物質の短期コース)腸内生態系に明確な選択圧をもたらす特定のパルス摂動に反応した糞便ウイルス群集についてはほとんど知られていない(Sommer et al.2017)。ファージは、細菌群集の多様性に影響を与えたり、進化的変化を促したり、細菌宿主に選択的な利点を与えたりと、様々な影響を与えることが分かっています(Abeles et al.2015)。特定の抗生物質による細菌分類群のシフトが直接的に腸内ビローム組成の二次的変化を引き起こすことは明らかであるが、常駐するバクテリオファージが負の選択によって細菌群集回復のダイナミクスに重要なレベルの制御を及ぼしていると考えられる(Abeles et al.2015)。さらに、腸管真核生物ウイルスであるマウスノロウイルスは、無菌マウスおよび抗生物質処理マウスにおいて、抗生物質による処理の効果を相殺することにより、常在菌の有益な機能を代替することが示された(Kernbauer et al.2014)。このように、ウイルスは抗生物質の擾乱に対応する腸のホメオスタシスの調節に重要な役割を果たすこともできる。
また、ウイルスは、その遺伝物質、ゲノムサイズ、ライフサイクル、感染経路、または持続性が異なり、極めて多様であることが示されている(Sharp 2002; Colson et al.2013; Popgeorgiev et al.2013; Colson et al.2017 )。ヒトは、真核細胞に感染するウイルス(真核ウイルス)と細菌に感染するウイルス(バクテリオファージ)からなるウイルスの大集団にコロニー化されています(Popgeorgiev et al.) ヒトの糞便には、1グラムあたり少なくとも109個のウイルス様粒子(VLP)が含まれると推定され(Minot et al. 2013)、これらのウイルスの多くはバクテリオファージとして同定されているものの、大部分は未同定のままです(Reyes et al. 2010, 2012; Minot et al. 2011)。さらに、糞便細菌のゲノムにはウイルス由来の遺伝要素(プロファージやレトロウイルス要素など)が潜んでおり、これらが活性化することでさらなる複雑化につながる可能性がある(Belshawら2004; Popgeorgievら2013; Handley 2016)。ヒト腸内ウイルスのメタゲノム解析により、極端な個体間多様性(=個体間の多様性)が明らかになった。これは、腸内に存在する細菌株に既にかなりの個人差があり、それに対してファージ捕食者の違いが影響していると考えられる(Rodriguez-Valeraら2009;Minotら2011)。ファージが異なる細菌に対して高い選択性を示すことはよく知られており、そのため、ファージ感受性(ファージタイピング)は、異なる細菌株を区別する効果的な手段として数十年にわたって使用されてきました(Sell et al.) また、宿主内ウイルスの急速な進化も、個体間の大きな変動に影響を与えている可能性がある。成体個体のウイルスコミュニティを調査する長期的な研究では、バクテリオファージのファミリーであるマイクロウイルス科は、置換率が高く、研究の終了までに新しいウイルス種を区別するのに十分な配列の発散値を引き起こすことが実証された(Minot et al.2013)。さらに、個々のビロームの構成は比較的安定していることが示唆されており、2.5年の研究期間を通して80%のウイルス形態が持続すると推定されている(Minot et al. 2013)が、同様の結果はより短期の研究でも観察されている(Reyes et al. 2010; Minot et al. 2011)。
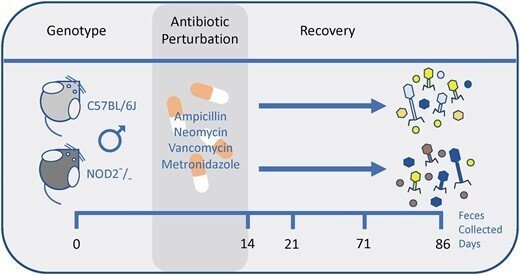
ここでは、C57BL/6J野生型(WT)およびNOD2ノックアウト(KO)マウスにおける糞便ウイルス群集の縦断的変動に対する抗生物質の摂動の影響を調べた(図1)(Moltzau Anderson et al.2020)。糞便ウイルス群のこのような明確な時間変化を解明することは、この十分に説明されていない微生物叢の層の役割と腸の健康への影響を理解する上で極めて重要であると思われる。
図1.
NOD2 KO(n = 3)およびC57BL/6J WT(n = 3)雄マウスを12日間(2~14日目)、抗生物質(アンピシリン、ネオマイシン、バンコマイシン、メトロニダゾール)の組み合わせで処理し、障害を与えた研究計画。マウスはその後、ウイルスコミュニティの回復を調査するために合計86日間モニターされた(14日目以降)。糞便サンプルは、0日目(治療前)、14日目(治療中)、21日目、71日目、86日目(回復期)に採取された。以前、我々はこの同じモデルで細菌と真菌のコミュニティを調査した(Moltzau Anderson et al.)
新しいタブで開くスライドをダウンロード
研究デザイン NOD2 KO(n = 3)およびC57BL/6J WT(n = 3)雄マウスを12日間(2~14日目)、抗生物質の組み合わせ(アンピシリン、ネオマイシン、バンコマイシン、メトロニダゾール)による治療で摂食させたことを実証した。マウスはその後、ウイルスコミュニティの回復を調査するために合計86日間モニターされた(14日目以降)。糞便サンプルは、0日目(治療前)、14日目(治療中)、21日目、71日目、86日目(回復期)に採取された。以前、我々はこの同じモデルで細菌および真菌コミュニティを調査した(Moltzau Anderson et al.2020)。
材料と方法
動物実験
すべての動物実験は、シュレスヴィヒ・ホルシュタイン連邦省の地方動物安全審査委員会の承認を受け、国内外の法律と方針に従って実施した[V 312-72241.121-33 (95-8/11) ]。全ての動物は、キールのクリスチャン・アルブレヒト大学のマウス施設に収容し、実験は以前に記載したように実施した(Moltzau Andersonら、2020)。簡潔には、単一のNOD2欠損雄マウスをC57BL/6J雌と交配してヘテロ接合体子孫を得(F1)、そこからWTおよびNOD2 KOブリーダー対を生成した(F2)。その後、次の2世代の雄の子孫を使用し、単一ケージで特定の病原体のない条件下で維持した。試験開始時(0日目)、マウスは約52週齢であった。マウスの腸内細菌叢に著しい摂動を引き起こすために、C57BL/6J WTおよびNOD2 KO雄マウスを、アンピシリン(1g/l)、バンコマイシン(500mg/l)、ネオマイシン(1g/l)、およびメトロニダゾール(1g/l)(Sigma-Aldrich)からなる広範囲な抗生物質を2週間処理することによって常在菌枯渇プロトコルの変形を使用した(Fagarasan et al. 2002; Rakoff-Nahoum et al. 2004; Vijay-Kumar et al. 2010)を新たに調製し、遮光瓶に入れた飲料水にアドリビタブルに投与した。糞ペレットは、試験期間中の86日間を通して直ちに採取し、必要なときまで-80℃で保存した。マウスは定期的に観察、体重測定し、試験終了時(86日目)に犠牲とした。飼育環境とサンプルの詳細については、補足表1を参照されたい。
ウイルスサンプルの処理
サンプルあたり2つの糞ペレットを、0.01M硫化ナトリウムおよび10mM EDTAを含む15mlのPBS緩衝液に、氷上で30分間再懸濁させた。サンプルを低速で2回遠心分離し(ThermoScientific Heraeus Multifuge 3SR)、4℃で30分間、細菌と汚染された植物材料を除去した。得られた上清を滅菌ろ過し、22,000rpm(Beckman SW41ローター)、4℃で2時間超遠心した。次に、ウイルスペレットを200μlトリス緩衝液(50mM Tris、5mM CaCl2、1.5mM MgCl2、pH 8. 0)に再懸濁し、そこから分離したウイルスの5 µlサブサンプルを採取し、2% (w/v) 水性酢酸ウラニルでのネガティブ染色による形態学的特性評価を行い、透過電子顕微鏡 (TEM) (Technai Bio TWIN) で80 kV、倍率4万~10万で可視化した。サンプルに2 µlのベンゾナーゼを加え、37℃で2時間インキュベートし、残存する核酸のコンタミネーションを除去した。
ウイルスDNAとRNAを抽出するために、22 µlの2M Tris-HCl (pH 8.5)/0.2 M EDTAの0.1容量、10 µlの0.5 M EDTA、268 µlのホルムアミドを加え、30分間、RTでインキュベートした。その後、1 µl のグリコーゲン、1,024 µl のエタノールを加え、サンプルを穏やかに混合し、常温で一晩インキュベートした。翌朝、サンプルを12,000×g、4℃で20分間遠心分離し、70%エタノールで洗浄後、100μlのTEバッファと1μlのメルカプトエタノールに再懸濁し、10μlの10%SDSと3μlのProteinase Kを加え、37℃で20分、56℃で15分インキュベートした。その後、400 µl の DNA 抽出バッファー CTAB (100 mM Tris pH 8.0, 1.4 M NaCl, 20 mM EDTA, 2% CTAB) と 1 µl の mercaptoethanol を加え、サンプルを 56℃で 15 分間インキュベートした。得られた上清に等量のクロロホルム:イソアミルアルコール(24:1)を加え、サンプルを13,000×gで5分間遠心分離した。上清を回収し、1 µl のグリコーゲン、10 µl のメルカプトエタノール、0.7 容量のイソプロパノールを加え、-20℃で一晩インキュベートした。翌朝、13,000×g、4℃で20分間遠心分離した後、上清を回収し、500μlの70%エタノールで洗浄し、-80℃に保存した。
VLPの抽出後、サンプルからエタノールを除去し、ペレットを風乾して20μlのRNAseフリーの濾過水に再懸濁させた。増幅は、以前に記載されたように、改良されたComplete Whole Transcriptome Amplification Kit(WTA2)(Sigma-Aldrich)を用いて行った(Conceição-Netoら、2015)。その後、GenElute PCR Clean-Up Kit (Sigma-Aldrich)を用いてPCR産物を精製した。サンプルは、ライブラリ構築の前に-20℃で一晩保存した。
ライブラリー構築
ライブラリーは、NexteraXTキット(Illumina)を用いて作成した(Conceição-Neto et al.2015)。定量化後、全サンプルの正規化プールを、2×150bp配列決定キット(Illumina)を用いてIllumina MiSeqで配列決定した。このWhole Genome Shotgunプロジェクトは、BioSampleアクセッション番号SAMN08534315からSAMN08534344とともに、NCBI BioProjectデータベース(https://www.ncbi.nlm.nih.gov/bioproject/?term=PRJNA434045)のBioProjectアクセッション番号PRJNA434045にリンクして寄託されています。
ウイルス群集の構成
Nextera XTアダプターを除去し、Trimomatic V.0.36 (Bolger et al. 2014)を用いてIlluminaペアエンドリード(2×150 bp)から配列リードをトリミングした。トリミングして品質管理した全サンプルのリードを、SPAdes V.3.1.10 (Bankevich et al. 2012) を用いてクロスアセンブルし、リファレンスウイルスメタゲノムを生成しました。コンティグはNCBIヌクレオチドデータベースに対してblastnを使用してコンタミネーションをスクリーニングした(Pruitt et al. 2012)。同一性が90%以上、長さが50%以上のコンティグは、配列内にウイルスのホールマーク遺伝子が検出されない場合に削除された。すべてのマウス糞便ウイルスのシーケンスリードを1,094,102コンティグにアセンブルした。参照ゲノムについては、1,000 bp以上の長さでカバレッジが10以上のコンティグを4,767個選択した。これらのコンティグを選択し、e値を10-5でカットオフした5,571,160個のウイルス配列を含むUniProtウイルスデータベースに対してblastxを用いて解析した(Bateman et al.2017)。参照ビロームの平均配列長は3,358、カバレッジは80であった。これらのコンティグのうち、48%はUniprotウイルスデータベースを用いて既知のウイルス配列に割り当てられた。最後に、参照ウイルスメタゲノムをVirSorter2 (Guo et al. 2021)でさらに分類した。ファージ構成の割り当てをさらに洗練するために、ツールvConTACT(Bolducら、2017)を使用して、すべてのタンパク質を配列と比較し、類似性ネットワークを生成して、BLASTP(Altschulら、1997)を使用してタンパク質のクラスタ(PC)にゲノムを識別した。次に、類似配列のグループを、インフレーション値2のMarkovクラスタリングアルゴリズムを用いてクラスタリングし、ウイルスクラスタ(VC)を作成した。組織化されたVCとPCから特定された関係情報を使用してモジュールプロファイルを作成し、分類学的同定のために分類することができました(Bolduc et al.2017)。ネットワークはCytoscape V.3.1.1 (Shannon et al. 2003)で可視化し、より多くのPCを共有するゲノムをより近くに配置した(補足図1)。
さらに、すべてのコンティグをコンティグアノテーションツールCAT(Von Meijenfeldt et al. 2019)で分類し、Rapid Annotation using Subsystem Technology(RAST)に提出して追加のウイルスのホールマーク遺伝子を同定した。さらに、VirHostMatcherNet(Wang et al. 2020)およびCAT(Von Meijenfeldt et al. 2019)を使用して、ウイルスと原核生物の相互作用を予測した。偽陽性の影響を減らすため、VirSorter2、CAT、RASTアノテーションに基づいてウイルス配列として割り当てられたコンティグのみにウイルスコミュニティ分析を集中させた。ウイルスと分類されたコンティグは、マウス糞便ウイルス群集を代表するOTUとして使用された。このサブセットは、約94%がdsDNAウイルス、5%がssDNAウイルス、1%がRNAウイルスからなる614個のコンティグから構成されている。各サンプルのリードは、Bowtie2 (Langmead and Salzberg 2012) とSAM tools (Li et al. 2009) を用いて、代表的なマウスウイルスOTUに対して個別にマッピングされた。各OTUの正規化カバレッジを、サンプルあたりの各ウイルスの相対的存在度の代理として使用した(Roux et al.2016)。
ウイルス群集組成は、コンピュータソフトウェアPRIMER V.7 (Clarke 1993; Anderson et al. 2008; Clarke and Gorley 2015) を用いて解析し、存在量データは標準化し、log + 1変換した。全サンプル間の類似度の推定は,Bray-Curtis類似度と非計量多次元尺度分析(MDS)により計算し,異なる処理群および時点間のウイルス群集組成のペアワイズ比較は,類似度検定(ANOSIMグローバル検定)(Clarke 1993)を用いて解析した.SIMPER解析は、観察された群集組成の違いに寄与する最も重要なウイルスOTUを検出するために使用された。これらの事前選択されたOTUはさらに、コンピュータソフトウェアIBM SPSS Statisticsを用いて、1因子ANOVAに続いてTukeyの正直有意差(HDS)検定で分析された。
ウイルス群集と細菌群集の関係
細菌群集の構成によって説明できるウイルス群集の変動、またはその逆を調べるために、コンピュータソフトウェアPRIMER V.7(Clarke 1993; Clarke and Gorley 2015)のRELATE分析(Anderson et al.2008) を使用した。この解析は、ウイルスと細菌のOTUの相対的な存在量に基づくものであった。生の細菌FASTQリードは、我々の以前の研究(Moltzau Anderson et al. 2020)からEBIのENAからAccession Number PRJEB21817(http://www.ebi.ac.uk/ena/data/view/PRJEB21817)の下で入手した。ウイルスおよび細菌群集のデータセットは標準化され、log(x + 1)変換された。ウイルス群集によって説明できる細菌群集組成の変動を調べるために、調整済みR2選択基準と前方選択手順を用いた距離ベースの冗長性モデリング(DISLM)を用いて、最小長10,000 bpの最も豊富な29のウイルスOTUを細菌OTUの相対存在量に適合させた。結果は、距離ベースの冗長性解析(dbRDA)(Anderson 2001; McArdle and Anderson 2001)を用いて可視化した。
結果
VLPの存在
糞便サンプル中のVLPの存在はTEMで観察され、形態的に異なるビリオンが確認された(Fig. 2)。超遠心分離により損傷したものもあるが、多数の多様なバクテリオファージが存在し(図2a〜c)、頭部、すなわちキャプシドと、場合によっては尾部の構造によって区別される(この構造以外のファージ形態も存在する(すなわち尾部がない))。ミオウイルス科の形態は、正二十面体(20面体)の頭部と収縮性の尾を持つものが存在した(図2c)。また、大腸菌によく感染するシホビル科の形態を示すラムダ様ファージ(λ)の構造も観察された(図2a、b)。キャプシドのタンパク質頭部は正20面体(図2b)と細長い形状(図2a)をしており、核酸を含んでいる。頭部は、先端に細長い尾部繊維を持つ尾部と結合している(宿主認識用)。尾部は中空管で構成され、感染時に核酸がこの中空管を通って宿主に侵入する。また、PPR(Peste des Petits Ruminants)ウイルスやMMTV(Murine Mammary Tumor virus)など、真核生物ウイルスに形態が類似したVLPも観察されている(図2d-f)。
図2.
a-c)シホウイルス科およびミオウイルス科のバクテリオファージ。 d-f)PPRウイルスまたはMMTVウイルスに形態的に類似した真核生物ウイルス。
新しいタブで開くスライドをダウンロード
マウス糞便から精製したVLPを2%酢酸ウラニル水でネガティブ染色した透過型電子顕微鏡写真(TEM)。
基準マウス糞便ウイルス群の構成
ウイルスコミュニティーは、主にCaudovirales目(ウイルスコンティグの71%)からなるdsDNAバクテリオファージで主に構成されていた。dsDNAファージコンティグの割り当てをさらに精密化するために、vConTACTを用いて、分類学的同定のための既知のウイルス参照ゲノムとウイルスおよびPCを生成した(補足図1)。ネットワーク解析の結果、Escherichia phage、Enterobacteria phage、Cellulophaga、Microviridae、Lactococcus phageなど、多数のクラスターが描かれた(Supplementary Fig. 1)。ウイルスコンティグの約16%はVirSorter2によってdsDNA真核生物ウイルスであるLavidaviridaeとNucleocytoplasmic large DNA virusと予測された。真核生物ウイルスの誤検出を減らすために、これらのコンティグをblastnとblastxによってNCBIの非冗長タンパク質データベースと比較したところ、そのほとんどが真核生物ではなく原核生物に高い配列類似性を持っていることが分かった。これらの偽陽性真核生物ウイルスは除去された。マウス糞便中に真核生物に感染するウイルス配列が数個同定された。レトロウイルス科のssRNAウイルス1個が、Murine leukemia virus(コンティグ3291)と塩基配列レベルで高い類似性を示すことが判明した。大麦に感染することが知られているEndornaviridae科のdsRNAウイルスHordeum vulgare alphaendornavirus(コンティグ811、1176、3773)が見いだされた。マウスの飼料の食餌組成には、他の植物原料の中に大麦が含まれていた。本研究で同定できたその他の植物関連ウイルスの候補は、Gemycircularvirusと高い配列類似性を有するGenomoviridae科のssDNAウイルス(コンティグ4238およびコンティグ2208)であった。
抗生物質投与後の糞便中のウイルス群集組成の不完全な回復
ウイルスOTUレベルに基づくウイルス群集のMDSは、日にちに基づく明確なクラスタリングを示した(図3)。処理前の0日目のサンプルはクラスター化し、抗生物質処理14日後に群集が有意に変化した(ANOSIM global test for test differences between time points: R statistics = 0.443, P = 0.001)。遺伝子型(WT vs NOD2-KO)間の差は、どの時点でも検出されなかった(ANOSIM global test for test differences between time points: R統計量=-0.03、P=0.753)。抗生物質投与は、ウイルス群集組成を有意に変化させた(ANOSIM Pairwise Test; 補足表2)。0日目から14日目までの平均的な非類似度の20%は、抗生物質処理後にガンマプロテオバクテリアに感染する4つのファージの相対量が増加し、バクテロイデス菌に感染する2つのファージが減少したことによって説明できた(SIMPER分析;補足表3)。その結果、Escherichia属に感染するファージ(contig 14, 32, 186, 3817)が増加し、Bacteroides属に感染すると予測されるファージ(Phage apr34 (contig 52), Microvirus (contig 996))は抗生物質処理後に減少した(n = 6, P < 0.01, one-way ANOVA, Tukey's HDS)ことがANOVAによって確認された。この結果をさらに検証するため、Primer V.7を用いて最も重要な50のウイルスコンティグを同定し、Bray-Curtis類似度を用いた完全連鎖クラスターに基づくシェードプロットを作成した(補足図2)。NMDS解析(図3)と連動して、サンプルは遺伝子型別ではなく日別にクラスタリングされ、0日目(治療前)と14日目(抗生物質治療中)はそれぞれクラスタリングされ、その後21、71、86日目で緩やかに連続するパターンが生じた(Supplement Fig.2)。
図3.
NOD2 KO(三角、n = 3)とC57BL/6J WT(丸、n = 3)のマウス糞便ウイルス群集組成のNonmetric mutidimentional scaling (NMDS) 解析。時間経過に伴うウイルスOTUの種レベルでの相対的存在度のBray-Curtis類似度指数に基づく解析(0日目=治療前、14日目=治療中、21-86日目=治療後)。
新しいタブで開くスライドをダウンロード
NOD2 KO(三角、n = 3)とC57BL/6J WT(丸、n = 3)のマウス糞便ウイルス群集組成のNonmetric mutidimentional scaling (NMDS) 解析。時間経過に伴うウイルスOTUの種レベルでの相対的存在度のBray-Curtis類似度指数に基づく解析(0日目=治療前、14日目=治療中、21-86日目=治療後)。
群集組成の軌跡は、時間の経過とともに、day 0に密集して回復に向かう(つまり、抗生物質投与前の初期群集に近い組成になる)ことがわかった(Fig. 3)。しかし、86日目のウイルス群集組成は完全には回復せず、0日目との平均非類似度86.64(0日目と86日目の差に関するANOSIM Pairwise Test: R statistics = 0.581, P = 0.002)と有意な差が残り、不完全な回復であった(図3、補足図2)。この非類似度のうち、20%はわずか6個のウイルスOTUで説明でき、そのうちMicrovirus (contig 935) とPhage apr34 (contig 52) は抗生物質の摂動後に大きく減少し、試験終了時(86日目)には検出できなかった(Supplementary Table 4)。さらに、ウイルス多様性を抗生物質処理前と処理後で比較すると、0日目(処理前)は、総種数(n = 6, F = 13.525, P < 0.001, 一元配置分散分析、テューキーHDS)、種の豊富さ(マーガレフ)(n = 6, F = 13.527, P < 0.001, 一元配置分散分析、テューキーHDS)ともにウイルスが著しく多様だった(図4)。
図4.
ウイルス多様性 抗生物質処理前(0日目)と処理後(14日目以降)の比較。0日目は、種の豊富さ(マーガレフ)と総種の両方でウイルス多様性が有意に高かった(n = 6, F = 13.525, P < 0.001, one-way ANOVA, Tukey's HDS)。
新しいタブで開くスライドをダウンロード
ウイルス多様性 抗生物質投与前(0日目)と投与後(14日目)の比較。0日目は、種の豊富さ(マーガレフ)および全種において有意に高いウイルス多様性を示した(n = 6, F = 13.525, P < 0.001, one-way ANOVA, Tukey's HDS)。
原核生物ウイルス群集組成の高い個体間変動
糞便中のバクテリオファージは多様であり、その相対的な存在量はマウスの個体間で変動していた。抗生物質投与前(0日目)のファージは、ミクロウイルス科、ファージapr34、その他のバクテロイデーテス属とファーミキューテス属に感染するファージが優勢であった(図5)。抗生物質の投与はファージ組成に強く影響し、大腸菌ファージが優勢な系に移行した。抗生物質の投与を停止してから1週間以内に、ウイルス群集の組成は大きく変動した。また、ウイルス群集組成の個体間変動が大きく、時間経過や遺伝子型間での明確な回復パターンは観察されなかった(図5)。OTUレベルの解析では、ガンマプロテオバクテリア、ファーミキューテス、バクテロイデテスのファージがシフトしていることが確認された。これらのファージ集団のシフトが有意であるかどうかを判断するために、VirHostMatcherNetとCAT分類学を用いて、細菌宿主の予測を行った。すべてのファージは、より高い系統レベルでの細菌宿主予測に基づいてグループ化された(すなわち、ガンマプロテオバクテリア、ファーミキューテス、バクテロイデテス)(Fig. 6)。抗生物質処理前のファージ集団は、バクテロイデーテス属とファーミキューテス属が同程度の割合を占めていました。抗生物質処理中(14日目)に群集組成に大きな変化が生じ、0日目の処理前とは明らかに異なっていた(図3、図5)。この間、ファージ群集はガンマプロテオバクテリアファージで占められていた(図6)。
図5.
NOD2 KO(n = 3)およびC57BL/6J WT(n = 3)マウスにおける原核生物ウイルス群集の経時的構成(0日目 = 前処理、14日目 = 抗生物質処理、21~86日目 = 後処理)。円グラフは、すべての配列リードをクロスアセンブリして作成したマウス糞便リファレンスビロームへの個々の配列リードのマッピングに基づく、原核生物ウイルスコンティグの相対的存在度を示す。ローマ字は個々のマウスを表す。
新しいタブで開くスライドをダウンロード
NOD2 KOマウス(n = 3)およびC57BL/6J WTマウス(n = 3)の原核生物ウイルス群集の経時変化(0日目=前処理、14日目=抗生物質処理、21-86日目=後処理)。円グラフは、すべての配列リードをクロスアセンブリして作成したマウス糞便リファレンスビロームへの個々の配列リードのマッピングに基づく、原核生物ウイルスコンティグの相対的存在度を示す。ローマ数字は個々のマウスを表す。
図6.
Bacteroidetes、Firmicutes、Gammaproteobacteriaファージの相対存在量(%)と対応する宿主細菌の相対存在量(%)を比較したもの。ファージはVirHostMatcherNetとCAT分類学によって予測された宿主細菌によってグループ分けされた。時間は日数で表す(0日目=前処理、14日目=抗生物質処理、21~86日目=後処理)(エラーバー=±2 SE、n=3)。
新しいタブで開くDownload slide
Bacteroidetes、Firmicutes、Gammaproteobacteriaファージの相対存在量(%)と対応する宿主菌の相対存在量(%)を比較した。ファージはVirHostMatcherNetとCAT分類学によって予測された宿主細菌によってグループ分けされた。時間は日数で表した(0日目=前処理、14日目=抗生物質処理、21-86日目=後処理)(エラーバー=±2 SE、n=3)。
抗生物質処理後、Bacteroidetesファージは21日目に44.8%から5.3%に有意に減少した(n = 6, F = 3.475, P = 0.025, one-way ANOVA, Tukey's HDS)。Firmicutesファージの相対量は、抗生物質処理終了時(14日目)には40%から17.8%へとわずかに影響を受けただけで、抗生物質処理前と比較して有意な影響はなかった。回復期(14日目以降)には、Gammaproteobacteriaファージの相対量が減少したが、Firmicutesファージは急速に回復し、21日目には抗生物質処理前と比較して高い相対量に達した(n = 6, F = 5.78, P = 0.013, one-way ANOVA, Tukey's HDS)。これは、71日目(n = 6, F = 20.4, P = 0.001, 一元配置分散分析、テューキーHDS)と86日目(n = 6, F = 18.3, P = 0.002, 一元配置分散分析、テューキーHDS)において、細菌群に見られるシフトと矛盾していた。Firmicutesファージとは対照的に、Bacteroidetesファージの回収は遅く、71日目からしか検出できなかった。興味深いことに、BacteriodetesとFirmicutesの細菌は、抗生物質の影響によって異なる影響を受け、Bacteroidetesは抗生物質処理12日後にほぼ完全に消滅したが、Firmicutes細菌群は14日目にまだ7%の相対量に達していた。
細菌群集とウイルス群集の組成変化には相関があり、細菌群集の組成変化(OTUレベル)はウイルス群集の組成変化と同様の方向と大きさで起こった(RELATE、Rho = 0.416, P = 0.001, 999 Permutations)。抗生物質による摂動後の時間(回復期間)は、両群とも主要な要因であった。29の最も重要なファージの距離ベースモデリング(DISLM)を用いると,Escherichiaファージ(コンティグ14)の相対量が抗生物質摂動から高くなり,細菌群集の変動の25.5%を説明した(図7)。他の4つのバクテリオファージと合わせて、このモデルは細菌変動の49%まで説明することができた(図7;補足表5)。
図7.
NOD2 KO(三角、n = 3)とC57BL/6J WT(丸、n = 3)のマウス糞便ウイルス群集組成を、R2選択基準および前進選択手順を用いたDistLM逐次試験で糞便細菌群集組成に適合させた経時的な拘束型dbRDAプロット。ベクトルオーバーレイの長さは、関連する予測変数の相対的な影響力を示す。時間は日数で表す(0日目=前処理、14日目=抗生物質処理、21日目〜86日目=後処理)。
新しいタブで開くダウンロードするスライド
NOD2 KO(三角、n = 3)とC57BL/6J WT(丸、n = 3)のマウス糞便ウイルス群集組成の時間推移を、R2選択基準と前進選択手順を用いたDistLM連続検定で糞便細菌群集組成に適合させた制約付きdbRDAプロット。ベクトルオーバーレイの長さは、関連する予測変数の相対的な影響力を示す。時間は日数で表した(0日目=前処理、14日目=抗生物質処理、21~86日目=後処理)。
考察
腸内細菌叢の研究は、主にバクテリオームとウイルスに焦点が当てられており、RNAウイルスはほとんどの研究に含まれていない。我々の知る限り、抗生物質投与後のIBDにおけるビロームの回復力特性はこれまで調査されたことがない。我々は、野生型マウスとNOD2 KOマウスの腸内ビロームの動態を、治療前、治療中、治療後の糞便を採取して縦断的に解析した。その結果、予想に反して、遺伝子型間でウイルス群集に有意な差は見られなかった。糞便サンプルは微生物群集の分析に広く用いられており、管腔内容物や粘膜サンプルによってNOD2の表現型や活性に関連する明確な違いが明らかになったかもしれないが、そのためには異なる時点でマウスを犠牲にしなければならず、個体内時間効果の力を失うことになる。しかしながら、マウス糞便中のウイルスには、宿主に感染するウイルス(レトロウイルス科、白血病ウイルスなど)、原核生物に感染するウイルス(バクテリオファージ、Caudoviralesなど)、そのどちらにも感染しないウイルス(植物ウイルス、Hordeum vulgare alphaendornavirusなど)など、形態的・遺伝的に多様であることが明らかにされた。
また、マウスの腸内ビロームは、ヒトの腸内ビロームといくつかの共通点がある。まず、マウス腸内ビロームの個体間変動が大きいことである。この個体間の高いウイルス多様性は、ヒトの腸内ビロームでも以前に報告されている(Reyes et al.2010; Norman et al.2015)。さらに、ヒト腸内ビロームと同様に、マウス腸内ビロームには、真核生物ウイルスの多様性がかなり低いことに加えて、主にバクテリオファージが大きな多様性を持つことが確立されている(Reyesら2010;Minotら2011、2013)。さらに、これまでの報告と一致して、最も豊富なウイルス分類は、Caudovirales目(すなわちCellulophagaファージ)およびMicroviridae科(すなわちParabacteroidesファージ)のバクテリオファージでした(Reyesら、2010;Minotら、2011;Normanら、2015)。
マウスの糞便に関連するRNAウイルスも同定され、最終的にこれらのRNAウイルスだけが真核生物ウイルスであることが確認された。これらの真核生物のウイルスのうち、いくつかは植物を宿主とすることが確認された(すなわち、オオムギに感染することが知られているHordeum vulgare alphaendornavirus)。植物性ウイルスの配列は、大麦、小麦、大豆製品、オート麦の外皮、テンサイのパルプなどを含む雑食性のマウスの食性を反映していると思われ、食性が腸管真核生物ウイルス群の構成に大きく関与していることが推測された。興味深いことに、これらのウイルスの検出は、ファージ多様性が最も低い14日目から多く見られた。さらに、これらの植物ウイルスは常に存在しているが、ファージコミュニティが大きいために検出できなかったと推測される。これまでのほとんどの研究では、分離・抽出時のフィルタリング方法の結果として、大型の真核生物のウイルスの存在は報告されていない(Conceição-Neto et al.2015)。例えば、フィルター法は細菌を効率的に除去しますが、フィルターはミミウイルスの99%以上、ヘルペスウイルスの90%を除去することも示されています(Vijay-Kumar et al.2010)。これらのウイルスは、当初考えられていたよりも多く存在している可能性がありますが、それらが宿主内でどのような役割を果たしているかはまだ分かっていません。また、ウイルス分画に由来する配列が、アセンブルされたレファレンスでは未分類であることが多いことからもわかるように、ウイルスの特徴を明らかにするためのレファレンスの整備が急務であることも述べておきたい。このため、一部のウイルスコンティグ(特に細菌ゲノムのプロファージ)が廃棄された可能性がある。しかし、既知のウイルスの特徴的な遺伝子を持たないコンティグのみが解析から除外された。さらに、ウイルスゲノムの断片化によって、配列の一部が削除される(すなわち、ウイルス特有の遺伝子を含まない部分)が、同じウイルスゲノムの他の部分(例えば、ファージ尾部繊維やシャフトタンパク質を含む)は保持され、最終的に参照ウイルスゲノムにとどまることになる。本研究で作成されたシーケンスカタログは、高品質のほぼ完全なゲノムで構成されており、今後のビロームの研究にとって重要な参考資料となるものである。
さらに、アンピシリン、バンコマイシン、ネオマイシン、メトロニダゾールを組み合わせた広域抗生物質を用いて、常在菌の枯渇を誘導した(Fagarasan et al.2002; Rakoff-Nahoum et al.2004; Vijay-Kumar et al.2010 )。この結果、抗生物質投与中と投与後のウイルス群集組成に大きな変化が生じた。抗生物質はウイルスを直接の標的としないが、我々の結果は、細菌群集の著しい変化を示し、それは糞便中バクテリオファージ群集の変化と一致した。決定的に、マイクロバイオームが抗生物質にどのように反応するかは、摂動前のマイクロバイオームの初期状態(すなわち、宿主要因、微生物種、機能多様性)と摂動の強さと大きさ(すなわち、抗生物質の用量、期間、スペクトル)の両方によって変化します(Sommer et al.2017年)。抗生物質による摂動前、ファージ集団はBacteroidetesファージとFirmicutesファージの割合が等しく、BacteroidetesとFirmicutesによって支配されていた細菌群集を反映していた。抗生物質処理中は、Bacteroidetes系ファージは大きく減少し、Gammaproteobacteria系ファージに置き換わっていた。抗生物質処理中の大腸菌/赤痢菌の伸長により,Lotka-Volterra/Kill-the-Winnerダイナミクスで予想されるように,それぞれのバクテリオファージ(すなわちEscherichia shigellaファージ)がブルームした可能性がある( Rodriguez-Valera et al.2009; Mirzaei and Maurice 2017)。この効果は、抗生物質カクテルの成分としてバンコマイシンを使用することによって引き起こされ、腸内細菌科と大腸菌を含むProteobacteria(Carvalhoら2012;Rooksら2014)の拡大を促進することが以前に示された。注目すべきは、腸内細菌科細菌の濃縮は、ディスバイオーシスの重要な指標であることである。興味深いことに、多数の研究が、NOD2リスクアレルと腸内細菌科の相対存在度の増加との間に有意な関連を示している(Garrettら、2007;Geversら、2014;Knightsら、2014)。しかし、ヒトにおける以前の研究とは異なり、NOD2 KOマウスに特異的なバクテリオファージコミュニティに特異的な変化は起こらなかった(Norman et al.2015)。抗生物質の摂動後、マウスの細菌およびウイルス群集における組成のシフトは有意に相関し、そこでは群集の変化が同様の方向と大きさで起こった。ガンマプロテオバクテリアファージは減少したが、ファーミキューテスファージは増加し、研究の終わりまで残存していた。このようなファージ群集の変化は、ファーミキューテス門の増加を通じて細菌群集にも反映された。一方、バクテロイデーテス属ファージは21日目以降に増加したものの、0日目と比較して相対的な存在量は低いままであった。これは、71日目および86日目に細菌群集のシフトが観察され、ファーミキューテス属細菌よりもバクテロイデット属細菌の相対量が多くなったことと相反する結果であった。抗生物質投与中に消失したクロセイバクターファージがバクテロイデス属細菌の制御因子であった可能性があり、腸内のウイルス-細菌群集動態が複雑であることがさらに明らかになった。しかし、最初の研究デザイン(Moltzau Anderson et al. 2020)から糞便材料が限られていたため、21日目と71日目の間のタイムポイントがなく、イベントの正確なタイミングについての洞察は制限されている。それにもかかわらず、摂動前と研究終了時の両方で、それらの細菌門(すなわちFirmicutesとBacteroidetes)に関連するファージが確認されたことから、これらのコミュニティのウイルス-細菌ダイナミクスが宿主の健康維持に重要な役割を担っていることが示唆される。最終的に、ウイルス群集は、0日目(処理前)に近い構造に再接近しているように見えたものの、回復に障害があった。
我々の研究は、糞便微生物群集の経時的な回復と回復力を研究する実験によるこれまでの観察を拡張するものである(Dethlefsenら、2008年、Davidら、2014年、Heinsenら、2015年、Moltzau Andersonら、2020年)。抗生物質の摂動は、ネズミの糞便ウイルス群集の大幅なシフトを引き起こした。抗生物質処理後、糞便ウイルス群の回復が遅れ、不完全であることが観察され、これは高い個体間変動によって特徴づけられた。NOD2シグナルが機能しない場合、この実験には何の影響もなかった。この結果は、抗生物質投与中および投与後のバクテリオファージ量の動態は、細菌群集のシフトと密接に関連しており、その損失はマイクロバイオームの乱れに寄与する可能性があることを示している。この結果は、健康および疾病における腸内生態系の回復力に寄与する因子をより良く理解することの重要性を浮き彫りにしています。現在、IBDから大腸がんまで、さまざまな腸の疾患において、マイクロバイオータを標的とした治療法が検討されていますが、ビロームの標的化は、その新しいアプローチとなり得るかもしれません。
データの利用可能性
ウイルスおよび細菌のデータセットは、NCBIのBioProjectのアクセッション番号PRJNA434045およびEBIのENAのアクセッション番号PRJEB21817から一般に入手可能である。詳細は、Materials and methodsの項を参照。
補足資料はG3オンラインに掲載されています。
謝辞
技術的な支援をいただいたInstitute of Clinical Molecular Biology (IKMB) のMelanie Vollstedtに感謝する。
資金提供
この研究は、マックス・プランク進化生物学研究所のIMPRS(JMA)、Deutsche Forschungsgemeinschaft (DFG) Excellence Cluster 2167 Precision Medicine in Chronic Inflammation (TI-1 to PR) およびDFG Collaborative Research Center (CRC) 1182 Origin and Function of Metaorganisms, C2 (PR) and C4.2 (TL) によりサポートされています。配列決定はCAU KielのDFG Sequencing Hub CCGAの支援を受けている(PR)。
利益相反
特になし。
引用文献
Abeles SR、Ly M、Santiago-Rodriguez TM、Pride DT. ヒトの口腔内および糞便中のウイルスに対する長期的な抗生物質治療の影響。PLoS One. 2015;10(8):e0134941.
Google ScholarCrossrefPubMedWorldCat
アルトシュールSF、マデンTL、シェーファーAA、チャンJ、チャンZ、ミラーW、リップマンDJ. Gapped BLASTとPSI-BLAST:新世代のタンパク質データベース検索プログラム。Nucleic Acids Res.1997;25(17):3389-3402。
Google ScholarCrossrefPubMedWorldCat
Anderson MJ. ノンパラメトリック多変量分散分析のための新しい方法。ということである。2001;26:32-46.
Google ScholarWorldCat
Anderson MJ, Gorley RN, Clarke KR. PERMANOVA+ for PRIMER: ソフトウェアと統計手法の手引き. Plymouth (UK): PRIMER-E Ltd.; 2008.
Google ScholarGoogle PreviewWorldCatCOPAC
SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing(SPAdesは新しいゲノムアセンブリーアルゴリズムとシングルセルシーケンスへの応用)。2012;19(5):455-477.
Google ScholarCrossrefPubMedWorldCat
Bateman A, Martin MJ, O'Donovan C, Magrane M, Alpi E, Antunes R, Bely B, Bingley M, Bonilla C, Britto R, et al. UniProt: the universal protein knowledgebase.(ユニプロット・ユニバーサルプロテイン・ナレッジベース)。Nucleic Acids Res. 2017;45(Database Issue):D158-D169.
グーグルScholarPubMedWorldCat
Belshaw R, Pereira V, Katzourakis A, Talbot G, Paces J, Burt A, Tristem M. Long-term reinfection of the human genome by endogenous retroviruses.(内因性レトロウイルスによるヒトゲノムの長期的再感染)。Proc Natl Acad Sci USA. 2004;101(14):4894–4899.
Google ScholarCrossrefPubMedWorldCat
このような場合、「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」。PeerJ. 2017;5:e3243.
Google ScholarCrossrefPubMedWorldCat
Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data(イルミナ配列データのための柔軟なトリマー)。Bioinformatics. 2014;30(15):2114–2120.
Google ScholarCrossrefPubMedWorldCat
Carvalho FA, Koren O, Goodrich JK, Johansson ME, Nalbantoglu I, Aitken JD, Su Y, Chassaing B, Walters WA, Gonzalez A, et al. 一過性のプロテオバクテリア管理不能がTLR5欠損マウスで慢性腸炎を促進させる。Cell Host Microbe. 2012;12(2):139-152.
Google ScholarCrossrefPubMedWorldCat
Chang JT. 炎症性腸疾患の病態生理(Pathophysiology of inflammatory bowel diseases). N Engl J Med. 2020;383(27):2652–2664. https://doi.org/10.1056/NEJMra2002697.
Google ScholarCrossrefPubMedWorldCat
クラークKR. を用いた地域社会構造の変化に関するノンパラメトリック多変量解析.を参照。1993;18(1):117-143.
Google ScholarCrossrefWorldCat
Clarke RK, Gorley RN. PRIMER v7: ユーザーマニュアル/チュートリアル, 1st edn. Devon (UK): PRIMER-E Ltd; 2015.
Google ScholarGoogle PreviewWorldCatCOPAC
Colson P, Fancello L, Gimenez G, Armougom F, Desnues C, Fournous G, Yoosuf N,illion M, La Scola B, Raoult D, et al. ヒトにおけるメガビロームの証拠。J Clin Virol. 2013;57(3):191-200.
Google ScholarCrossrefPubMedWorldCat
ミミウイルス:アメーバの巨大ウイルスの発見を先導する。Nat Rev Microbiol. 2017;15(4):243-254.
Google ScholarCrossrefPubMedWorldCat
Conceição-Neto N, Zeller M, Lefrere H, De Bruyn P, Beller L, Deboutte W, Yinda CK, Lavigne R, Maes P, Van Ranst M, et al. Modular approach to customize sample preparation procedures for viral metagenomics: a reproducible protocol for virome analysis.ウイルスメタゲノミクスに向けたサンプル調製手順をカスタマイズするモジュール式アプローチ。Sci Rep. 2015;5:16532.
Google ScholarCrossrefPubMedWorldCat
David LA, Materna AC, Friedman J, Campos-Baptista MI, Blackburn MC, Perrotta A, Erdman SE, Alm EJ. 宿主のライフスタイルは、1日のタイムスケールでヒトの微生物叢に影響を与える。Genome Biol. 2014;15(7):R89.
Google ScholarCrossrefPubMedWorldCat
Dethlefsen L、Huse S、Sogin ML、Relman DA. ヒト腸内細菌叢に対する抗生物質の広範な影響、深い16S rRNA配列決定によって明らかにされた。PLoS Biol.
Google ScholarCrossrefPubMedWorldCat
腸内フローラのホメオスタシスにおける活性化誘導型シチジンデアミナーゼの重要性。Science. 2002;298(5597):1424–1427.
Google ScholarCrossrefPubMedWorldCat
Frank DN, St Amand AL, Feldman RA, Boedeker EC, Harpaz N, Pace NR. ヒト炎症性腸疾患における微生物群集の不均衡の分子・系統学的特徴づけ。Proc Natl Acad Sci USA. 2007;104(34):13780–13785.
Google ScholarCrossrefPubMedWorldCat
Garrett WS, Lord GM, Punit S, Lugo-Villarino G, Mazmanian SK, Ito S, Glickman JN, Glimcher LH.の各氏。自然免疫系におけるT-bet欠損により誘発される感染性潰瘍性大腸炎。Cell. 2007;131(1):33-45.
Google ScholarCrossrefPubMedWorldCat
Gevers D, Kugathasan S, Denson LA, Vázquez-Baeza Y, Van Treuren W, Ren B, Schwager E, Knights D, Song SJ, Yassour M, et al. 新規発症のクローン病における治療不要なマイクロバイオーム(The treatment-naive microbiome in new-set Crohn's disease. Cell Host Microbe. 2014;15(3):382-392.
Google ScholarCrossrefPubMedWorldCat
Girardin SE、Boneca IG、Viala J、Chamaillard M、Labigne A、Thomas G、Philpott DJ、Sansonetti PJ. Nod2は、ムラミルジペプチド(MDP)検出によるペプチドグリカンの一般的なセンサーである。J Biol Chem. 2003;278(11):8869–8872. https://doi.org/10.1074/jbc.C200651200.
Google ScholarCrossrefPubMedWorldCat
Guo J, Bolduc B, Zayed AA, Varsani A, Dominguez-Huerta G, Delmont TO, Pratama AA, Gazitua MC, Vik D, Sullivan MB, et al. VirSorter2: a multi-classifier, expert-guided approach to detect diverse DNA and RNA viruses.(多様なDNAおよびRNAウイルスを検出するマルチクラシファイア、エキスパートガイドアプローチ)。BMC: マイクロバイオーム。2021;9(1):37.
Google ScholarWorldCat
ハンドリーSA. The virome: a missing component of biological interaction networks in health and disease(バイローム:健康と病気における生物学的相互作用ネットワークの欠落した構成要素)。Genome Med. 2016;8(1):32.
Google ScholarCrossrefPubMedWorldCat
Heinsen FA, Knecht H, Neulinger SC, Schmitz RA, Knecht C, Kühbacher T, Rosenstiel PC, Schreiber S, Friedrichs AK, Ott SJ.の項参照。パロモマイシンによる抗生物質治療中および治療後の管腔内および粘膜関連腸内細菌叢の動的な変化。Gut Microbes. 2015;6(4):243-254.
Google ScholarCrossrefPubMedWorldCat
Hugot JP, Chamaillard M, Zouali H, Lesage S, Cézard JP, Belaiche J, Almer S, Tysk C, O'Morain CA, Gassull M, et al. NOD2 leucine-rich repeat variantsとクローン病感受性の関連性. Nature. 2001;411(6837):599–603. https://doi.org/10.1038/35079107.
Google ScholarCrossrefPubMedWorldCat
猪原直樹、小倉優子、Fontalba A、Gutierrez O、Pons F、Crespo J、深瀬和彦、稲村周平、楠本聡、橋本正樹、他。 NOD2を介した細菌性ムラミルジペプチドの宿主認識。クローン病への影響。J Biol Chem. 2003;278(8):5509–5512. https://doi.org/10.1074/jbc.C200673200.
Google ScholarCrossrefPubMedWorldCat
腸内細菌は、常在菌の有益な機能を代替することができる。Nature. 2014;516(7529):94–98.
Google ScholarCrossrefPubMedWorldCat
Knights D, Silverberg MS, Weersma RK, Gevers D, Dijkstra G, Huang H, Tyler AD, van Sommeren S, Imhann F, Stempak JM, et al. 複雑なホスト遺伝学が炎症性腸疾患におけるマイクロバイオームに影響を及ぼしています。Genome Med. 2014;6(12):107.
Google ScholarCrossrefPubMedWorldCat
Kobayashi KS, Chamaillard M, Ogura Y, Henegariu O, Inohara N, Nuñez G, Flavell RA. Nod2による腸管での自然免疫と適応免疫の制御。Science. 2005;307(5710):731–734. https://doi.org/10.1126/science.1104911.
Google ScholarCrossrefPubMedWorldCat
ラングミードB、サルツバーグSL. Bowtie 2による高速ギャップリード・アライメント。Nat Methods. 2012;9(4):357-359.
Google ScholarCrossrefPubMedWorldCat
この論文では、「ゲノム解読のためのデータ処理」をテーマとしています。このような場合、「Sequence Alignment/Map format and SAMtools」(配列アラインメント/マップ・フォーマットとSAMツール)を使用します。バイオインフォマティクス. 2009;25(16):2078–2079.
Google ScholarCrossrefPubMedWorldCat
DUOX2由来の活性酸素種は、NOD2が介在する抗菌反応のエフェクターである。J Cell Sci. 2009;122(Pt 19):3522-3530. https://doi.org/10.1242/jcs.050690.
Google ScholarPubMedWorldCat
受容体相互作用型プロテインキナーゼ2が介在するマイトファジーは、ウイルス感染時のインフラマソーム活性化を制御する。Nat Immunol. 2013;14(5):480-488.
Google ScholarCrossrefPubMedWorldCat
Manichanh C, Rigottier-Gois L, Bonnaud E, Gloux K, Pelletier E, Frangeul L, Nalin R, Jarrin C, Chardon P, Marteau P, et al. クローン病における糞便微生物相の減少がメタゲノム手法により明らかにされた。Gut. 2006;55(2):205-211.
Google ScholarCrossrefPubMedWorldCat
McArdle BH, Anderson MJ. 多変量モデルの群集データへの当てはめ:距離ベースの冗長性分析に関するコメント。Ecology. 2001;82(1):290-297.
Google ScholarCrossrefWorldCat
Minot S, Bryson A, Chehoud C, Wu GD, Lewis JD, Bushman FD. ヒト腸内細菌群の急速な進化。Proc Natl Acad Sci USA. 2013;110(30):12450–12455.
Google ScholarCrossrefPubMedWorldCat
Minot S, Sinha R, Chen J, Li H, Keilbaugh SA, Wu GD, Lewis JD, Bushman FD. ヒトの腸内ビローム:個人間変動と食事への動的応答。ゲノム研究 2011;21(10):1616-1625.
Google ScholarCrossrefPubMedWorldCat
Mirzaei MK, Maurice CF. ヒト腸管におけるメナジ・ア・トロワ:宿主、細菌、ファージ間の相互作用。Nat Rev Microbiol. 2017;15(7):397-408.
Google ScholarCrossrefPubMedWorldCat
Moltzau Anderson J, Lipinski S, Sommer F, Pan W-H, Boulard O, Rehman A, Falk-Paulsen M, Stengel ST, Aden K, Häsler R, et al. NOD2 influences trajectories of intestinal microbiota recovery after antibiotic perturbation.腸内細菌の回復に影響を及ぼすNOD2。Cell Mol Gastroenterol Hepatol. 2020;10(2):365-389.
Google ScholarCrossrefPubMedWorldCat
Norman JM, Handley SA, Baldridge MT, Droit L, Liu CY, Keller BC, Kambal A, Monaco CL, Zhao G, Fleshner P, et al. 炎症性腸疾患における腸内ビロームの疾患特異的変化(Disease Specific changes in the enteric virome in inflammatory bowel disease).Cell.Mol.Gastenterol.Hepol.Co. Cell. 2015;160(3):447-460.
Google ScholarCrossrefPubMedWorldCat
小椋佳、Bonen DK、Inohara N、Nicolae DL、Chen FF、Ramos R、Britton H、Moran T、Karaliuskas R、Duerr RH、他。 クローン病感受性に関連するNOD2におけるフレームシフト突然変異。Nature. 2001;411(6837):603–606. https://doi.org/10.1038/35079114.
Google ScholarCrossrefPubMedWorldCat
Ott SJ, Musfeldt M, Wenderoth DF, Hampe J, Brant O, Fölsch UR, Timmis KN, Schreiber S. Reduction in diversity of the colonic mucosa associated bacterial microflora in patients with active inflammatory bowel disease.(オットSJ、ムスフェルトM、ウェンデルートDF、ハンペJ、ブラントO、フェルシュUR、ティミスKN、シュライバーS.)。Gut. 2004;53(5):685-693.
Google ScholarCrossrefPubMedWorldCat
巨大ウイルスを中心としたサイレントヒトビロームの記述。Intervirology. 2013;56(6):395-412.
Google ScholarCrossrefPubMedWorldCat
Pruitt KD, Tatusova T, Brown GR, Maglott DR. NCBI Reference Sequences (RefSeq): current status, new features and genome annotation policy.NCBI参照配列(RefSeq)の現状、新機能、ゲノムアノテーションの方針。Nucleic Acids Res. 2012;40(Database Issue):D130-D135.
Google ScholarPubMedWorldCat
腸内細菌叢のToll様受容体による認識は、腸のホメオスタシスに必要である。Cell. 2004;118(2):229-241.
Google ScholarCrossrefPubMedWorldCat
Reyes A, Haynes M, Hanson N, Angly FE, Heath AC, Rohwer F, Gordon JI. 一卵性双生児とその母親の糞便微生物叢に含まれるウイルス。Nature. 2010;466(7304):334–338.
Google ScholarCrossrefPubMedWorldCat
Reyes A, Semenkovich NP, Whiteson K, Rohwer F, Gordon JI. 次世代シーケンサーによるヒト腸内ファージ集団の解析。Nat Rev Microbiol. 2012;10(9):607-617.
Google ScholarCrossrefPubMedWorldCat
この論文では、ファージによる捕食を通して微生物集団のゲノム情報を説明します。Nat Rev Microbiol. 2009;7(11):828-836.
Google ScholarCrossrefPubMedWorldCat
Rooks MG, Veiga P, Wardwell-Scott LH, Tickle T, Segata N, Michaud M, Gallini CA, Beal C, van Hylckama-Vlieg JE, Ballal SA, et al. Gut microbiome composition and function in experimental colitis during active disease and treatment-induced remission.実験大腸炎における腸内細菌の組成と機能。Isme J. 2014;8(7):1403-1417.
Google ScholarCrossrefPubMedWorldCat
Rosenstiel P, Sina C, End C, Renner M, Lyer S, Till A, Hellmig S, Nikolaus S, Fölsch UR, Helmke B, et al. 腸管上皮細胞におけるNOD2およびTLR4を介してDMBT1の制御は、細菌の認識および侵入を調節している。J Immunol. 2007;178(12):8203–8211. https://doi.org/10.4049/jimmunol.178.12.8203.
Google ScholarCrossrefPubMedWorldCat
Roux S, Solonenko NE, Dang VT, Poulos BT, Schwenck SM, Goldsmith DB, Coleman ML, Breitbart M, Sullivan MB.(ルー S、ソロネンコ NE、ダン VT、プーロス BT、シュウェンク SM、ゴールドスミス DB、コールマン ML、ブライトバート M、サリバン MB)。二本鎖および一本鎖DNAウイルスの定量的ウイルス学に向けて。PeerJ. 2016;4:e2777.
Google ScholarCrossrefPubMedWorldCat
Sabbah A, Chang TH, Harnack R, Frohlich V, Tominaga K, Dube PH, Xiang Y, Bose S. Activation of innate immune antiviral responses by Nod2(Nod2による自然免疫抗ウイルス反応の活性化)。Nat Immunol. 2009;10(10):1073–1080.
Google ScholarCrossrefPubMedWorldCat
Schofield D, Sharp NJ, Westwater C. Phage-based platforms for the clinical detection of human bacterial pathogen(ファージを用いたヒト細菌病原体の臨床検出プラットフォーム)。Bacteriophage. 2012;2(2):105-283.
Google ScholarCrossrefPubMedWorldCat
Sell TL, Schaberg DR, Fekety FR. クロストリジウム・ディフィシル菌のバクテリオファージおよびバクテリオシンのタイピングスキーム(Bacteriophage and bacteriocin typing scheme for Clostridium difficile. J Clin Microbiol. 1983;17(6):1148–1152.
Google ScholarCrossrefPubMedWorldCat
この論文では、「生物分子間相互作用ネットワークの統合的モデルのためのソフトウェア環境」であるCytoscapeを紹介し、その中で「生物分子間相互作用ネットワークの統合的モデルのためのソフトウェア環境」を紹介しています。ゲノム研究会 2003;13(11):2498-2504.
Google ScholarCrossrefPubMedWorldCat
Sharp PM. ヒトウイルスの多様性の起源。Cell. 2002;108(3):305-312.
Google ScholarCrossrefPubMedWorldCat
Sommer F, Moltzau Anderson J, Bharti R, Raes J, Rosenstiel P. The resilience of the intestinal microbiota influences health and disease(腸内細菌叢の回復力は健康と病気に影響を及ぼす。Nat Rev Microbiol. 2017;15(10):630-638.
Google ScholarCrossrefPubMedWorldCat
Vijay-Kumar M、Aitken JD、Carvalho FA、Cullender TC、Mwangi S、Srinivasan S、Sitaraman SV、Knight R、Ley RE、Gewirtz AT. Toll様受容体5を欠損したマウスにおけるメタボリックシンドロームと腸内細菌叢の変化。Science. 2010;328(5975):228–231.
Google ScholarCrossrefPubMedWorldCat
Von Meijenfeldt FAB, Arkhipova K, Cambuy DD, Coutinho FH, Dutilh BE. CATとBATを用いた未知の微生物配列とビンのロバストな分類法。Genome Biol. 2019;20:217.
Google ScholarCrossrefPubMedWorldCat
Wang W, Ren J, Tang K, Dart E, Ignacio-Espinoza JC, Fuhrman JA, Braun J, Sun F, Ahlgren NA.(ワン・ワン、レン・ジェイ、タン・ケイ、ダート・イー、イグナシオ・エスピノザJC、ファールマンJA、ブラウンJ、サンF、アルグレンNA ウイルスと原核生物の相互作用を予測するためのネットワークベースの統合フレームワーク。NAR Genomics Bioinformatics. 2020;2(2):
Google ScholarWorldCat
Wehkamp J, Harder J, Weichenthal M, Schwab M, Schäffeler E, Schlee M, Herrlinger KR, Stallmach A, Noack F, Fritz P, et al. クローン病のNOD2(CARD15)変異は粘膜αディフェンシン発現低下と関連している。Gut. 2004;53(11):1658–1664. https://doi.org/10.1136/gut.2003.032805.
Google ScholarCrossrefPubMedWorldCat
双子におけるパイロシークエンス研究は、胃腸の微生物プロファイルが炎症性腸疾患の表現型によって異なることを示す。Gastroenterology. 2010;139(6):1844–1854.e1.
Google ScholarCrossrefPubMedWorldCat
この記事が気に入ったらサポートをしてみませんか?
