
動物の単細胞祖先に至る血球の進化史の追跡
貪食細胞、顆粒球、骨髄形成|2022年12月15日
動物の単細胞祖先に至る血球の進化史の追跡
長畑洋介、増田恭子、西村雄二、井川智克、川岡慎平、北脇俊雄、難波康人、小川誠司、菅浩志、佐藤豊、高折昭文、川本浩史
クロスマーク 更新情報を確認する
ブラッド(2022)140(24)。2611-2625.
https://doi.org/10.1182/blood.2022016286
記事履歴
関連する内容
に関連する記事です。最初の血球はいつ、どのように進化したのか?
スプリットスクリーン
シェアアイコン
共有
ツールアイコン
ツール
PDF






キーポイント
動物の共通祖先は、単細胞生物から貪食プログラムを受け継ぎ、最初の血球が出現した。
マウス造血系では、CEBPαがポリコーム複合体によって抑制され、非食細胞系が維持されている。
ビジュアルアブストラクト
グラフィック
拡大表示する スライドをダウンロードする
要旨
血球は、動物の共通祖先において食細胞として出現し、その後、進化の過程で血小板、赤血球、リンパ球などの新しい血球系譜が出現したと考えられている。しかし、この推測は遺伝学的証拠に基づくものではなく、異なる種の食細胞は異なる起源を持つという主張も可能である。また、初期の血球がどのように進化したのか、古代の動物が食細胞のプログラムを単独でデノボに開発したのか、それとも祖先の単細胞生物から重要なプログラムを受け継いだのか、明らかにされる必要がある。そこで、血球の進化史をたどり、遺伝子発現プロファイルを種間で比較したところ、様々な動物種の食細胞や単細胞生物であるCapsaspora (C. owczarzaki) の食細胞は転写的に互いに類似していることが明らかになりました。また、食細胞とC. owczarzakiは共通の食作用プログラムを持ち、CEBPαは食細胞とC. owczarzakiの両方で高発現している唯一の転写因子であることを明らかにしました。さらに、CEBPαが非食細胞である血液細胞の食細胞プログラムを駆動する機能は、ツチノコ、カイメン、C. owczarzakiで保存されていることを明らかにした。さらに、マウスの造血系において、非食細胞系を維持するためのCEBPαの抑制は、ポリコーム複合体によって共通して達成されることを明らかにした。これらの知見は、最初の血液細胞はCEBPαによって駆動される単細胞生物プログラムを継承して出現し、そのプログラムは進化の過程で様々な動物種の食細胞にもシームレスに継承されてきたことを示唆している。
対象:造血幹細胞、食細胞、顆粒球、骨髄造血
はじめに
赤血球やリンパ球など様々な系統の血液細胞の中で、マクロファージや好中球などの食細胞は、海綿のような形態的には非常に単純な多細胞生物1 を含むあらゆる動物で見られる一方、より複雑な動物ではより多くの系統のタイプが見られることから、進化的に最も古い血液細胞であると考えられてきた2-5 。このため、動物の共通祖先において、進化的に最初の血液細胞が食細胞として出現し、進化の過程で、食細胞以外の様々な系統が最初の食細胞から進化してきたと推測されている。この問題に関して、我々は、マウスの造血系において、赤血球系、T細胞系、B細胞系へのプライミングを行う初期前駆細胞に食細胞の産生能が保持されていることを明らかにしてきた6-10。このような知見に基づき、我々は、これらの系統の前駆細胞における食細胞の潜在能力の保持は、これらの系統のそれぞれが祖先の食細胞から進化した系統発生過程の名残であると提唱している2,11。この名残は、他の脊椎動物でも見出されており、サメ、骨魚類、カエルの血小板、赤血球、B細胞は食細胞能を持つ12-14。
ここで一つ注意すべきなのは、すべての食細胞が系統発生上同じ起源を持つという前提で、このような推測が成り立つということである。しかし、このモデルを支持する遺伝学的証拠は不十分であり、異なる動物種の食細胞が異なる起源を持つという収斂進化の可能性を主張することができる。さらに、最初の血球がどのように進化したのかについても、まだ明らかにされていない。一つは、古代の動物が食細胞のプログラムを単独で開発した場合、もう一つは、祖先の単細胞生物から食細胞の重要なプログラムを受け継いだ場合です。
この問題を解決するために、様々な動物種の食細胞に共通のプログラムが存在するのか、また、そのプログラムが単細胞生物にも共有されているのかを明らかにすることにした。そのため、様々な動物種の食細胞、非食細胞、および単細胞生物の遺伝子発現プロファイルを比較した。
方法
マウス
Ert2Cre-Cdkn2a-/-Ring1a-/-Ring1bfl/fl, Ert2Cre-CAGflox-stop-GFP-Cdkn2a-/-Ring1a-/-Ring1bfl/fl および LckCre-Cdkn2a-/-Ring1a-/-Ring1bfl/fl マウスは我々の動物施設において作製・維持されたものである。すべてのマウスは、当社の動物施設において、特定の病原体を含まない状態で維持された。すべての実験は、京都大学動物実験委員会のガイドラインに従って行われ、我々の機関委員会によって承認された。
チュニケーション
Ciona intestinalis (type A; Ciona robustaとも呼ばれる) 成虫をNational BioResource Project for Cionaから入手した。
莢膜藻類(Capsaspora
Capsaspora owczarzakiは、既報の通り、ATCC 1034培地で23℃に維持した15。
データおよびコードの入手
EMBL-EBI に登録されているマウスの公開データ(supplemental Table 1, Blood ウェブサイトで公開)および既報のマウス、ツチノコ、カイメン、C. owczarzaki、Salpingoeca rosetta、Creolimax fragrantissima のデータを解析した15-23。また、ツマグロツメガエル食細胞およびRing1a/bノックアウト(KO)骨髄細胞のRNAシーケンス(RNA-seq)データは、DNA Data Bank of Japanデータベース(DRA013007およびDRA014437)で入手可能である。
異種間トランスクリプトーム比較
Musculus, C. intestinalis, Amphimedon queenslandica, C. owczarzaki のホモログを OrthoFinder で同定した(補足表2)24。4種間で共通して保存されているホモログ群を選択し、種を超えた比較に用いた(補足表3)。S. rosetta と C. fragrantissima を加えた 6 種の異種間解析も行った(補足表 4-5)。
転写因子(TF)および貪食関連遺伝子
TFおよび貪食・リソソーム関連遺伝子の選定には、AmiGO2データベース(http://amigo.geneontology.org/amigo)を使用した(補足表6)。
マウス前駆細胞の単離
胸腺または骨髄(BM)の単細胞懸濁液を調製し、蛍光活性化セルソーティングにより前駆細胞を単離した。ゲーティング方法を補足図1に示す。
CEBPαおよびRing1Bをコードするベクター
CEBPαおよびRing1Bのコドン最適化DNA配列は、GeneArt(Thermo Fisher Scientific)を用いて合成した(補足表7)。
レトロウイルスの作製と導入
CEBPαおよびRing1BをコードするベクターをPlat-E細胞(CosmoBio)にトランスフェクトし、上清を採取した。トランスダクションのため、精製した前駆細胞を上清に再懸濁し、1000×g、32℃で90分間遠心分離を行った。
ファゴサイトーシスアッセイ
pHrodo-greenザイモサンまたはStaphylococcus aureus beads(Invitrogen)を各培養液に添加した。1時間後、培地をリン酸緩衝生理食塩水に交換し、蛍光顕微鏡を用いてファゴサイトーシスを観察した。
RNA抽出とリアルタイム定量ポリメラーゼ連鎖反応
RNeasy kit(Qiagen)を用いてTotal RNAを単離した。相補的DNA合成は、SuperScript IV VILO Master Mix相補的DNA合成キット(Invitrogen社製)を用いて行った。リアルタイムポリメラーゼ連鎖反応は、PowerUp SYBR Green Master Mix (Applied Biosystems) を用いて行い、StepOnePlus (Applied Biosystems) で解析した。
チューニカー食細胞およびRing1a/b KOミエロイド細胞のRNA-seq
SMART-Seq v4 Ultra Low Input RNA Kit for Sequencing (Takara) とNextera XT DNA Library Prep kit (Illumina) を用いてライブラリを作成し、NovaSeq 6000 (Illumina) でシークエンスを行った。
Ring1bのin vitro欠失
分離した前駆細胞をTSt425細胞またはTSt4-DLL126細胞と4〜12日間共培養し、4-hydroxytamoxifen(4-OHT)によりRing1bを欠失させた。
BMキメラマウス
溶血した全BM細胞(2×106個)を、腹腔内照射(4Gy)したRag2-/-マウスに静脈注射した。長期観察用に、1×106個のBM細胞に1×106個のコンペティター細胞を移植した。
統計解析
生存率はKaplan-Meier法を用いて推定し、log-rank検定を用いて比較した。連続変数とカテゴリー変数は、それぞれ両側t検定とフィッシャー正確検定を用いて比較した。
さらなる実験の詳細は、supplemental methodsに記載されている。
結果
マウス、フナクイムシ、海綿の貪食細胞は単細胞生物と転写的に類似している
マウス(M. musculus)、ホヤ(C. intestinalis)、海綿(A. queenslandica)、単細胞生物(以下Capsaspora)の4種について、様々な系統や段階の細胞の遺伝子発現プロファイルを比較しました(図1A)。無脊椎動物では、脊索動物に属し脊椎動物に近いツチノコと、最も古く脊椎動物から遠いカイメンから選択した27,28。単細胞生物のうち、Capsaspora は系統学的に動物に近く、Metazoa と共に Holozoa と呼ばれるクレードを形成することから選択した(図 1A)29-31 まず、4 種間で保存されたホモログを検索し、3237 ホモロググループが特定され、マウスで 5911 遺伝子、tunicate で 4031 遺伝子、capsaspora で 5443 遺伝子、4096 遺伝子が 3237 ホモロググループに割 り当てられた。次に、遺伝子発現プロファイルをホモロググループに基づいて比較した(補足図2A)。予想通り、マウス、ホヤ、海綿、Capsasporaは互いに大きく異なっていた(補足図2B)。血液細胞では、マクロファージが非貪食細胞よりもCapsasporaに類似していた(図1C-D)。また、マクロファージは好中球よりもCapsasporaに類似しており、これは多葉核を持つ好中球が脊椎動物に特有であることと一致している32。 マウスデータセット間のバッチ効果を除くため、マウス細胞の単一データセットを用いてCAGE(cap analysis gene expression)法による比較も実施した(図1C)。RNA-seqとCAGEの両データセットによる解析では、マウス細胞のうちマクロファージ、肝細胞、線維芽細胞、脂肪細胞はCapsasaporaと高い類似性を示した(図1B-C)。肝細胞、線維芽細胞、脂肪細胞は貪食能を持つことが知られているため、33-35 マクロファージとこれら3系統の細胞は食細胞として分類することが可能である。主成分(PC)分析では、マウスとツチノコの食細胞、食細胞能を持つことが知られている海綿体古細菌1、Capsasporaは互いに類似性を示していた(図1D)。
図1.
マウス、ホヤ、海綿の貪食細胞は、単細胞生物と転写的に類似している。(A) マウス、ホヤ、海綿、襟鞭毛虫、Capsaspora、Ichthyosporea、真菌の系統樹。(B-C) マウスの様々な細胞系とCapsasporaのピアソン相関を示すヒートマップ。3237の保存された相同性に基づき、Capsasporaの3つのステージとマウスの30系統(B)または15系統(C)で遺伝子発現プロファイルを比較した。RNA-seq (B) または CAGE 法 (C) で調べたトランスクリプトームデータを解析した。(D) 4種の様々な系統やステージのPC解析。(D) 4種(Capsaspora, sponge, tunicate, and mouse)の様々な系統やステージのPC解析。3237の保存されたホモログの発現量を正規化し、比較した。(E)Capsaspora糸状体ステージまたはマウスマクロファージで高発現している遺伝子の数をマウスESCと比較したベン図。(F) Capsaspora filopodial stageの高発現遺伝子325個のうち、様々なマウス細胞系列で共有される遺伝子の頻度。マクロファージと他の細胞系との差の統計的有意性も示している。(G)マウスESCsと比較して、Capsaspora filopodial stageで高発現した325遺伝子とCapsaspora filopodial stageで低発現した2252遺伝子のうち、食作用関連遺伝子の頻度を示している。マウスマクロファージでマウスESCsより高発現している食作用関連遺伝子とリソソーム関連遺伝子の頻度を示している。マウスESCsと比較してマクロファージで高発現している遺伝子の頻度を赤で、非貪食性血液細胞の頻度を黒で示す。(H) マウス貪食細胞(左)とカプサスポラ(右)の細胞学的性質をWright-Giemsa染色により検討した。(I-J) Capsasporaの貪食活性は、pHrodo-greenビーズを巻き込むことで評価し(I)、貪食細胞の頻度はフローサイトメトリーで評価した(J)。データは2つの独立した実験の代表値である。∗p<.05、*p<.01、**p<.0001。ESCs, embryonic stem cells; PtC, peritoneal cavity.
拡大表示する pptをダウンロード
マウス、ホヤ、海綿の貪食細胞は単細胞生物と転写が類似している。(A)マウス、ホヤ、海綿、襟鞭毛虫、Capsaspora、Ichthyosporea、真菌の系統樹。(B-C) マウスの様々な細胞系とCapsasporaのピアソン相関を示すヒートマップ。3237の保存された相同性に基づき、Capsasporaの3つのステージとマウスの30系統(B)または15系統(C)で遺伝子発現プロファイルを比較した。RNA-seq (B) または CAGE 法 (C) で調べたトランスクリプトームデータを解析した。(D) 4種の様々な系統やステージのPC解析。(D) 4種(Capsaspora, sponge, tunicate, and mouse)の様々な系統やステージのPC解析。3237の保存されたホモログの発現量を正規化し、比較した。(E)Capsaspora糸状体ステージまたはマウスマクロファージで高発現している遺伝子の数をマウスESCと比較したベン図。(F) Capsaspora filopodial stageの高発現遺伝子325個のうち、様々なマウス細胞系列で共有される遺伝子の頻度。マクロファージと他の細胞系との差の統計的有意性も示している。(G)マウスESCsと比較して、Capsaspora filopodial stageで高発現した325遺伝子とCapsaspora filopodial stageで低発現した2252遺伝子のうち、食作用関連遺伝子の頻度を示している。マウスマクロファージでマウスESCsより高発現している食作用関連遺伝子とリソソーム関連遺伝子の頻度を示している。マウスESCsと比較してマクロファージで高発現している遺伝子の頻度を赤で、非貪食性血液細胞の頻度を黒で示す。(H) マウス貪食細胞(左)とカプサスポラ(右)の細胞学的性質をWright-Giemsa染色により検討した。(I-J) Capsasporaの貪食活性は、pHrodo-greenビーズを巻き込むことで評価し(I)、貪食細胞の頻度はフローサイトメトリーで評価した(J)。データは2つの独立した実験の代表値である。∗p<.05、*p<.01、**p<.0001。ESCs, embryonic stem cells; PtC, peritoneal cavity.
次に、Capsasporaとマウスの様々な細胞系がどの程度高発現遺伝子を共有しているかを調べ、ESCsよりも高発現している遺伝子の数を調べました。Capsasporaは325個、マクロファージは545個の遺伝子を高発現し、101個の遺伝子を共有していた(図1E)。マクロファージは他の血球系に比べ、Capsasporaと多くの遺伝子を共有していた(図1Fおよび補足図3-4)。肝細胞もCapsasporaと多くの遺伝子を共有し、非血液細胞ではマクロファージとより多くの遺伝子を共有していた(補足図3-4)。Kyoto Encyclopedia of Genes and Genomesのパスウェイ解析では、Capsaspora、マクロファージ、肝細胞が共有する遺伝子にリソソーム関連遺伝子が含まれていることが示された(補足図5)。AmiGO2データベースを用いたジーンオントロジー解析の結果、Capsasporaで高発現している325個の遺伝子は、低発現の2252個の遺伝子と比較して、ファゴサイトーシス/リソソーム関連遺伝子の頻度が高いことがわかった(図1G)。これらのデータは、Capsasporaとマウスの食細胞の類似性を形成しているのは、食細胞・リソゾーム関連遺伝子であることを示唆していた。実際、Capsaspora細胞は、細胞質にいくつかの空胞を持つマウスマクロファージ様細胞質(図1H)と強固な貪食活性(図1I-J)を示していた。これらのデータから、食細胞の転写プロファイルはCapsasporaと動物の共通祖先から保存されていることが示唆された。
食細胞と単細胞生物はCEBPα駆動の食細胞プログラムを共有している
次に、Capsasporaとマウスマクロファージの遺伝子発現プロファイルを、マウスESCsと非食細胞である血液細胞、Lin-Sca1+ckit+細胞、T細胞、B細胞、巨核球、赤血球と比較検討した。11個の遺伝子はマウスマクロファージとCapsasporaの両方で高発現し(図2A、補足図6A)、これら11個の遺伝子はリソソーム関連であり、これらの遺伝子はファゴソーム/リソソーム経路で食作用に貢献していることが示唆された(図2B、補足図6B)。また、11個の遺伝子のうち9個は肝細胞で高発現していた(補足図6A)。次に、どのTFがCapsasporaとマウスの両方の食細胞で共通して重要な役割を果たしているかを明らかにしようとした。その結果、4種間で62のTFが保存されていることがわかり、それらの発現量を比較した。3237個の保存遺伝子に基づく比較(図1B-C)と同様に、62個の保存TFに基づく比較でも、マウス食細胞はマウス非食細胞よりもCapsasporaに近いことがわかった(図2Cおよび補足図7A-B)。CEBPαは、マウスESCsおよび非食細胞性血液細胞と比較して、Capsasporaとマウスマクロファージの両方で高度に発現している唯一のTFであった(図2D-Fおよび補足図8A)。CEBPαホモログのいくつかの領域、特にDNA結合bZIPドメインは、4種の間で保存されていた(補足図9)。他のTFも4種間で保存されていた(補足図8B)。また、系統樹ではどれが機能的なCEBPαホモログなのか区別できなかったため、別のCEBPホモログであるCEBPγも検討した(補足図10)。しかし、CEBPγの発現量はCapsasporaでは高発現していないことがわかった(補足図8C)。Capsaspora、マクロファージ、肝細胞におけるGATA1-6ホモログの発現レベルは、巨核球や赤血球よりも低く(補足図8D)、EBF1-4のそれはB細胞よりも低い(補足図8E)。GATAとEBFファミリーの発現レベルは、Capsasporaと一部のマウス非造血系細胞で比較的高いことから、これらのTFはCapsasporaとマウス非造血系細胞系列間で保存されたプログラムを決定していると考えられた36-38。PU.1とIRFはマウス骨髄系細胞で重要だが39、40それらのホモログはCapsasporaで検出されていない(補遺図8B)。遺伝子発現量をCapsasporaの3つのステージ間で比較したところ、CEBPαは凝集ステージよりも糸状体ステージや嚢胞ステージでより多く発現していた(図2F)。マウスマクロファージとCapsasporaで高発現している11遺伝子のうち、PLA2G15も糸状体ステージと嚢胞ステージでより多く発現していた(図2F、補足図6A)。PLA2G15はリソソームタンパク質であり、ヒトの食細胞による宿主防御と排出に関与している41,42。これらのデータから、PLA2G15の発現を含むCEBPα駆動の食細胞プログラムが単細胞生物と脊椎動物の間で保存されていることが示唆された。
図2.
貪食細胞と単細胞生物はCEBPα駆動の貪食プログラムを共有している。(A,D) Capsasporaまたはマウスマクロファージの高発現遺伝子(A)とTF(D)の数を、マウスESCsおよび非食細胞性血液細胞と比較したベン図。(B) マウスESCsおよび非貪食性血液細胞と比較してCapsasporaおよびマウスマクロファージで高発現した11遺伝子に関与するKEGGパスウェイのトップ8。(C)4種の様々な系統やステージのPC解析。(C) 4種(Capsaspora, sponge, tunicate, and mouse)の様々な系統やステージのPC解析。62の保存されたTFの発現レベルが比較された。(E) Capsaspora、マウスマクロファージ、マウスESCs、マウス非貪食性血液細胞におけるTFの発現量(zスコア)のスケールヒートマップ。マウスESCsや非貪食性血液細胞よりもCapsasporaやマウスマクロファージで高発現する4つのTFを選択した。発現量は8つの細胞グループ間でスケーリングした。(F) CEBPαホモログおよびPLA2G15ホモログのCapsasporaおよび様々なマウス細胞系譜における発現レベル。データは平均値±平均値の標準誤差である。Capsasporaの3ステージ間の差の統計的有意性を示す、*P < .05, *P < .01. KEGG, Kyoto Encyclopedia of Genes and Genomes.
拡大表示する pptをダウンロード
食細胞と単細胞生物は、CEBPα駆動の食細胞プログラムを共有している。(A,D) Capsasporaまたはマウスマクロファージの高発現遺伝子(A)とTF(D)の数を、マウスESCsおよび非食細胞性血液細胞と比較して示したベン図。(B) マウスESCsおよび非貪食性血液細胞と比較してCapsasporaおよびマウスマクロファージで高発現した11遺伝子に関与するKEGGパスウェイのトップ8。(C)4種の様々な系統やステージのPC解析。(C) 4種(Capsaspora, sponge, tunicate, and mouse)の様々な系統やステージのPC解析。62の保存されたTFの発現レベルが比較された。(E) Capsaspora、マウスマクロファージ、マウスESCs、マウス非貪食性血液細胞におけるTFの発現量(zスコア)のスケールヒートマップ。マウスESCsや非貪食性血液細胞よりもCapsasporaやマウスマクロファージで高発現する4つのTFを選択した。発現量は8つの細胞グループ間でスケーリングした。(F) CEBPαホモログおよびPLA2G15ホモログのCapsasporaおよび様々なマウス細胞系譜における発現レベル。データは平均値±平均値の標準誤差である。Capsasporaの3ステージ間の差の統計的有意性を示す、*P < .05, *P < .01. KEGG, Kyoto Encyclopedia of Genes and Genomes.
また、襟鞭毛虫(S. rosetta)とIchthyosporea(C. fragrantissima)を加えた種間解析も行った。この解析でも、様々な種の食細胞は互いに、また単細胞生物との類似性を示した(補足図11A-C)。マウスの細胞系では、マクロファージと脂肪細胞が単細胞生物と高い類似性を示した(補足図11B-C)。Hgdはマウスマクロファージ、Capsaspora、C. fragrantissimaで高発現していた(補足図11D)。しかし、S. rosettaとC. fragrantissimaはともにCEBPαを持たないため、マウスマクロファージ、Capsaspora、C. fragrantissimaのすべてで高発現しているTFは検出されなかった。CEBPα以外の重要な遺伝子が、これらの細胞の類似性を決定しているのかもしれない。
ツチノコと海綿の食細胞はCEBPαホモログを高度に発現している
次に、CEBPαの発現量が、海綿体とフナムシの食細胞と非食細胞の間で異なるかどうかを検討した。海綿体では、体腔内を循環し貪食能を有するという点で血液細胞と同様の挙動を示すアーキサイトに着目した1。アーキサイトの解析から、CEBPα発現量は貪食関連遺伝子やPLA2G15と正の相関があったが、CEBPγレベルは相関がなかった(図3A)。
図3.
ツチノコと海綿の食細胞はCEBPαホモログを高度に発現している。(A) 海綿体食細胞のlog2 (TPM + 1)値による散布図。x軸は海綿CEBPαとCEBPγを示す。y軸は食細胞関連遺伝子とPLA2G15の総発現量を示す(これらの解析ではCEBPαホモログは食細胞関連遺伝子から除外した)。(B) CEBPα、CEBPγ、食細胞関連遺伝子のtunicateにおけるlog2 (TPM + 1)値による発現量。食細胞のトランスクリプトームデータはRNA-seqで調べ、その他の系統のデータはGhost Database (http://ghost.zool.kyoto-u.ac.jp/cgi-bin/gb2/gbrowse/kh/)から得た発現配列タグ数に基づいている。(C)心臓穿刺によりツチノコの血球を吸引した。採取した血球はpHrodoビーズとインキュベートし、フローサイトメトリーで解析した。(D)血球の大きさ、自家蛍光、飲み込まれたビーズの蛍光からフローサイトメトリーで解析した結果、血球の大きさ、自家蛍光、飲み込まれたビーズの蛍光が一致した。(E) RT-qPCRにより、CEBPα、CEBPγ、PLA2G15の発現量を正規化(Gapdh = 1)して評価した。データは平均値±平均値の標準誤差である。∗p < 0.05, p < 0.001, p < 0.0001.
拡大表示する pptをダウンロード
ツチノコと海綿の食細胞はCEBPαホモログを高度に発現している。(A) 海綿体食細胞のlog2 (TPM + 1)値による散布図。x軸は海綿体CEBPαとCEBPγを示す。y軸は食細胞関連遺伝子とPLA2G15の総発現量を示す(これらの解析ではCEBPαホモログは食細胞関連遺伝子から除外した)。(B) CEBPα、CEBPγ、食細胞関連遺伝子のtunicateにおけるlog2 (TPM + 1)値による発現量。食細胞のトランスクリプトームデータはRNA-seqで調べ、その他の系統のデータはGhost Database (http://ghost.zool.kyoto-u.ac.jp/cgi-bin/gb2/gbrowse/kh/)から得た発現配列タグ数に基づいている。(C)心臓穿刺によりツチノコの血球を吸引した。採取した血球はpHrodoビーズとインキュベートし、フローサイトメトリーで解析した。(D)血球の大きさ、自家蛍光、飲み込まれたビーズの蛍光からフローサイトメトリーで解析した結果、血球の大きさ、自家蛍光、飲み込まれたビーズの蛍光が一致した。(E) RT-qPCRにより、CEBPα、CEBPγ、PLA2G15の発現量を正規化(Gapdh = 1)して評価した。データは平均値±平均値の標準誤差である。∗p < .05、****p < .001、****p < .0001。
また、ホヤの血球とCEBPホモログおよび貪食関連遺伝子の発現についても検討した。CEBPαと貪食関連遺伝子は血球、特に貪食細胞で高発現していたが、CEBPγは高発現していなかった(図3B)。そこで、CEBPαがホヤの様々な血液系細胞で異なる発現をしているかどうかを調べるために、ホヤから血球を採取した(図3C)。この血球を、(1)サイズが小さい(血球)、(2)自家蛍光を発する(モルラ細胞)、(3)飲み込んだビーズが蛍光を発する(食細胞)、(4)これらの特徴が陰性(その他の血球)という特徴に基づいて4分画にソートした(図3D)。その結果、CEBPαとPLA2G15の発現量は、血球の他の系統に比べて食細胞で著しく高いことがわかったが、CEBPγの発現量はそうでないか、あるいはわずかであった(図3E)。これらのデータは、海綿とフナムシの両方において、CEBPαが共通して食細胞プログラムを発揮していることを示しているのかもしれない。
CEBPαの食細胞プログラム駆動機能は単細胞生物から保存されていた
まず、マウスのプロB細胞に、マウス、フナクイムシ、海綿動物、CapsasporaのCEBPαを形質導入した(図4A)。tunicateとspongeのCEBPα、およびマウスのCEBPαは、これらのB前駆細胞をCD11bを発現する細胞に変換したが、CapsasporaのCEBPα、およびspongeとCapsasporaのCEBPγは変換しなかった(図4Bおよび補足図12A)。チュニケートまたは海綿のCEBPαのいずれかによって誘導されたCD11b+細胞の大部分はマクロファージのように見え、効率的な貪食活性を示した(図4C-D)。生成されたCD11b+細胞にはD-J配列のIgH遺伝子が存在し(補足図12B)、プロB細胞由来であることが示された。CapsasporaのCEBPαが食細胞プログラムを駆動する可能性があるかどうかを明らかにするために、我々はさらに他の系統前駆細胞について検討した。MkPs、ErPs、DN3 T細胞前駆細胞について検討した。その結果、CapsasporaのCEBPαはMkPsをCD11b+食細胞に変換したが、CapsasporaのCEBPγは変換しなかった(図4E-Gおよび補足図12C)。また、マウス、tunicate、海綿、CapsasporaのCEBPαはErPをCD11b+細胞に変換することがわかった(図4H、補足図12D)。DN3 T細胞前駆細胞は、マウスと海綿のCEBPαによってCD11b+細胞に変換されたが、tunicateとCapsasporaのホモログによって変換されなかった(図4Iおよび補足図12E)。
図4.
食細胞プログラムを駆動するCEBPαの機能は、単細胞生物から保存されてきた。(A) マウスCEBPαとそのホモログ(tunicate, sponge, Capsaspora由来)をプロB細胞に導入し、4日後にフローサイトメトリーにより解析した。(B、E、H-I)プロB細胞(B)、MkPs(E)、ErPs(H)およびDN3細胞(I)に、マウス、tunicate、spongoma、またはCapsaspora CEBPαを導入し、次に示した系譜マーカーについてフローサイトメトリーにより調査した。データは2〜4回の独立した実験の代表値である。(C,F) 様々なCEBPαホモログをプロB細胞(C)およびMkP(F)に導入して生成したCD11b+細胞を選別し、その細胞学をWright-Giemsa染色(左)により検討した。その貪食活性は、pHrodo-green ビーズを飲み込むことによって評価した(右)。(D,G)プロB細胞(D)とMkP(G)から生成されたCD11b+細胞の貪食活性をフローサイトメトリーで評価した。(J) マウスCEBPαをプロB細胞に導入して生成したリング状または多葉状の核を持つ好中球様細胞のWright-Giemsa染色像。(K)Wright-Giemsa染色による細胞診で評価した細胞型の頻度。マウス、チュニカー、またはスポンジCEBPαを導入した細胞(n=100)を調べた。(L)CEBPα導入2日後のプロB細胞における好中球関連遺伝子の相対的発現。β-アクチンで正規化した2-ΔΔCT値による相対的発現レベル(0日目=1)を示した。データは3反復の平均±標準誤差である。∗∗p<0.01、**p<0.001。DN3, double-negative 3; ErPs, erythroid progenitors; MkPs, megakaryocyte progenitors.
拡大表示する pptをダウンロード
CEBPαの食細胞プログラム駆動機能は、単細胞生物から保存されている。(A) マウスCEBPαとそのホモログ(tunicate, sponge, Capsaspora由来)をプロB細胞に導入し、4日後にフローサイトメトリーにより解析した。(B、E、H-I)プロB細胞(B)、MkPs(E)、ErPs(H)およびDN3細胞(I)に、マウス、tunicate、spongoma、またはCapsaspora CEBPαを導入し、次に示した系譜マーカーについてフローサイトメトリーにより調査した。データは2〜4回の独立した実験の代表値である。(C,F) 様々なCEBPαホモログをプロB細胞(C)およびMkP(F)に導入して生成したCD11b+細胞を選別し、その細胞学をWright-Giemsa染色(左)により検討した。その貪食活性は、pHrodo-green ビーズを飲み込むことによって評価した(右)。(D,G)プロB細胞(D)とMkP(G)から生成されたCD11b+細胞の貪食活性をフローサイトメトリーで評価した。(J) マウスCEBPαをプロB細胞に導入して生成したリング状または多葉状の核を持つ好中球様細胞のWright-Giemsa染色像。(K)Wright-Giemsa染色による細胞診で評価した細胞型の頻度。マウス、チュニカー、またはスポンジCEBPαを導入した細胞(n=100)を調べた。(L)CEBPα導入2日後のプロB細胞における好中球関連遺伝子の相対的発現。β-アクチンで正規化した2-ΔΔCT値による相対的発現レベル(0日目=1)を示した。データは3反復の平均±標準誤差である。∗∗p<0.01、**p<0.001。DN3, double-negative 3; ErPs, erythroid progenitors; MkPs, megakaryocyte progenitors.
拡大表示する pptをダウンロード
CEBPαの食細胞プログラム駆動機能は、単細胞生物から保存されている。(A) マウスCEBPαとそのホモログ(tunicate, sponge, Capsaspora由来)をプロB細胞に導入し、4日後にフローサイトメトリーにより解析した。(B、E、H-I)プロB細胞(B)、MkPs(E)、ErPs(H)およびDN3細胞(I)に、マウス、tunicate、spongoma、またはCapsaspora CEBPαを導入し、次に示した系譜マーカーについてフローサイトメトリーにより調査した。データは2〜4回の独立した実験の代表値である。(C,F) 様々なCEBPαホモログをプロB細胞(C)およびMkP(F)に導入して生成したCD11b+細胞を選別し、その細胞学をWright-Giemsa染色(左)により検討した。その貪食活性は、pHrodo-green ビーズを飲み込むことによって評価した(右)。(D,G)プロB細胞(D)とMkP(G)から生成されたCD11b+細胞の貪食活性をフローサイトメトリーで評価した。(J) マウスCEBPαをプロB細胞に導入して生成したリング状または多葉状の核を持つ好中球様細胞のWright-Giemsa染色像。(K)Wright-Giemsa染色による細胞診で評価した細胞型の頻度。マウス、チュニカー、またはスポンジCEBPαを導入した細胞(n=100)を調べた。(L)CEBPα導入2日後のプロB細胞における好中球関連遺伝子の相対的発現。β-アクチンで正規化した2-ΔΔCT値による相対的発現レベル(0日目=1)を示した。データは3反復の平均±標準誤差である。∗∗p<0.01、**p<0.001。DN3, double-negative 3; ErPs, erythroid progenitors; MkPs, megakaryocyte progenitors.
次に、CEBPαのホモログが機能的にどの程度類似しているかを調べた。CEBPαはマウス好中球の分化に関与することが知られており、実際にマウスCEBPαはプロB細胞をリング状または多葉状の核を持つ好中球様細胞に変換したが、tunicateと海綿体のCEBPαはほとんどそうしなかった(図4J-K)。また、マウスや海綿のCEBPαを導入したプロB細胞は、プロB細胞を同程度に食細胞に変化させ、様々な遺伝子の発現量を比較した(図4B)。Cebpa遺伝子発現の直接的な結果を調べるために、まだCD11bを発現し始めていない2日目に細胞を回収した(補足図12F-G)。スポンジCEBPαはマウスCEBPαと同程度に食細胞関連遺伝子をアップレギュレートしたが、マウスCEBPαは好中球関連遺伝子の発現誘導とB細胞関連遺伝子の抑制においてスポンジCEBPαより優れていた(図4Lおよび補足図12H)。
ポリコームを介したCEBPαの抑制は、マウスの様々な造血系譜の維持に必要である。
マウスの血液細胞において、CEBPαは食細胞、すなわち骨髄系細胞のマスターレギュレーターとして機能しており、非食細胞系前駆細胞を骨髄系細胞に変換する可能性がある43-50ことから、非食細胞系細胞の維持にはCEBPαが厳格に抑制されていなければならないことが示唆される。我々は、CEBPαがどのように非食細胞系細胞で抑制されているかを明らかにすることを試み、主要なエピジェネティックリプレッサーの一つであるポリコーム複合体が食細胞プログラムの抑制に関与していると仮定した51。Ring1Bの発現量は、CEBPαと同様に骨髄系細胞よりも非貪食系細胞で高かった(補足図13A-B)。CEBPαをコードするCebpa遺伝子座は、DN3細胞、プロB細胞、ErPs、MkPsではH3K27me3で強くマークされているが、骨髄系細胞ではマークされていないことがわかった(補足図13C)。一方、PU.1をコードするSpi1遺伝子座はH3K27me3でマークされていないことが分かった(補足図13D)。また、Cebpa遺伝子座ではRing1Bの結合が確認された(補足図13E)。CEBPαがポリコームによって抑制されていることを確認するために、Ert2Cre -Cdkn2a-/-Ring1a-/-Ring1bfl/fl マウスからの各系統の前駆細胞に4-OHTを用いてRing1bを欠失させた(補足図13F)。Ring1a/bを欠損させると、数日以内にすべての系統でCEBPαの発現レベルが著しく上昇した(補足図13G)。これらのデータから、ポリコーム複合体は様々な非食細胞系で共通してCEBPαを抑制していることが示された。
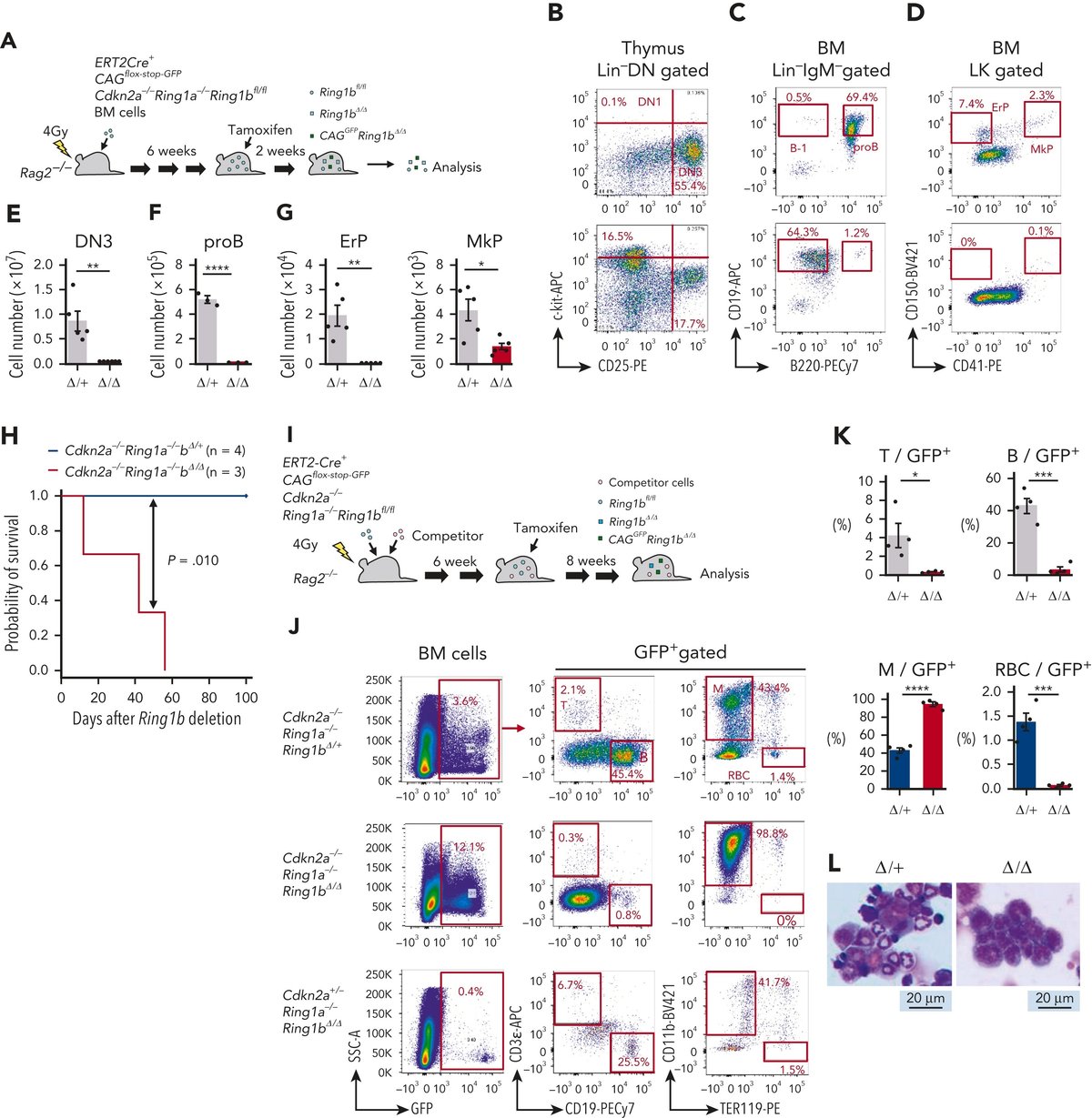
次に、ポリコームが介在するCEBPαの抑制が生理的に重要であるかどうかを検討した。Ert2Cre-CAGflox-stop-GFP-Cdkn2a-/-Ring1a-/-Ring1bfl/flマウスのBM細胞を皮下照射したRag2-/-マウスに移植し、BMキメラマウスを作成した。移植6週間後にタモキシフェン投与によりRing1bを欠失させ、2週間後にマウスを解析した(図5A)。Ring1a/b KO BMキメラマウスでは、緑色蛍光タンパク質陽性(GFP+)画分中の胸腺細胞、二重陽性細胞、DN細胞、DN3細胞の数が減少し、DN1細胞の数が増加した(図5B、E、補足図14A-B、E)。また、プロB細胞の減少とCD19+B220-細胞と定義されるB-1前駆細胞の増加が見られた(図5C,Fおよび補足図14F)。造血幹細胞を含むLin-Sca1+ckit+細胞は減少したが、Lin-Sca1-ckit+細胞は増加した(補足図14C,G)。ErPとMkPの割合は減少し、一般的な骨髄系前駆細胞は増加し、巨核球-赤血球系前駆細胞はそのままであった(図5D、G、補足図14D、H)。BMキメラマウスの造血は著しく損なわれていたため、数ヶ月で死亡した(図5H)。
図5.
ポリコームによるCEBPαの抑制は、マウスの様々な造血系の維持に必要である。 (A,I) ポリコーム機能の条件付き不活性化の実験手順。Ert2Cre-CAGflox-stop-GFP-Cdkn2a-/-Ring1a-/-Ring1bfl/flマウスまたはErt2Cre-CAGflox-stop-GFP-Cdkn2a-/-Ring1a-/-Ring1bfl/+マウスのBM細胞を皮下照射Rag2-/-マウスに静注により競合細胞なし(A)または(I)で移殖させた。6週間後、移植したマウスにタモキシフェンを腹腔内投与し、血球内のRing1bを消失させた。Ring1b欠失の2週間後(A)または8週間後(I)に、マウスを犠牲にし、解析した。(B-D) GFP+胸腺細胞(B)とGFP+BM細胞(C-D)のフローサイトメトリープロファイル。上下のパネルは、それぞれコントロール(Δ/+;Cdkn2a-/Ring1a-/Ring1bΔ/+、パネルBおよびDではn=5、パネルCではn=3)およびRing1a/b KO(Δ/Δ;Cdkn2a-/Ring1a-/Ring1bΔ/Δ、パネルBではn=6、パネルCではn=3、パネルDではn=5)マウスのデータである。(E-G)コントロール(黒)およびRing1a/b KO(赤)マウスのGFP+ DN3細胞(E)、プロB細胞(F)、ErPsおよびMkPs(G)の個体数。(H) 腹腔内照射したRag2-/-マウスのBM移植後の生存曲線とKaplan-Meierプロット。青線はコントロール(Cdkn2a-/-Ring1a-/-Ring1bΔ/+, n = 4)、赤線はRing1a/b KO(Cdkn2a-/-Ring1a-/-Ring1bΔ/Δ, n = 3)マウスの生存曲線をそれぞれ示している。生存率の差の統計的有意性は、log-rank検定で算出した。(J)コントロール(n=4)、Cdkn2a-/-バックグラウンドのRing1a/b KO(n=4)、Cdkn2a+/-バックグラウンドのRing1a/b KO(n=3)マウスの全BM細胞のフローサイトメトリープロファイルと競合細胞との比較を示す。(K) コントロールマウス(青)とRing1a/b KOマウス(赤)のGFP+ BM細胞中の骨髄系細胞、赤血球、T細胞、B細胞の割合と競合細胞。(L) コントロールマウスとRing1a/b KOマウスのBMスミアとコンペティター細胞のWright-Giemsa染色。データは平均値±平均値の標準誤差。∗p < .05、*p < .01、*p < .001、*p < .0001。
拡大表示する pptをダウンロード
ポリコームによるCEBPαの抑制は、マウスの様々な造血系維持に必要である。 (A,I) ポリコーム機能の条件付き不活性化の実験手順。Ert2Cre-CAGflox-stop-GFP-Cdkn2a-/-Ring1a-/-Ring1bfl/flマウスまたはErt2Cre-CAGflox-stop-GFP-Cdkn2a-/-Ring1a-/-Ring1bfl/+マウスのBM細胞を皮下照射Rag2-/-マウスに静注により競合細胞なし(A)または(I)で移殖させた。6週間後、移植したマウスにタモキシフェンを腹腔内投与し、血球内のRing1bを消失させた。Ring1b欠失の2週間後(A)または8週間後(I)に、マウスを犠牲にし、解析した。(B-D) GFP+胸腺細胞(B)とGFP+BM細胞(C-D)のフローサイトメトリープロファイル。上下のパネルは、それぞれコントロール(Δ/+;Cdkn2a-/Ring1a-/Ring1bΔ/+、パネルBおよびDではn=5、パネルCではn=3)およびRing1a/b KO(Δ/Δ;Cdkn2a-/Ring1a-/Ring1bΔ/Δ、パネルBではn=6、パネルCではn=3、パネルDではn=5)マウスのデータである。(E-G)コントロール(黒)およびRing1a/b KO(赤)マウスのGFP+ DN3細胞(E)、プロB細胞(F)、ErPsおよびMkPs(G)の個体数。(H) 腹腔内照射したRag2-/-マウスのBM移植後の生存曲線とKaplan-Meierプロット。青線はコントロール(Cdkn2a-/-Ring1a-/-Ring1bΔ/+, n = 4)、赤線はRing1a/b KO(Cdkn2a-/-Ring1a-/-Ring1bΔ/Δ, n = 3)マウスの生存曲線をそれぞれ示している。生存率の差の統計的有意性は、log-rank検定で算出した。(J)コントロール(n=4)、Cdkn2a-/-バックグラウンドのRing1a/b KO(n=4)、Cdkn2a+/-バックグラウンドのRing1a/b KO(n=3)マウスの全BM細胞のフローサイトメトリープロファイルと競合細胞との比較を示す。(K) コントロールマウス(青)とRing1a/b KOマウス(赤)のGFP+ BM細胞中の骨髄系細胞、赤血球、T細胞、B細胞の割合と競合細胞。(L) コントロールマウスとRing1a/b KOマウスのBMスミアとコンペティター細胞のWright-Giemsa染色。データは平均値±平均値の標準誤差。∗p<.05、*p<.01、*p<.001、*p<.0001。
血液細胞におけるRing1a/b KOの長期的な効果を評価するために、Ring1a/b KO BM細胞と正常な造血に寄与するはずの競合BM細胞との移植を行った(図5I)。Ring1bの欠失から8週間後、ほぼ全てのGFP+Ring1a/b KO細胞はCD11b+骨髄系細胞になった(図5J-K)。さらに、Ring1a/b KOマウスのBMは骨髄系細胞で占められ貧血の様相を呈し、3ヶ月以内に死亡した(図5Lおよび補足図15A-D)。これらのGFP+Ring1a/b KO骨髄系細胞はCD34を発現しており、未熟な芽球のように見えた(補足図15E-F)。胸腺細胞やBM細胞の様々な系譜の前駆細胞が減少しており、Ring1a/b KO骨髄系細胞は白血病芽球に変化して正常な造血を乱していることが示された(補足図15G-L)。次に、Cdkn2a KOを伴わないRing1a/b単独KOが白血病を引き起こすかどうかを検討した。その結果、Cdkn2a+/-Ring1a-/-Ring1bΔ/Δ細胞を持つマウスは白血病を発症せず、GFP+細胞も消失した(図5Jおよび補足図15D)。この結果から、Cdkn2a+/-を残したままRing1a/bをKOするとCdkn2aが過剰発現し、以前T細胞特異的KO例で報告したようにKO細胞のアポトーシスが起こることが示唆された53。
Ring1a/b KOにより様々な系統の前駆細胞が食細胞の始原的な系統に戻された
BMキメラマウスでは、様々な系統の前駆細胞が減少し、骨髄系細胞が増加していることが示された(図5)。次に、各系統の前駆細胞から骨髄系細胞への細胞運命変換が起こっているかどうかを検証した。まず、Ring1a/b KOマウスの骨髄系細胞はIgH遺伝子の再配列を持つが、コントロールマウスのものは再配列を認めなかった(補足図16A)。8匹のRing1a/b KO BMキメラマウスのうち、5匹はIgH遺伝子が再配列された骨髄系細胞を持っていた。これらのデータは、B細胞がin vivoで骨髄系細胞に変換されたことを示している。Ring1a/b KOによって様々な系統の前駆細胞が骨髄系細胞に変換されるかどうかを調べるために、Ert2Cre-Cdkn2a-/-Ring1a-/-Ring1bfl/flマウスのDN3細胞、プロB細胞、ErP、MkPを4-OHTと共にあるいは無しで培養した(図6A)。これらの前駆細胞は既にそれぞれの系統に決定されていたため、コントロール細胞はその系統の同一性を維持していた(図6B)。一方、Ring1bの欠失により、これらの前駆細胞はCD11b+マクロファージ様細胞を生じさせた(図6B-C)。DN3およびpro-B由来の骨髄系細胞は、それぞれV-DJ配列のTCR遺伝子およびIgH遺伝子を有しており、TおよびB系統の前駆細胞に由来することが確認された(補足図16B-C)。また、in vitroでプロB細胞からB-1期を経て骨髄系細胞への系統変換が観察され(補足図16D-E)、BMキメラマウスのB-1細胞数の増加とも一致した(図5C、補足図14F)。我々は以前、LckCreによるRing1a/b KOがT細胞をB細胞に変換することを報告した53。再びLckCre-Cdkn2a-/-Ring1a-/-Ring1bfl/flマウスを解析し、Ring1a/b KO DN3細胞のCD19発現を確認したが、一部はB220発現を欠くB-1表現型だった(補遺図16F)。また、LckCreマウスのDN3細胞は、IgHとTcrb遺伝子が再配列されたB系統の細胞を介して骨髄系細胞に変化した(補足図16G-J)。Ring1b欠失は非食細胞系細胞を食細胞に変換したが、Ring1b過剰発現は食細胞を非食細胞系細胞に変換しなかった(補足図17A-E)。このことは、ポリコーム複合体が非食細胞系細胞の維持に役割を果たすが、非食細胞系細胞の誘導には関与していないことを示すものである。
図6.
Ring1a/b KOにより、様々な系統の前駆細胞が食細胞の始原的な系統に戻されることがわかった。(A) Ert2Cre-Cdkn2a-/-Ring1a-/-Ring1bfl/fl マウスから分離したDN3細胞、プロB細胞、ErPs、MkPsを10 ng/mL の幹細胞因子存在下でTSt4細胞またはTSt4-DLL1細胞と4 OHTあり/なしで4日から12日間共培養した。Flt3-L、インターロイキン1α(IL-1α)、IL-3、IL-7、腫瘍壊死因子α、顆粒球マクロファージコロニー刺激因子の10 ng/mLの存在下で、4-OHTの有無にかかわらず、TSt4またはTSt4-DLL1細胞を共培養した。ErPs と MkPs には、それぞれ 2 U/mL のエリスロポエチンと 50 ng/mL のトロンボポエチンを添加した。(B) 培養した細胞のフローサイトメトリープロファイル。データは3つの独立した実験の代表値である。(C) 生成されたCD11b+細胞の細胞学的性質をWright-Giemsa染色で調べ(左)、その貪食活性をCD11b-BV421染色によるpHrodo-greenビーズで評価した(右)。(D) Capsasporaと様々なマウス細胞系におけるRing1A/Bホモログの発現量。(E) マウスの正常およびRing1a/b KO骨髄系細胞とCapsasporaのピアソン相関を示すヒートマップ。
拡大表示する pptをダウンロード
Ring1a/b KOにより様々な系統の前駆細胞が食細胞の始原的な系統に戻されることがわかった。(A) Ert2Cre-Cdkn2a-/-Ring1a-/-Ring1bfl/flマウスから分離したDN3細胞、プロB細胞、ErP、MkPを幹細胞因子10 ng/mL 存在下でTSt4またはTSt4-DLL1細胞とともに4〜12日間4-OHT併用または非共培養を行った。Flt3-L、インターロイキン1α(IL-1α)、IL-3、IL-7、腫瘍壊死因子α、顆粒球マクロファージコロニー刺激因子の10 ng/mLの存在下で、4-OHTの有無にかかわらず、TSt4またはTSt4-DLL1細胞を共培養した。ErPs と MkPs には、それぞれ 2 U/mL のエリスロポエチンと 50 ng/mL のトロンボポエチンを添加した。(B) 培養した細胞のフローサイトメトリープロファイル。データは3つの独立した実験の代表値である。(C) 生成されたCD11b+細胞の細胞学的性質をWright-Giemsa染色で調べ(左)、その貪食活性をCD11b-BV421染色によるpHrodo-greenビーズで評価した(右)。(D) Capsasporaと様々なマウス細胞系におけるRing1A/Bホモログの発現量。(E) マウスの正常およびRing1a/b KO骨髄系細胞とCapsasporaのピアソン相関を示すヒートマップ。
最後に、Ring1A/Bホモログの発現レベルはCapsasporaでは低く(図6D)、Ring1a/b KO骨髄系細胞は正常骨髄系細胞よりもCapsasporaと類似していることが分かった(図6E)。これらのデータから、Ring1a/b KOはマウス細胞をCapsasporaに近い始原的な状態に戻し、Ring1A/Bが進化の過程で新しい系統を獲得する役割を担っていることが示唆された。
考察
動物は単細胞生物から進化し、29,30,54-57、典型的な糸状虫の特徴を示すことが知られているCapsasporaは、系統学的に動物に近い。我々は、Capsasporaが貪食能を持ち、CEBPαの高発現を特徴とする動物の食細胞に類似した遺伝子発現プロファイルを示すことを明らかにした。さらに、CEBPαのホモログがマウスの非食細胞前駆細胞を食細胞に変換することを示した。
ここで、血球の進化史におけるシナリオを提案する。単細胞の祖先が多細胞生物を形成するようになったとき、上皮に囲まれた体腔構造が形成されたはずである。その際、体腔内をパトロールして病原体や死細胞を貪食して排除するような細胞の祖先が体腔内にいれば、有利になるはずであった。そこで、多細胞生物は、CEBPαが駆動する食作用の祖先プログラムを受け継いで、そのような細胞を保持することに成功した後に生き残り、初期血球の誕生をもたらしたはずである(図7)。
図7.
血球の進化を示す模式図。単細胞生物の表現型が多細胞動物の食細胞として継ぎ目なく受け継がれている。脊椎動物はポリコーム複合体を用いてCEBPαを抑制することにより、様々な系統の血球を獲得してきた。ポリコームが機能しなくなると、造血は貪食細胞のみの原始的なものに戻ってしまう。
拡大表示する pptをダウンロード
血球の進化を示す模式図。単細胞生物の表現型の構成要素が、多細胞動物では食細胞としてシームレスに受け継がれてきた。脊椎動物はポリコーム複合体を用いてCEBPαを抑制することにより、様々な系統の血球を獲得してきた。ポリコームが機能しなくなると、造血は貪食細胞だけの原始的なものに戻された。
その後、動物の進化の過程で、巨核球系、赤血球系、T細胞系、B細胞系が生成されるようになった。ウニが血液凝固機能を持つ血球を持つことは初期の研究で報告されており62、巨核球系は棘皮動物の分岐より早い時期に分離していた可能性がある。巨核球系が造血系分化経路において早期に分岐していることは63、その進化的な早期分離を反映しているはずである。脊索動物では、原索動物のレベルで、血球はいくつかの系統に分別される5,64。この知見に基づき、我々はCEBPαが食細胞系血球に特異的に発現することを明らかにした。脊椎動物の進化史において、顎なし魚と顎あり魚に分岐する前に、赤血球系とリンパ系が生じたはずである。65-67 脊椎動物の造血において、CEBPαは食細胞に特異的に発現しており、本研究から、脊椎動物において非食細胞系を維持するためのCEBPαの抑制がポリコーム複合体によって共通して達成されていることが明らかになった(図7)。Ring1a/b KOがCdkn2a非存在下で白血病発生をもたらすという知見は、さらに、Cdkn2aが確実な造血のために採用され、ポリコーム複合体の機能不全がアポトーシスをもたらすことを示唆している(図5Jおよび補足図15D)。
脊椎動物の造血では、食細胞系の血液系とCEBPαも分岐している。脊椎動物の祖先では、ツナギ類から分離した後、ゲノムの4重化が起こったことが知られており68,69、脊椎動物は4重のCEBPα遺伝子を持つ。CEBPα、CEBPβ、CEBPδ、CEBPεの4つの遺伝子が存在する。このようなCEBPαの4重化により、脊椎動物は様々な貪食性血液細胞を獲得した。例えば、CEBPδとCEBPεは顆粒球に重要である46, 70, 71。脊椎動物の骨髄細胞に必須の他のTF、例えばPU.1やIRFのホモログはカプサスポラで発見されなかった(補足図8B)。これらの遺伝子は単細胞生物から多細胞生物に進化した後に出現し、脊椎動物が樹状細胞などの別の貪食性血液細胞を獲得することを可能にした可能性がある。
さらに、本研究で得られた知見が、祖先の単細胞生物の多細胞化に関して何らかの示唆を与えているかどうかを論じる。食作用は単細胞真核生物では一般的であるが72,73、CEBPのホモログは糸状虫にしか見つかっていない60。先祖の糸状虫がCEBPαを獲得し、シス制御系61とともに食作用のプログラムを制御できるようになったはずである。図2Fおよび図6Dより、CEBPαホモログの発現が低く、Ring1A/Bホモログの発現が高いことから、ポリコーム複合体がCEBPαと食作用の抑制に関与していることが示唆された。ポリコームが介在するCEBPαの抑制が、凝集と多細胞化に寄与してきたと推測したくなる。
注目すべきは、CEBPαが発現していることが知られている肝細胞、線維芽細胞、脂肪細胞が、カプサスポラに類似していることである。これらの細胞は食作用があることが知られているため、これらの細胞もCEBPαが駆動するCapsasporaプログラムを受け継いでいる可能性がある。このようなプログラムが、これらの細胞の進化史の中でシームレスに維持されてきたかどうかを明らかにするためには、さらなる研究が必要である。もう一つの未解決の問題は、プロトストミア血球の進化史である。CEBPαを駆動源とするプログラムがシームレスに継承されてきたのか、それとも別のTFsを駆動源とするプログラムが継承されてきたのか、今後の解明が待たれるところである。
本研究は、動物の祖先が単細胞生物から受け継いだCEBPα駆動の食細胞プログラムを活性化することで初代食細胞が誕生したという動物界における血液細胞の起源を明らかにし、脊椎動物の造血において食細胞プログラムが抑制されて非食細胞系細胞が維持される分子機構、すなわちポリコムを介したCEBPαの後成的抑制を明らかにしたものであり、その結果、動物界の血液細胞がどのように進化してきたかを明らかにしました。
謝辞
Rag2-/-マウスを提供してくださったShimon Sakaguchi(大阪大学)、Ert2CreマウスおよびCdkn2a/-Ring1a/-Ring1bfl/flマウスを提供してくださった小関晴彦(理研)およびMiguel Vidal( Centro de Investigaciones Biologicas)、CAGflox-stop-GFPマウスを提供してくださった宮崎純一(大阪大学)、Ellen V. Rothenberg(Caltech) に謝意を表します。Rothenberg (Caltech) and Hiroyuki Hosokawa (Tokai University) for kindly provided the pMXs-IRES-hNGFR vector; and Peter Burrows (University of Alabama at Birmingham) for critical reading of the manuscript.以上である。
この研究は、日本学術振興会 科学研究費補助金(B)(JP15H04743)および科学研究費補助金 新学術領域研究(JP19H05747)の資金援助を受けて行われたものである。また、LiMe所長研究費2022(No.6)の支援も受けた。
著者名
寄稿。長畑佑樹とH.K.はプロジェクトの構想・設計を、長畑佑樹、K.M.、T.I.、西村佑樹、S.K.はマウスを用いて、長畑佑樹とY.Sはツチノコを用いて、長畑佑樹はカプサスポラを用いて実験方法の設計と最適化を、Y. Nagahata, H.S..., 長畑裕子、H.S.、Y.S.が実験を行い、長畑裕子、H.S.、Y.S.がデータを解析し、T.K.、Y.Nannya、S.O、および A.T.-K が実験を行う上でアドバイスを行い、長畑裕子、 K.M. および H.K が論文を執筆しました。
利益相反の開示:著者らは、競合する金銭的利害関係を申告していない。
通信員。606-8507 京都市左京区聖護院川原町53番地 川本浩司; e-mail: kawamoto@infront.kyoto-u.ac.jp.
参考文献
1.
密度勾配遠心法とフローサイトメトリーにより分離したインド淡水海綿(Eunapius carteri)細胞の食作用効率と細胞毒性反応:形態・機能解析(Mukherjee S, Ray M, Ray S.). Zoology (Jena). 2015;118(1):8-18.
Google ScholarCrossref PubMed
2.
川本英俊、井川智、増田耕一、和田浩、桂由美:造血前駆細胞の系統制限マップ:ミエロイドベースモデルの本質. Immunol Rev. 2010;238(1):23-36.
Google ScholarCrossref PubMed
3.
Cooper MD, Alder MN. 適応免疫系の進化。Cell. 2006;124(4):815-822.
Google ScholarCrossref PubMed
4.
Boehm T. 脊椎動物の免疫の進化。2012;22(17):R722-732.
Google ScholarCrossref PubMed
5.
哺乳類に似た複雑な造血系を脊索動物で発見。Nature. 2018;564(7736):425-429.
Google ScholarCrossref PubMed
6.
Katsura Y, Kawamoto H. Stepwise lineage restriction of progenitors in lympho-myelopoiesis(リンパ骨髄形成における前駆細胞の段階的系統制限). 桂由美, 川本浩司. 2001;20(1):1-20.
Google ScholarCrossref PubMed
7.
マウス胎児肝臓における造血幹細胞のT、Bおよび骨髄系へのコミットメントの直接的証拠。Int Immunol. 1997;9(7):1011-1019.
Google ScholarCrossref PubMed
8.
骨髄リンパ球系前駆細胞:T細胞およびB細胞を生成する造血の重要な中間段階。J Immunol. 2002;169(7):3519-3525.
Google ScholarCrossref
9.
TCR遺伝子再配列の開始に先立ってT細胞系譜が決定される。J Immunol. 2007;179(6):3699-3706.
Google ScholarCrossref
10.
成体T細胞前駆細胞は骨髄系の潜在能力を保持している。Nature. 2008;452(7188):768-772.
Google ScholarCrossref PubMed
11.
リンパ系とミエロイド系の発生上の密接な関係。川本浩司 2006;27(4):169-175.
Google ScholarCrossref PubMed
12.
哺乳類初期のBリンパ球は、強力な貪食能と殺微生物能を持っている。Nat Immunol. 2006;7(10):1116-1124.
Google ScholarCrossref PubMed
13.
血小板による貪食は、下等脊椎動物において保存された自然免疫機構である。Front Immunol. 2014;5:445.
Google ScholarCrossref PubMed
14.
ストークス EE、ファーキン BG. ポートジャクソンシャーク(Heterodontus portusjacksoni)の末梢血の研究、特に血小板について。Br J Haematol. 1971;20(4):427-435.
Google ScholarCrossref PubMed
15.
Sebe-Pedros A, Irimia M, Del Campo J, et al. Regulated aggregative multicellity in a close unicellular relative of metazoa(メタゾアに近い単細胞生物における制御された集合的多細胞性)。Elife. 2013;2:e01287.
Google ScholarCrossref PubMed
16.
プロモーターレベルの哺乳類発現アトラス。Nature. 2014;507(7493):462-470.
Google ScholarPubMed
17.
Choi J, Baldwin TM, Wong M, et al. Haemopedia RNA-seq: a database of gene expression during haematopoiesis in mice and humans(マウスとヒトの造血過程における遺伝子発現データベース)。Nucleic Acids Res. 2019;47(D1):D780-D785.
Google ScholarCrossref PubMed
18.
今井KS、日野K、八木K、佐藤N、佐藤Y. ホヤ胚における転写因子とシグナル伝達分子の遺伝子発現プロファイル:遺伝子ネットワークの包括的理解に向けて。開発. 2004;131(16):4047-4058.
Google ScholarCrossref PubMed
19.
ホヤの統合データベース:機能ゲノミクスに向けて. また、このデータベースは、「ゲノム情報」とも呼ばれている。
Google ScholarCrossref PubMed
20.
菅浩志, 陳瑞禎, 趙亮, 他:Capsaspora ゲノムから単細胞動物の複雑な先史時代が明らかになった。Nat Commun. 2013;4:2325.
Google ScholarCrossref PubMed
21.
曽我部聡、Hatleberg WL、Kocot KM、他:多能性と動物の多細胞化の起源。Nature. 2019;570(7762):519-522.
Google ScholarCrossref PubMed
22.
de Mendoza A, Suga H, Permanyer J, Irimia M, Ruiz-Trillo I.(デ・メンドーサ、スガ、ペルマニア、イリミア、ルイズ・トリロ)。動物の近縁種における複雑な転写制御と真菌様形質の独立進化。Elife. 2015;4:e08904.
Google ScholarCrossref PubMed
23.
Fairclough SR, Chen Z, Kramer E, et al. チョアン鞭毛虫Salpingoeca rosettaにおける前メタゾアゲノム進化と細胞分化の制御. ゲノムバイオロジー2013;14(2):R15.
Google ScholarCrossref PubMed
24.
Emms DM, Kelly S. OrthoFinder: Phylogenetic Orthology inference for comparative genomics. Genome Biol. 2019;20(1):238.
Google ScholarCrossref PubMed
25.
Ohmura K, Kawamoto H, Fujimoto S, Ozaki S, Nakao K, Katsura Y. Emergence of T, B, and myeloid lineage-committed as well as multipotent hemopoietic progenitors in the aorta-gonad-mesonephros region of the day 10 fetuses of the mouse. J Immunol. 1999;163(9):4788-4795.
Google Scholar
26.
増田和彦、久場川博史、井川高志、他:ペアの免疫グロブリン様受容体の発現により定義される前胸腺T細胞の発生。升田和彦、窪川秀樹、井川高志、他。
Google ScholarCrossref PubMed
27.
Turner EC. 新原生代初期の微生物礁に含まれる針葉樹体の化石の可能性。Nature. 2021;596(7870):87-91.
Google ScholarCrossref PubMed
28.
このような場合、「カンブリア紀の難問:新第三紀紀の微生物礁の化石」と呼ばれる。カンブリア紀の難問:動物の初期史における早期の分岐と後期の生態学的成功。サイエンス. 2011;334(6059):1091-1097.
Google ScholarCrossref PubMed
29.
また、このような環境下において、「環境問題」を解決するためには、「環境問題」を解決することが重要である。を参照。2008;3(5):e2098.
Google ScholarCrossref PubMed
30.
Google ScholarCrossref PubMed
31.
Torruella G, de Mendoza A, Grau-Bove X, et al. Phylogenomics reveals convergent evolution of lifestyles in close relatives of animals and fungi. Curr Biol. 2015;25(18):2404-2410.
Google ScholarCrossref PubMed
32.
Hartenstein V. Blood cells and blood cell development in the animal kingdom. Annu Rev Cell Dev Biol. 2006;22:677-712.
Google ScholarCrossref PubMed
33.
曽爾俊之、村田靖、大平晃、西園英治、田中光輝、ハーバートDC. 肝細胞が外来物質を貪食する証拠。Anat Rec. 1992;233(4):543-546.
Google ScholarCrossref PubMed
34.
脂肪組織は、その局在によって貪食および殺微生物活性に差がある。Int J Obes Relat Metab Disord. 2001;25(9):1275-1280.
Google ScholarCrossref PubMed
35.
Romana-Souza B, Chen L, Leonardo TR, Chen Z, DiPietro LA. 皮膚線維芽細胞によるアポトーシス細胞の貪食:創傷治癒のための新規パスウェイ。FASEB J. 2021;35(4):e21443.
Google ScholarCrossref PubMed
36.
モルケンチンJD、カルヴァコラヌDV、マーカムBE. 転写因子GATA-4は、α-ミオシン重鎖遺伝子の心筋特異的な発現を制御する。このように、心筋の遺伝子発現を制御する転写因子GATA-4は、心筋に特異的に発現している。
Google ScholarPubMed
37.
Milatovich A, Qiu RG, Grosschedl R, Francke U. 初期Bリンパ球、脂肪細胞、嗅覚神経細胞に発現する組織特異的転写活性化因子(EBFまたはOlf-1)遺伝子は、ヒト第5染色体バンドq34およびマウス第11染色体近傍に存在する。Mamm Genome. 1994;5(4):211-215.
Google ScholarCrossref PubMed
38.
このような場合、「臓器別」、「細胞別」、「組織別」の3つに分類することができます。2021;24(6):102551.
Google ScholarCrossref PubMed
39.
Nerlov C, Graf T. PU.1 は、多能性造血前駆細胞において骨髄系へのコミットメントを誘導する。Genes Dev. 1998;12(15):2403-2412.
Google ScholarCrossref PubMed
40.
ICSBP/IRF-8レトロウイルス導入はin vitroで樹状細胞の発生を回復させる。Blood. 2003;101(3):961-969.
ArticleGoogle ScholarCrossref PubMed
41.
Shayman JA, Tesmer JJG. ライソゾームホスホリパーゼA2。Biochim Biophys Acta Mol Cell Biol Lipids. 2019;1864(6):932-940.
Google ScholarCrossref PubMed
42.
Mota AC, Dominguez M, Weigert A, Snodgrass RG, Namgaladze D, Brune B. Lysosome-dependent LXR and PPARdelta activation upon efferocytosis in human macrophage. Front Immunol. 2021;12:637778.
Google ScholarCrossref PubMed
43.
B細胞からマクロファージへの段階的なリプログラミング。Cell. 2004;117(5):663-676.
Google ScholarCrossref PubMed
44.
Laiosa CV, Stadtfeld M, Xie H, de Andres-Aguayo L, Graf T. C/EBPαおよびPU.1転写因子によるT細胞前駆細胞からマクロファージおよび樹状細胞へのリプログラミング。Immunity. 2006;25(5):731-744.
Google ScholarCrossref PubMed
45.
Collombet S, van Oevelen C, Sardina Ortega JL, et al. lymphoid and myeloid cell specification and transdifferentiation のロジカルモデリング。Proc Natl Acad Sci U S A. 2017;114(23):5792-5799。
Google ScholarCrossref PubMed
46.
Cirovic B, Schonheit J, Kowenz-Leutz E, et al. C/EBP-induced transdifferentiation reveals granulocyte-macrophage precursor-like plasticity of B cells.(C/EBPによる分化転換は、B細胞の顆粒球マクロファージ前駆体様可塑性を明らかにする). Stem Cell Rep. 2017;8(2):346-359.
Google ScholarCrossref
47.
Suh HC, Gooya J, Renn K, Friedman AD, Johnson PF, Keller JR. C/EBPalphaは、赤血球分化を阻害し、骨髄系分化を誘導することにより、多能性前駆細胞における造血系細胞の運命を決定する。Blood. 2006;107(11):4308-4316.
ArticleGoogle ScholarCrossref PubMed
48.
Zhang DE, Zhang P, Wang ND, Hetherington CJ, Darlington GJ, Tenen DG. CCAATエンハンサー結合蛋白質α欠損マウスにおける顆粒球コロニー刺激因子シグナル伝達と好中球の発達の欠如。Proc Natl Acad Sci U S A. 1997;94(2):569-574.
Google ScholarCrossref PubMed
49.
転写因子 C/EBP アルファ非存在下での造血幹細胞再増殖能と自己再生能の向上。Immunity. 2004;21(6):853-863.
Google ScholarCrossref PubMed
50.
Giladi A, Paul F, Herzog Y, et al.造血前駆細胞の単細胞特性解析と恒常性と摂動的造血におけるその軌跡。Nat Cell Biol. 2018;20(7):836-846.
Google ScholarCrossref PubMed
51.
Piunti A, Shilatifard A. PolycombファミリーおよびCOMPASSファミリーによる遺伝子発現のエピジェネティックバランス。Science. 2016;352(6290):aad9780.
Google ScholarCrossref PubMed
52.
Wang H, Wang L, Erdjument-Bromage H, et al. Role of histone H2A ubiquitination in Polycomb silencing(ポリコームサイレンシングにおけるヒストンH2Aユビキチン化の役割)。Nature. 2004;431(7010):873-878.
Google ScholarCrossref PubMed
53.
ポリコムを介したエピジェネティックなB細胞分化の抑制がT細胞のB細胞への分化を促進することを明らかにした。Genes Dev. 2016;30(22):2475-2485.
Google ScholarCrossref PubMed
54.
Sebe-Pedros A, Degnan BM, Ruiz-Trillo I. メタゾアの起源:単細胞の視点。Nat Rev Genet. 2017;18(8):498-512.
Google ScholarCrossref PubMed
55.
キャバリア-スミスT、チャオEE. Choanozoa, apusozoa, and other protozoa and early eukaryote megaevolutionの系統図。J Mol Evol.
Google ScholarCrossref
56.
Steenkamp ET, Wright J, Baldauf SL. 動物や菌類の起源は原生生物である。このような場合、「萌芽的研究」と呼ばれる。
Google ScholarCrossref PubMed
57.
このような場合、「動物がどのように生まれたか?また、このような研究成果をもとに、「動物の起源:単細胞から多細胞への移行における祖先の再構築」を提案した。2021;11(2):200359.
Google ScholarCrossref PubMed
58.
このような場合、「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」。J Invertebr Pathol. 1979;33(2):159-170.
Google ScholarCrossref
59.
Owczarzak A, Stibbs HH, Bayne CJ. Schistosoma mansoni母胞子嚢のBiomphalaria glabrataから分離したアメーバによるin vitroでの破壊:超微細構造の研究。J Invertebr Pathol. 1980;35(1):26-33.
Google ScholarCrossref
60.
Sebe-Pedros A, de Mendoza A, Lang BF, Degnan BM, Ruiz-Trillo I. また、このような環境下において、「環境保護」、「環境保全」、「社会貢献」、「環境保全活動」の3つの活動を推進する。このような背景のもと、本学科では、「生物多様性」をキーワードに、生物多様性の保全に向けた取り組みを進めています。
Google ScholarCrossref PubMed
61.
Cell. 2016;165(5):1224-1237.
Google ScholarCrossref PubMed
62.
キンドレッドJE. アーバシアの内臓周囲液におけるファゴサイトーシスと血液凝固.を "Biol Bull "と呼ぶ。1921;41(3):144-152.
Google ScholarCrossref
63.
このような場合、「臓器移植」を行う前に、「臓器移植」を行う前に、「臓器移植」を行う前に、「臓器移植」を行う前に、「臓器移植」を行う前に、「臓器移植」を行う前に、「臓器移植」を行う前に、「臓器移植」を行う前に、「臓器移植」を行う必要があります。Cell. 2013;154(5):1112-1126.
Google ScholarCrossref PubMed
64.
Ciona robusta hemocyte populational dynamics and PO-dependent cytotoxic activity. Dev Comp Immunol. 2020;103:103519.
Google ScholarCrossref PubMed
65.
Hagerstrand H, Danieluk M, Bobrowska-Hagerstrand M, et al. ヤツメウナギ(Lampetra fluviatilis)赤血球;形態、超微細構造、主要な細胞膜タンパク質とリン脂質、および細胞骨格組織。1999;16(2):195-204.
Google ScholarPubMed
66.
Pancer Z, Amemiya CT, Ehrhardt GR, Ceitlin J, Gartland GL, Cooper MD. アグナサンウミヤツメにおける可変リンパ球受容体の体細胞多様化。Nature. 2004;430(6996):174-180.
Google ScholarCrossref PubMed
67.
このような場合、「臓器移植」と呼ばれる。Nature. 2011;470(7332):90-94.
Google ScholarCrossref PubMed
68.
大野晋一郎:遺伝子重複による進化. Springer; 1970.
69.
Furlong RF, Holland PW. 脊椎動物は八倍体であったか?また、そのような生物学的な観点からの研究も行っている。
Google ScholarCrossref PubMed
70.
スコット・LM、シヴィン・CI、ロース・P、フリードマン・AD。骨髄単球系細胞における 3 種類の C/EBP ファミリーの新規時間的発現パターン。Blood. 1992;80(7):1725-1735.
ArticleGoogle ScholarCrossref PubMed
71.
CCAAT/enhancer binding protein epsilon 欠損マウスにおける造血障害、骨髄異形成、早期致死。Proc Natl Acad Sci U S A. 1997;94(24):13187-13192.
Google ScholarCrossref PubMed
72.
Cavalier-Smith T. 真核生物の食作用の起源と原生動物の系統分類. このような場合、「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」「痒いところに手が届く」。2002;52(Pt 2):297-354.
Google ScholarPubMed
73.
Yutin N, Wolf MY, Wolf YI, Koonin EV. 食作用と真核形成の起源。Biol Direct。2009;4:9.
Google ScholarCrossref PubMed
著者ノート
ホモログの発現量は、補足表3および5に記載した。その他の出典は、この論文のオンライン版で入手可能であった。
この論文のオンライン版には、データ補足があります。
本号には、本論文に関するBlood Commentaryが掲載されています。
本論文の出版費用の一部はページチャージの支払いによって賄われた.したがって,この事実を示すためにのみ,本論文は18 USCセクション1734に基づき,ここに「広告」と表示される.
© 2022 by The American Society of Hematology(米国血液学会
補足データ
補足の方法、参考文献、図、表- pdfファイル
補足表2- xlsxファイル
補足表3- xlsxファイル
補足表4- xlsxファイル
補足表5- xlsxファイル
補足表6- xlsxファイル
所属機関経由でサインイン
コメントを追加
第140巻 第24号
2022年12月15日
前の記事
次の記事
広告掲載
所属機関経由でサインインする
注目の記事
最初の血球はいつ、どのように進化したのか?
トーマス・グラーフ、血液、2022年
リング1の仲間はAML幹細胞を維持する
ヒーリー、ブラッド、2018年
Ring1AとRing1BはGlis2の発現を抑制し、マウスMOZ-TIF2 AML幹細胞を維持する
シマ, 血液, 2018
ヒトとマウスの肝形成における細胞系列分化の単一細胞トランスクリプトームレベルでの比較解析
Xin Wangら、Cell Research、2020年
アデノウイルスベクターを用いたHBVゲノムの骨髄への移行は、マウスの造血状態の変化をもたらす
Wenbo Zhuら、Acta Biochimica et Biophysica Sinica、2021年
インターロイキン 18 は皮膚炎症を悪化させ、イミキモド誘発乾癬のマウスモデルにおける微小膿瘍と鱗屑形成に影響を及ぼす
Niu, Xue-Li et al., Selections from Chinese Medical Journal, 2019年
による搭載
