
初心者の菌叢解析 Qiime2で解析(4) ファイルの準備編 ~manifestファイル~
自分のPCでQiime2を動かすことを目指してnoteを書いています。興味のある方はこれまでの内容もご確認いただければと思います。
1.Qiime2を動かすのに必要なファイル
以前の記事でも記載しましたが、必要なファイルが4つあります。
1. Sequenceファイル(.fastqもしくは.fastq.gz)
2. manifestファイル(.csv)
3. sample-metadataファイル(.txt)
4. classifierファイル(.qza)
今回は2つ目のmanifestファイルの準備を行います。
2.manifestファイルとは
manifestファイルとは、fastqファイルをQiime2用に変換する時に使用するファイルです。
具体的には、fastqファイルの名前を指定すると共に、どこにそのfastqファイルが入っているのかという保存場所の指示、そして、そのfastqファイルがforward配列なのかreverse配列なのかという配列の向きを指示するファイルです。
1.名前の指示 2.fastqファイルの保存場所の指示 3.配列の向きの指示
Qiime2の解析では、manifestファイルの通りにfastqファイルを認識し、Qiime2用のファイル(アーティファクト[.qza])に変換します。
3.解析用フォルダの作成
まずはデスクトップ上に今回用のフォルダを作成します。
私は「Qiime2_test」というファイルを作成しました。
初心者の方は同じ名前で作成するのが良いと思います。

また、今後の解析でファイル名をつける際に気を付ける点を以下に示します。
1.全角文字を使用しない
2.スペースを入れない × Qiime2 test (コマンドが切れて認識されます)
3.スペースの代わりにはアンダーバーを使用する ○ Qiime2_test
4.シャープを使用しない(#は認識されない場合があります)
5.スラッシュを使用しない (/はファイルの階層を表す記号のため)
これらを使用するとQiime2を動かした際にエラーが出る場合があります。
4.fastqファイルの移動
作成したQiime2_testフォルダの中に「sequence」というファイルを作成し、そこに解析するfastqファイルを入れます。

雑多なファイルが入っていますが、「1_L001_R1_001.fastq.gz」という感じの名前は、マウス糞便由来配列データの圧縮ファイルになります。
一方で、「DRR099585.fastq」という感じのデータは大阪湾由来の菌叢解析データになります。
R1はforward配列でR2はreverse配列になり、_1がforward配列_2がreverse配列になります。
5.manifestファイルの作成
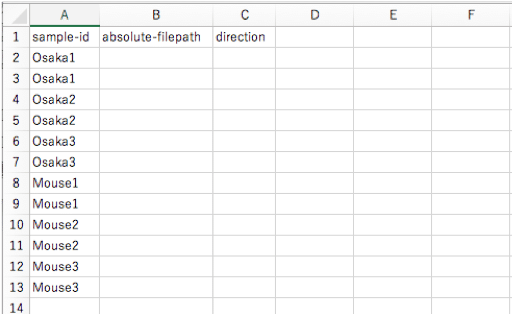
エクセルファイルを立ち上げ1行目に「sample-id」, 「absolute-filepath」, 「direction」とそれぞれセルを分けて打ち込みます。(タイプミスに注意)

6.サンプル名の入力
次に、サンプル名を「sample-id」の下に入れていきます。フォワードとリバースについて、それぞれ入れる必要がある為、同じサンプルの名前を2回入れていくことになります。
ここで入れた名前が解析の際のサンプル名になっていきます。
今回は大阪湾のデータとマウスのデータなので、以下の様に入れました。
7.絶対パスの入力
次に「absolute-filepath」を入れていきます。ここにはその名の通り、ファイルの絶対パスを入れていきます。
例えばOsaka1のフォワード配列は(デスクトップ)→(Qiime2_test)→(sequence)→(DRR099585_1.fastq)というようにPC内では格納されています。
これを絶対パスで表すと私の場合は以下の様になります。
/Users/ユーザー名/Desktop/Qiime2_test/sequence/DRR099585_1.fastq
スラッシュ( / )はファイルの切り替わりを示しており、まさにそのファイルがどこにいるかを示す住所になります。また、そのファイルへの道筋でもあるので「file path」というわけです。
ですので、このパスを入れることで、Qiime2は目的のファイルがどこにあるのか見つけることが出来ます。
ただ、実際に解析を行う場合はまずQiime2_testのフォルダを開いて、その中で解析を行いますので、Qiime2_testのフォルダより上流のパスは必要ありません。
よって、現在地を表す「PWD」という記号を使います。こちらを使用すれば、ユーザー名が違うPCでもパスを通せますし、下流が変化しなければファイルをまとめて移動してもパスは変化しないことになります。
フォワード配列とリバース配列のパスを入れていくと以下の様になります。先頭の「$」マークを忘れないでください。

2行目のファイルパスは(PWD=Qiime2_test)の中の(sequence)フォルダの中の(DRR099585_1.fastq)ファイルを示していることになります。
8.配列の向きの入力
最後に配列の向きを入力します。
小文字で「forward」もしくは「reverse」と入れていきます。

9.ファイルの保存
「名前をつけて保存」を選択し「manifest」と名前をつけます。また、ファイルの保存形式は「CSV UTF-8 (コンマ区切り)(.CSV)」を選びます。保存先は「Qiime2_test」のフォルダに入れてください。

manifestファイルの作成は以上になります。
10.manifestファイル作成の注意点
1.タイプミス
これは最も多いミスだと思います。
ファイルの保存名からファイルの中身に至るまで、1文字のミスでエラーが出てきます。私は「reverse」を「reverce」と間違えており、エラーでました。「PWD」を「PDW」としたこともありました。
2.サンプル名に「#」を使用する。
サンプル名(sample-id)に番号を付けるため、#を入れたことがあったのですが、エラーが出るので入れないでください。
3.ファイルの保存形式ミス
.CSVの形式で保存できていない場合があります。保存後、テキスト系のソフトで開いて、文字がコンマで区切られているか確認してください。
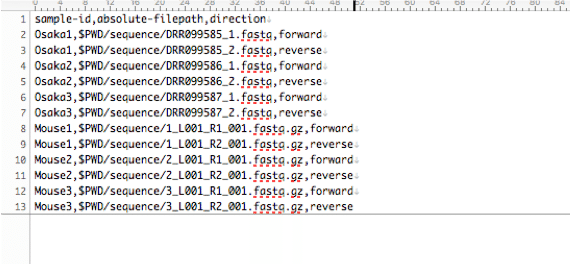
エクセルで編集し上書き保存をした際、保存形式がかわってしまったことがありました。その時はエクセルで開き直すと以下の様に全てのセルがくっついていました。この場合は再度作成し直してください。

11.これまでに出会ったエラーコード
今回作成したmanifestファイルを用いて、Qiime2にfastqファイルの取り込みをしてもらいます。しかし、manifestファイルに不備があるとエラーコードが出ます。今まで見てきたエラーコードを紹介します。
1.保存場所のミスの場合
(1/1) Invalid value for '--input-path': Path 'manifest.csv' does not exist.こちらはエラーコードの通り、manifestファイルがないといわれております。原因は以下が考えられます。
1.作成したmanifestファイルがちゃんと任意のフォルダの中にない
2.保存形式が.csvになっていない(もしくは指示通りの拡張子)
3.ファイル名が間違っている
2.タイプミスの場合
There was a problem importing manifest.csv:
manifest.csv is not a(n) PairedEndFastqManifestPhred33 file:
Found header on line 1 with the following labels: ['sampl-id', 'absolute-filepath', 'direction'], expected: ['sample-id', 'absolute-filepath', 'direction']こちらはmanifestファイルの中にミスがある場合です。
中身を読みますと「ファイル内の1行目」名前が間違っているそうです。
よく見ると「sample-id」の部分が、「sampl-id」になっています。
3.配列の向きの入力ミス
An unexpected error has occurred:
Each sample id can have only one forward read record in a paired-end read manifest, but the following sample ids were associated with more than one forward read record: Osaka1
See above for debug info.これは「reverse」のところに「forward」と入れてみたときのエラーです。エラーコード内でちゃんとそのことが指摘されています。
4.サンプル名に「#」を入れたときのミス
An unexpected error has occurred:
Empty cells are not supported in manifest files. Found one or more empty cells in this record: Osaka,nan,nan
See above for debug info.こちらはsample-idの部分に「#」を入れたときのエラーです。「#」は以下の文字を認識しないようにするコードのため、ここではエラーになりました。「Osaka1」を「Osaka#1」にしたエラーです。
今回は以上になります。
長文にお付き合いいただきありがとうございました。
