
初心者の菌叢解析 Qiime2で解析(1)Qiime2インストール編(2023.3.14追記)
1.はじめに
消化管の中に生息する「腸内細菌」は今や一大研究テーマとなっており、人々の健康の維持・増進を考える上では避けられないキーワードです。
1990年より開始された「ヒトゲノム計画」によって培われた次世代シーケンス技術は、これまで完全にブラックボックスであった腸内の微生物環境を解析する強力なツールとして現在活用されています。
その中で明らかとなってきたことは、腸内細菌は非常に複雑な生態系を構成しており、宿主と密接に相互作用しているということでした。
腸内細菌はその複雑さから「腸内フローラ(花畑)」や「腸内細菌叢」と呼ばれています。
このように重要な腸内細菌叢ですが、その解析にはPCを用いたバイオインフォマティクス的な手法が必要になってきます。
この度、腸内細菌叢解析に携わることになりまして、解析プラットフォームの「Qiime2」を動かす必要性が出てきました。
初心者ですが、色々なサイトを見ながら試行錯誤し、一旦は解析出来そうな感じになりました。
初心者が気軽に菌叢解析を始められるような、ハードルの低いサイトや書籍が無いため、これまでのQiime2の使い方等を共有させていただければと思い投稿を始めました。
ここではQiime2をmacのPC上で動かせるようにするまでを記載いたします。Windowsで行いたい方は、別の記事を投稿しておりますので、そちらをご確認ください。
腸内細菌叢を自分で解析できるようになれば、研究費の大幅な節約になりますし、多角的な研究データの解釈が可能になります。
これから、卒論、修論、博論を控える学生の中で、腸内細菌叢解析を行いたい方のFigureが一つでも増えればと考えています。
また、Qiime2の使い方を試行錯誤している方の助けになれば幸いです。
どうぞお付き合いのほどよろしくお願いいたします。
2.Anacondaのインストール
以前はDockerを使用していましたが、こちらの方が簡単だったので、Anacondaを経由してQiime2を動かしたいと思います。
以下のAnacondaのホームページからダウンロードを行います。
インストールの詳細等は他に分かりやすい記事を出している方が沢山いらっしゃいますので、私もそういった方に助けられています。
インストールを行った後に「ターミナル」を起動します。
以下のコマンドを入力しエンターキーを押せば(入力)すれば、
使用できるコマンドが列挙され、無事にAnacondaがインストールされたことを確認できます。
condaコマンドを入力すると以下の様な出力が得られると思います。
ただ、「zsh: command not found: conda」のようなエラーが出る場合もあります。その場合は、以下に対処法を示してあります。

「zsh: command not found: conda」のようなエラーが出た場合、
以下のコマンドにて「conda環境」を有効に出来ます。
/Users/(ユーザー名)/opt/anaconda3/bin/conda init zsh(ユーザー名)にはご自身で設定したPCのユーザー名が入ります。
上記コマンドを実行したのちに、ターミナルを再起動し、再度「conda」のコマンドを入れてみてください。
それでもうまくいかない場合は、
まずはアプリケーションの「Anaconda Navigator」を起動します。
これは、Anacondaのインストールに伴って、Macのアプリケーションとしてダウンロードされていると思います。
Anaconda Navigatorの左のタブから「Environments」を選択し、「base (root)」右側の再生ボタンを押します。
その中に、「Open Terminal」がありますので、そこをクリックします。

そうするとターミナルが起動し、conda環境がアクティベーションされます。
ここで起動したターミナルでは、「conda」が使用できると思います。
起動したターミナルは以下の画像のようになっていると思います。
ここの赤枠内がそれぞれのAnacondaがインストールされている場所です。
この赤枠のコマンドの後ろに「conda init zsh(もしくはbash)」を付加したコマンドを新しいターミナルに入力して実行します。
その後、ターミナルを再起動し、「conda」を実行してみてください。

「bash」だったら「conda init bash」で実行する。
3.Qiime2の環境構築
Qiime2をインストールしますが、ポイントとして、インストールは最新のバージョンではなく「Version: 2021.11」をインストールしています。
本当は最新のものが良いと思いますが、今後動かしたい「PICRUSt2」のはこちらのバージョンでないと動きません。
必要に応じて最新バージョンをインストールして下さい。
(2023.3.14追記)
最新のMacを使用する場合、CPUが「M1, M2チップ」に変更になっていると思います。
いわゆる「Apple Silicon」の場合は最新の「Version: 2023.2」でのみQiime2を起動可能となっています。
3-1. Anacondaのアップデート
コマンドをターミナルに打ち込み、Anacondaのアップデートを確認します。
conda update conda確認が出ますので、半角の「y」を打ち、Enter(return)キーを押します。
Proceed ([y]/n)? y
3-2. wgetのインストール
以下のコマンドを打ち込み、Web上からダウンロードを行うツール「wget」をインストールします。
conda install wget確認が出ますので、「y」を打ち込み、Enter(return)キーを押します。
3-3. condaの環境下でQiime2をインストールする
以下のコマンドを打ち込み、Qiime2をインストールします(2021.11)。
wget https://data.qiime2.org/distro/core/qiime2-2021.11-py38-osx-conda.yml
conda env create -n qiime2-2021.11 --file qiime2-2021.11-py38-osx-conda.ymlインストールが開始されると必要な各パッケージがダウンロードされます。
M1、M2チップ搭載のMacの場合は、「Version: 2023.2」以降のQiime2をインストールする必要がありますので、以下のコマンドを入れてください。
wget https://data.qiime2.org/distro/core/qiime2-2023.2-py38-osx-conda.yml
CONDA_SUBDIR=osx-64 conda env create -n qiime2-2023.2 --file qiime2-2023.2-py38-osx-conda.yml
conda activate qiime2-2023.2
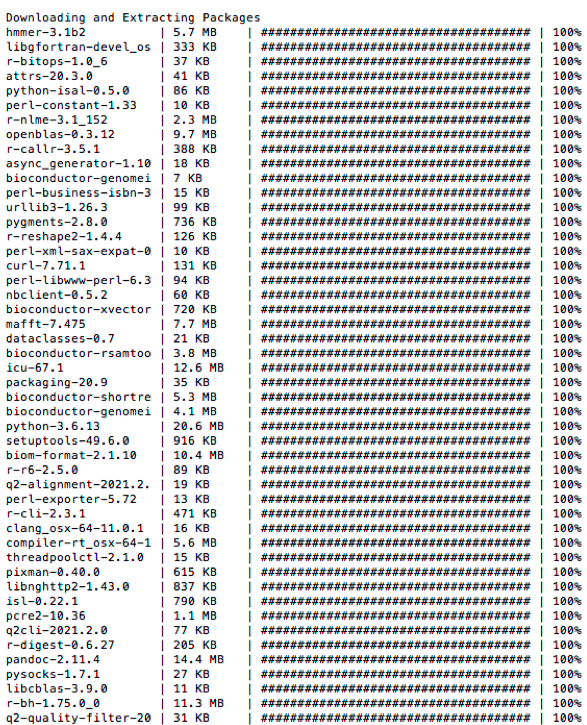
conda config --env --set subdir osx-64同様に以下のようなインストール画面が見えると思います。
4.Qiime2の環境をアクティベート
以下のコマンドを打ち込み、Qiime2を起動します。
conda activate qiime2-2021.11続いて、以下のコマンドを打ち込むことで、動作確認が行えます。
qiime --help以下の画像の様にQiime2のコマンドラインを見ることが出来ると思います。
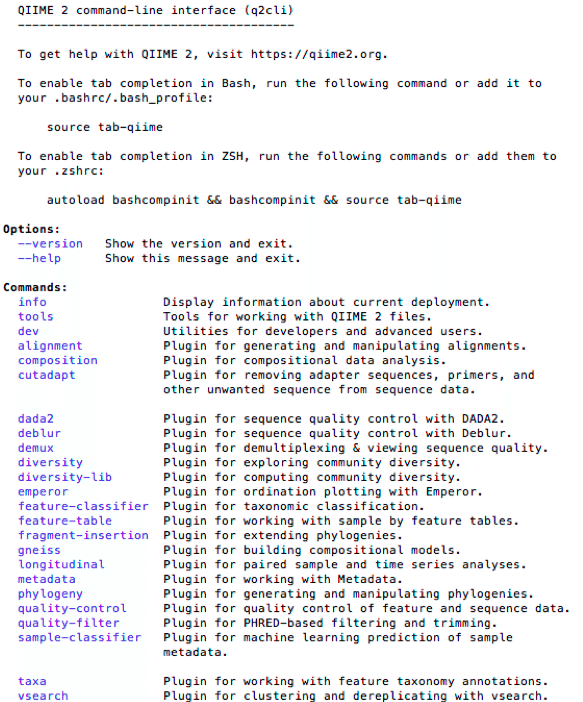
インストールまでは以上となります。
Qiime2の環境から出る場合は以下のコマンドを入れてください。
conda deactivate次はPICRUSt2のインストールを行おうと思います。
不備等あると思いますが、ご容赦下さい。
5.参照
インストールは以下のサイトの方法を参照しております。
※上記のページでは最新のDocumentsを参照するように進められますので、以下の「Show me the content on this page」をクリックすれば該当バージョンのページが確認可能です。
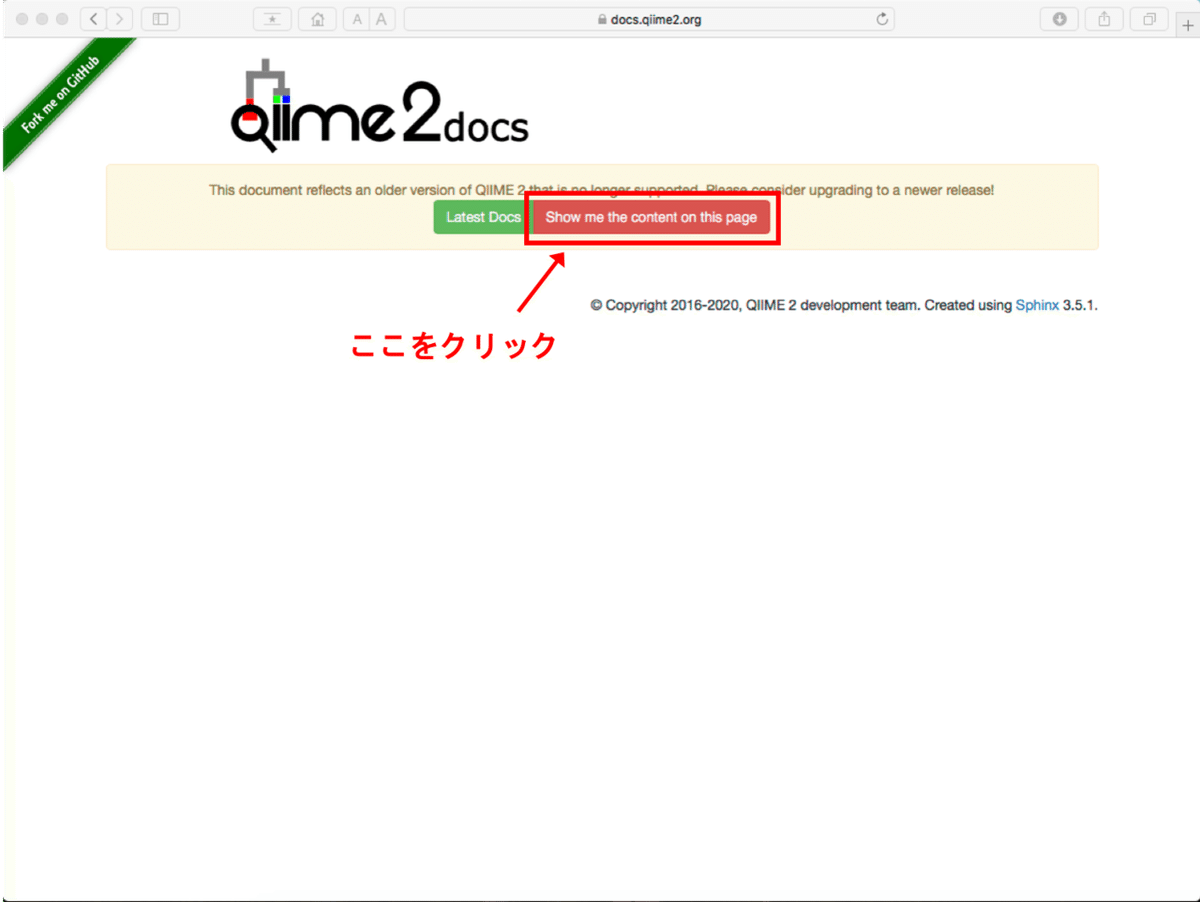
最新バージョンのQiime2をインストールする場合は上図の「Latest Docs」の方から行けると思います。
