
初心者の菌叢解析 Qiime2で解析(14) 菌叢のグラフを作成する
本投稿ではこれまでに解析した菌叢解析の結果からグラフを作成することを目的としています。
興味のある科や属、株を抽出し、その存在割合のグラフ作成を行います。
本解析にはこれまでのQiime2解析結果が必要になりますので、興味のある方は過去の投稿もあわせてご確認ください。
1.Qiime2の起動と解析ディレクトリの作成
まずは解析ディレクトリに移動し、Qiime2を起動します。
cd /Users/ユーザー名/Desktop/Qiime2_test
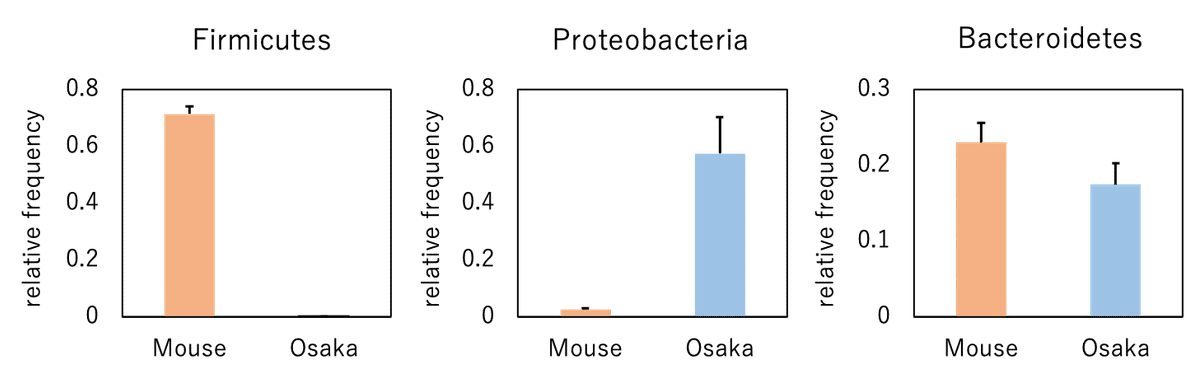
conda activate qiime2-2021.2今回はLevel 2(門レベル)での解析結果を抽出し、グラフを作成します。
そこで、まずはLevel 2の表を出力する為のディレクトリを作成します。
mkdir taxonomy_table taxonomy_table/Level_2デスクトップ上の「Qiime_test」ディレクトリ内に「taxonomy_table」ディレクトリが作成され、その中に「Level_2」のディレクトリが作られたと思います。
今回の解析で必要なファイルは以下の2つです。
1.table.qza
2.taxonomy.qza
これらファイルの取得方法は以下の投稿をご確認ください。
2.ファイルの出力
以下のコマンドを打ち込み、表を出力します。
qiime taxa collapse \
--i-table table.qza \
--i-taxonomy taxonomy.qza \
--p-level 2 \
--o-collapsed-table taxonomy_table/Level_2/collapsed_table.qza「--i-table table.qza」と「--i-taxonomy taxonomu.qza」はインプットするファイルを指定しています。
「--p-level 2 」は出力ファイルののレベル(階層)を指定しており、
Level 2〜7の数字を入れることで、各階層のデータを出力できます。
違う階層を出力する場合は、違うディレクトリを「taxonomy_table」の中に作成することをおすすめします。
うまくいけば「collapsed_table.qza」ファイルが出力されます。
表の出力結果として、シーケンスのリード数としてデータを出力する方法と相対値として出力する方法の2つがあります。
リード数のデータはシーケンサーのスペックやサンプル調製の手技で変化しますので、基本的には相対値(%)で解析すると思います。
そのため、データを相対値に変換していきます。
以下のコマンドで、相対頻度に変換します。
qiime feature-table relative-frequency \
--i-table taxonomy_table/Level_2/collapsed_table.qza \
--o-relative-frequency-table taxonomy_table/Level_2/relative-collapsed_table.qza3.テキストファイルへの変換
次にファイルをエクスポートし、テキストファイルに変換します。
qiime tools export \
--input-path taxonomy_table/Level_2/relative-collapsed_table.qza \
--output-path taxonomy_table/Level_2/exported-feature-tablebiom convert \
-i taxonomy_table/Level_2/exported-feature-table/feature-table.biom \
-o taxonomy_table/Level_2/exported-feature-table/table.from_biom_w_taxonomy.txt \
--to-tsv \
--header-key taxonomyうまく進めば「Level_2」ディレクトリの中に「exported-feature-table」ディレクトリが生成され、その中に「table.from_biom_w_taxonomy.txt」が生成されたと思います。
4.グラフの作成
出力したテキストファイルをエクセルで開きます。

門レベルでの相対存在量のデータを取得できました。
このデータを用いて、グラフを作成してみます。

今回は以上になります。
読んでいただきありがとうございました。
