
【W3】部分構造による化合物フィルタリング_06_Step3_前編
【本パート(W3)の目的】
いくつか私たちのスクリーニングライブラリーに含めたくない部分構造があります。このトークトリアルでは、そのような好ましくない部分構造の様々なタイプを学び、そしてRDKitを使ってそれらの部分構造を見つけ、ハイライトする方法を学びます。
上記はPython版のT3の説明ですが、W3の目的も同じです。
【化合物部分構造フィルタリングの結果】
これまでPAINSとBRENKでのフィルタリングを体験しました。
すなわち、好ましくない部分構造を検索式とした検索を行いました。
Step3は、RDKitを使ってそれらの部分構造を見つけ、ハイライトする方法を学びます。
また、出力前のデータ整形などを行います。

今回はStep3上部の4つのノードについて紹介します。
【Concatenate】

同じ形式の複数ファイルを上下にくっつけて1つにすることができます。
今回上下にくっつけるのは、PAINS(Step1)とBRENK(Step2)の両フィルターでのヒットすなわち除外された化合物群をまとめます。
設定:

例えばカテコール類縁体などPAINSとBRENK両方でヒットすると2個目のデータはRowIDの末尾に”_dup”が付きます。
Column handlingについてはまっきーさんの記事を引用します。
くっつける際に、KNIMEはコラム名が同じもの同士をくっつけてくれます。コラム数が違って、片方にない場合は欠損値として空白セル(KNIMEでは"?" / "missing value")として入力されます。
このとき、くっつける2テーブルは、「コラム名だけでなくデータ型までそろえる」ことを鉄則としてください。
今回は揃っていますので特に注意は必要ないです。
結果:
Step1とStep2の結果を合わせて
394+2349=2743行になりました。
【RDKit Molecule Highlighting】

Creates an SVG column showing a molecule with highlighted atoms and bonds based on information in the input table. A molecule column as well as column(s) with a list of the atom and/or bond indexes to be highlighted needs to be provided.
入力テーブルに原子(と結合)のリストがあると、その部分を強調表示したSVG形式のカラムを出力します。デモデータで見ていきましょう。
設定:

入力に”Atom List”というカラムがあり、該当する原子団を赤色に強調表示します。
結果:
Descriptionに記載されている忌避構造の原子のIDがAtom Listにリスト形式で入力されているので、Highlighting substructuresカラムでは下図のように強調されています。
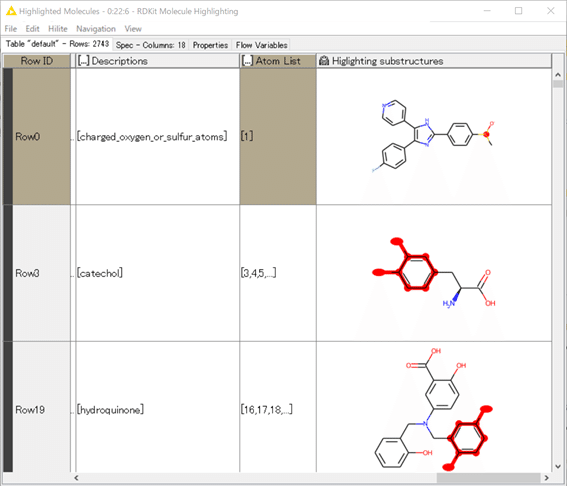
便利な機能ですね。RDKitは化合物の表示に関しても数多くの機能が実装されています。Pythonを使うことに抵抗がなければ下記の記事は一見の価値があると思います。
【Column Filter】
既に何回か利用しているノードですね。データ整形には必須ともいえるノードです。念のためまっきーさんの記事は引用しておきます。
設定:

結果は省略します。次のTable Viewに表示もしくはCSVファイルに出力したいデータ種を選定しました。
【Table View】
W1-2でも出力で頻用していますので説明は必要ないでしょう。
設定:

結果:
Viewの左半分

右半分

SVG形式だと構造式もTable Viewに表示できていいですね。
さて上記のTable Viewですが、W3のメタノードの外にあるTable View(上)つまり
Compounds with PAINS/Brenkと全く同じ設定なので、こちらの説明で代えさせていただきます。このパターン多いですね。

では次回はW3の最終回、Step3の下部へ進めます。
いいなと思ったら応援しよう!

