
【W4】リガンドベーススクリーニング_06_Step2_MorganとMACCS
【本パート(W4)の目的】
化合物をエンコード(記述子、フィンガープリント)し、比較(類似性評価)する様々なアプローチを取り扱います。さらに、バーチャルスクリーニングを実施します。
上記はPython版のT4の説明ですが、W4の目的も同じです。
そのための教材として
既知のEGFR阻害剤ゲフィチニブ(Gefitinib)をクエリとして使用し、EGFRに対して試験済みの化合物データセットの中から類似した化合物を検索します。
【類似性評価結果の比較】
Step1で化合物をエンコードし、Step2を体験中です。
今回はMorganとMACCSを用いた類似性評価結果の比較をしてみます。
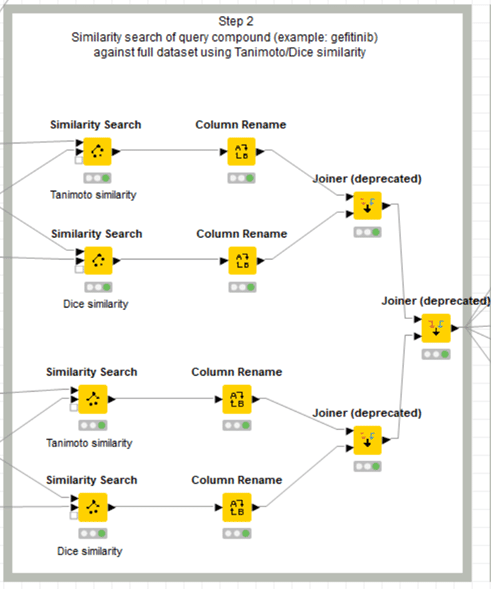
上図の上半分はすでに説明した通りです。
①Morgan2フィンガープリントでTanimoto係数を指標とした類似度評価
Morgan2フィンガープリントでDice係数を指標とした類似度評価
とJoinerでのデータ統合
下半分は
A) MACCSフィンガープリントでTanimoto係数を指標とした類似度評価
B) MACCSフィンガープリントでDice係数を指標とした類似度評価
と
C) Joinerでのデータ統合
そして
D) 上下パートのJoinerでのデータ統合
ですので、使うノードの種類は同一で、ごくわずかに設定が異なるのみです。
一部だけ設定を以下に示して、ほかは省略させていただきます。
A)のSimilarity Searchノード設定
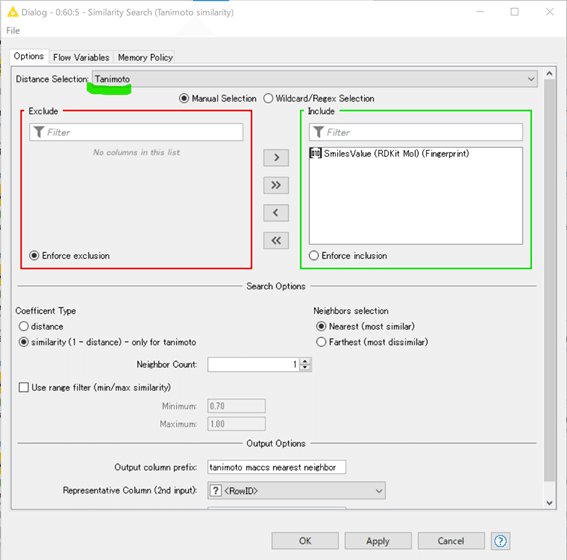
MACCSフィンガープリントでTanimoto係数を指標とした類似度評価となります。
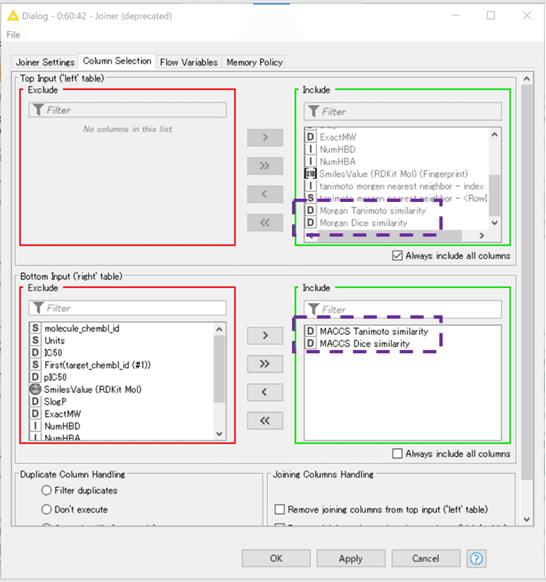
D)の4つの類似度評価結果の統合
設定:
結果:フォントサイズやカラムの並びは編集してあります
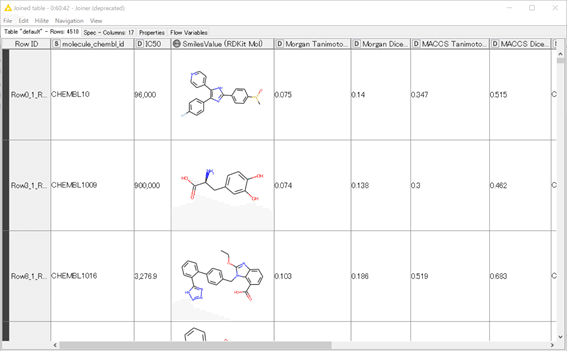
一見して
Morgan2よりMACCS
TanimotoよりDice
を用いた類似度の方が高めにスコアリングされているのが見て取れます。
以上でStep2は完了です。次はW4の最終Step3へ進めます。
おまけ:
各化合物ではなく全体の傾向も見てみましょう。
【Statistics】
Step2の最後のJoinerに以下のように2つのノードを繋げます。
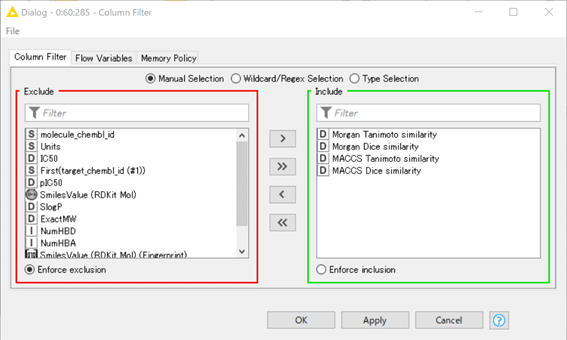
Column Filterで集計したいカラム4つだけに絞り、
Statisticsノード

このノード、意外に日本語での紹介記事が少ないです。
使い方は簡単です。上記記事を引用します。
Node Repository」から「Statistics」ノードを配置し、「Column Rename」ノードの出力ポートと接続します。「Statistics」ノードの信号マークが黄色に変わるのでそのまま処理を実行します。
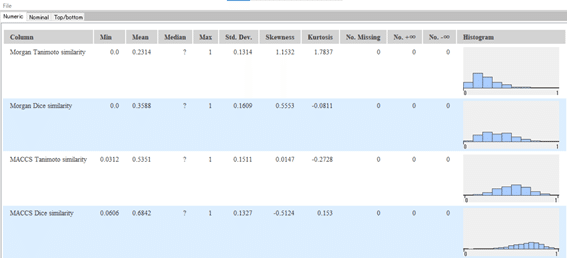
実行完了した「Statistics」ノードを右クリックし、メニュー中段の「View:Statistics View」を選択すると、統計情報(最大値、最小値、標準偏差など)が表示されます。
ただし、今回は扱うデータ数などの都合でデフォルト設定だとエラーが出ます。
WARN Statistics 0:60:284 Maximum number of unique possible values (1000) exceeds for column(s): "Morgan Tanimoto similarity","Morgan Dice similarity"
そこで設定を下記のようにしますとエラーは出ないです。
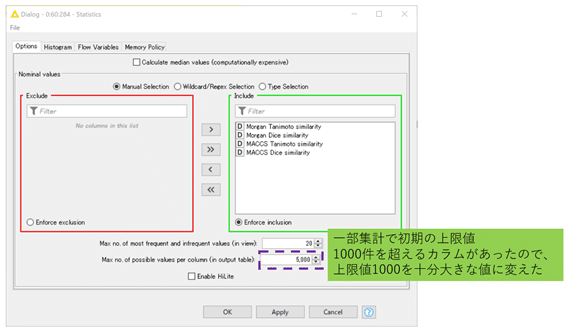
一部集計で1000件を超えるカラムがあったのでエラーが出ました。
元データが4510行だったら5000あれば集計しても上限を超えないです。
(今回だと実際には2000とかでも大丈夫)
結果:
メニュー中段の「View:Statistics View」を選択
日本語化したノードディスクリプションより
このノードでは、最小値、最大値、平均値、標準偏差、分散、中央値、全体の合計、すべての数値列における欠損値の数と行数など
をまとめて算出してくれるので便利です。
今回はヒストグラムを眺めることにします。
実行完了した「Statistics」ノードを右クリックし、メニューの下から3番目「Statistics Table」を選択
表示されたTableでカラムの並べ替えをしました。
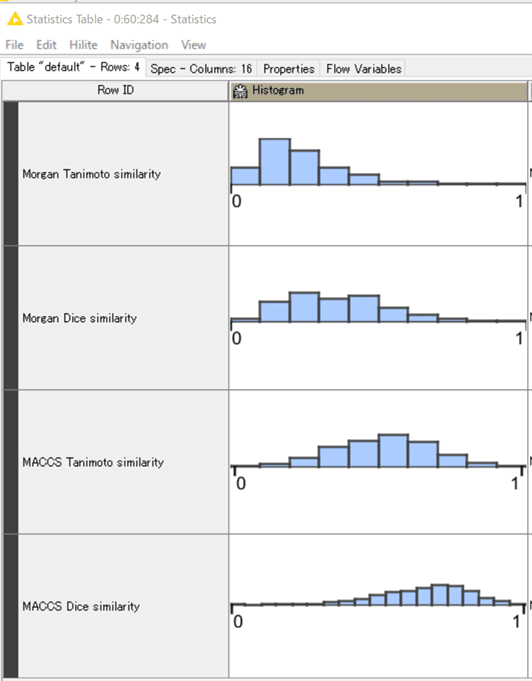
残念ながらカラム数が揃えられていないので比較はしにくいのですが、
各類似度評価で類似度がどんな分布になっているかは大まかに見ることができます。
例えばですけど、類似度0.7以上をクライテリアとして化合物をフィルタリングしたとして、上記4種の類似度評価方法でヒット数がかなり異なることは自明ではないかと思います。
いいなと思ったら応援しよう!

