【W6】最大共通部分構造_04_Step2
【W6の目的】
サイズの大きい化学データのクラスタリングとクラス分類は、医薬品探索の化学を利用する広範な領域において、研究を導き、分析し、知識を発見することにとって不可欠です。
一つ前のトークトリアルで、化合物をグループ化する方法(クラスタリング)を学び、一つのクラスターに含まれる化合物がお互いに似通っており、共通の骨格(scaffold)を共有していることを見つけました。視覚的に調べることに加えて、ここでは化合物セットが共通してもつ最大の部分構造を計算する方法を学びます。
上記はPython版TeachOpenCADDのT6についてのmagattacaさんの記事から引用しましたが、KNIME版のW6も同様です。
【MCSの可視化】
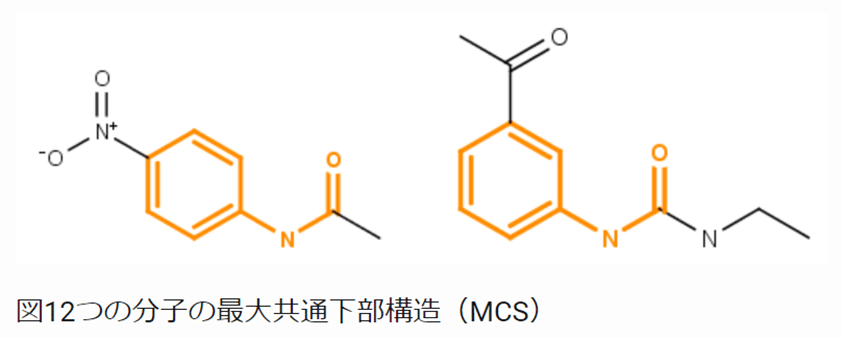
最大共通部分構造(maximum common structure、MCS)は2つあるいはそれ以上の対象化合物に含まれる最大の部分構造として定義されています。
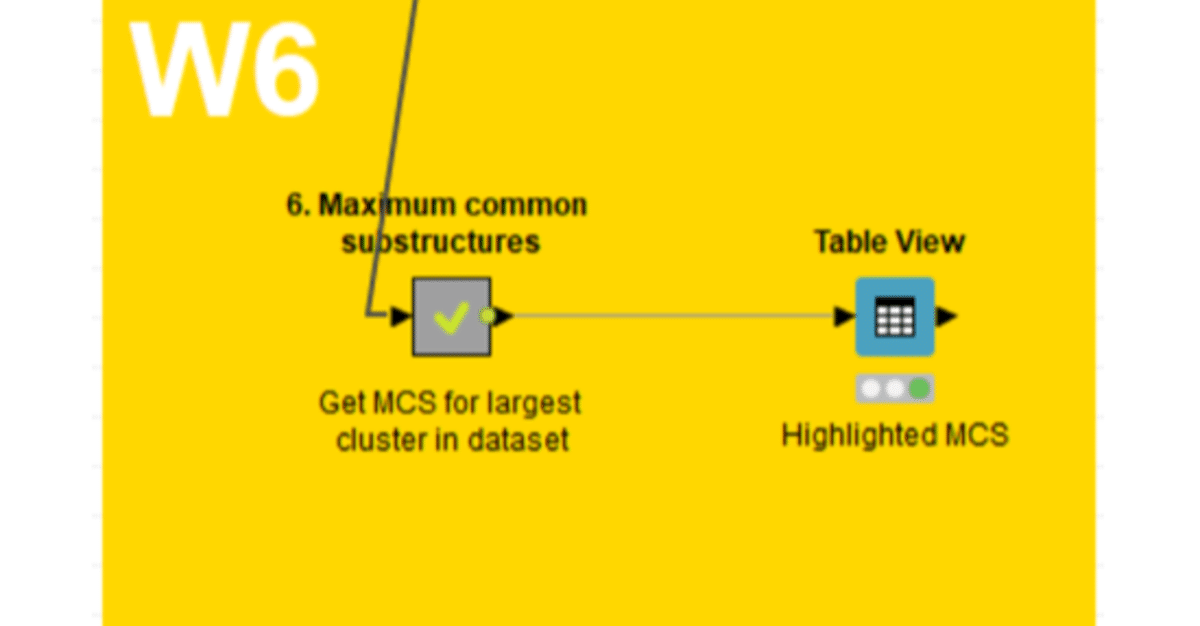
前回までのStep1でMCS解析を体験しました。
Step2はデータ表示のための後処理とTable形式での可視化です。

【Column Filter】
TeachOpenCADDではもはやおなじみのノードだと思います。
出力処理の前は大体このノードを使っているのではないでしょうか?
最近では下記の記事でも扱いましたので技術紹介は割愛します。
設定:

結果:

以下は個人的な感想になりますが、MCSを可視化したいという目的ならもっと表示カラムを削減してもいいように思いました。
また、上記設定では左ウィンドウで定義したカラムだけを省く(Enforce exclusion)のですが、逆に右ウィンドウで定義したカラムだけを表示するように設定(Enforce inclusion)しておくと、今後workflowを編集した際にも表示されるカラム種が固定されます。
設定方法の一例を以下に示しておきます。
設定例:

以上細かい余談でした。
【Table View】
こちらもTeachOpenCADDでは多用されてきたノードですので技術面は割愛でいいと思います。今回のデモデータではSmilesValue (RDKit Mol) (Highlighting)カラムがSVG imageという画像データなので化合物の構造も表示できますね。
あと、W6 Step2のTable ViewノードはMetanodeの外にある下図のノードと設定が同一ですのでこちらでまとめて扱います。
設定:

以下再び感想です。
ここでColumn Filter では残したIC50とpIC50を非表示に設定するという設計は意外でした。それならUnitやFirst(target_chembl_id(#1))も非表示でもいいのではと思いました。
一方で、workflow修正を色々する中でTable Viewでの表示項目まで細かくは修正影響を追わなくても、ケモインフォマティクスの学習という大きな目的は達せられると考えます。
もし気づいた人は自分でカスタマイズすればより理解が深まる機会ともいえるのではないでしょうか?
さて余談ばかりになってきたのでこれぐらいにして結果を見てみます。
結果: Interactive Viewにて

MCS部分が赤色に着色され、化合物の配置もそろえて一覧表示されますので各化合物のMCSとそれ以外の部分を一目で把握できて便利です。
またも余談ですが、
今回のデモデータのMCSにはマイケルアクセプター構造が含まれていますね。
以前に取り上げたアファチニブも思い出しました。
実はMCS解析前の156化合物にはよりアファチニブに部分構造が一致する化合物も含まれてはいたのですが、除外された10%すなわち156-142 = 14化合物側でしたのでこのViewでは表示されないです。
以上でW6の全workflowを体験しました。次回はW7に行く前にMCSの解析条件を変える体験をします。
おまけ:
【MSCでのクラスタリング JKlustor】
MCSは創薬化学者にとって直観的に理解しやすそうな化合物分類法であると思っていただけたでしょうか?使ったことがないのですがJKlustor技術も紹介します。
LibMCS 階層的な最大共通部分構造(maximum common substructures)に基づきクラスタリングをおこないます。ダイバシティー解析や focused set profilingに用いることができます。
LibMCSの説明サイトをGoogle翻訳して引用します。
ライブラリMCSは、複合ライブラリの最大共通部分構造(またはMCS)を階層的に検索します。最大共通部分構造の概念は、2つのグラフ(この場合は2つの化学構造)に対して定義され、両方の構造に共通する最大の部分グラフ(部分構造)を指します。
化合物のセット内のすべての化合物のペアワイズ比較(正確な方法)は、n・(n-1)/ 2 MCS計算を伴うため、実際には実行不可能です。ここで、nはライブラリ内の化合物の数を示します。 。1000の構造の場合、499,500のペアワイズ計算が必要です。このような比較が1つでもかなり複雑で、かなりの計算時間が必要です。たとえば、特定のハードウェアで1回のMCS検索の計算時間が10ミリ秒である場合、1000ライブラリの完全な評価には4995秒、つまり約1.5時間(5000分子の場合は35時間)かかります。
LibMCSによって適用される構造ベースの階層的クラスタリングは、2つの実質的な利点(高速、ターボ法)で上記の問題を克服するための実行可能なアプローチを提供します。

パトコア社の下記ブログを見つけて「おおっ」と見てみました。
化合物のライブラリーサイズが増加していることに伴い、類似の性質・構造を持つ化合物群に分類する手法が発達してきました。化学構造のフィンガープリントを使った構造類似度に基づく分類法(ウォード法、k平均法)や、部位構造に着目した分類法(MCS、BMF)が挙げられます。この目的でChemAxonからはJKlusterというパッケージが提供されています。
分析方法は置いておいて、
…「お、おぅ」となりました。
分類法のひとつLibMCSもいつかくわしく解説していただけたら嬉しいです!
いいなと思ったら応援しよう!

