
【W2】指標による化合物フィルタリング_03_Step2
【本パート(W2)の目的】
W1でChEMBLから取得した化合物群を
ルールオブファイブ(Ro5)のクライテリアに基づきChEMBLから集めた化合物をフィルタリングします。
薬らしくない分子を取り除く手法の一例を学ぶことが目的です。
(原典)
https://magattaca.hatenablog.com/entry/2020/04/12/180559
【Step1のおさらい】
「2. Molecular filtering: ADME and lead-likeness criteria」メタノードの中の
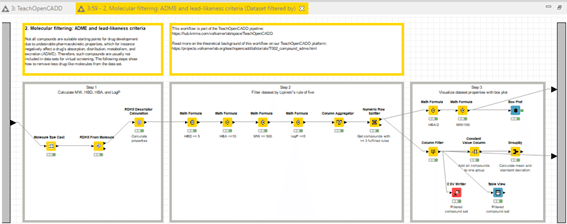
Step1: Calculate MW, HBD, HBA, and LogP
でRo5の判定に用いる、4つの化学計算を終えました。
Step2は判定パートとなります。

【リピンスキーのルールオブファイブ(Ro5)】
早速ですがMagattacaさんの記事を引用します。
生物学的利用可能性(バイオアベイラビリティ)は重要なADME特性で、化合物の構造のみに基づいてこの特性を計るために、リピンスキーのルールオブファイブ(Lipinski's rule of five)が発明されました。これは経験則で、化合物の経口投与によるバイオアベイラビリティを見積もるのに役立ちます。
ルールオブファイブによると、ある化合物が次のルールの一つ以上を破っているとき、経口で体内に吸収される可能性が低くなるとされています。そのルールとは即ち:
• 分子量が500ダルトン以下
• 水素結合アクセプターが10以下
• 水素結合ドナーが5以下
• LogP (オクタノールー水 分配係数) <= 5
LogPは分配係数あるいはオクタノールー水分配係数とも呼ばれます。通常、疎水性(例:1-オクタノール)と親水性(例:水)の相の間における化合物の分配を測定します。
疎水性分子は水に対する溶解度が低くなる一方で、より親水性の分子(例:水素結合のアクセプターとドナーの数が多いもの)や大きな分子(分子量の大きいもの)は、リン脂質二重膜を透過しづらくなります。
ルールオブファイブに関して、全ての数字が5の倍数になっています。これがこのルールの名称の起源となっています。
(Adv. Drug Deliv. Rev. (1997), 23, 3-25)
今回、W2のStep2では上記4条件のうち、3つを満たせばよいと少し緩めて化合物選抜をします。
【Math Formula】 (HBD <=5)

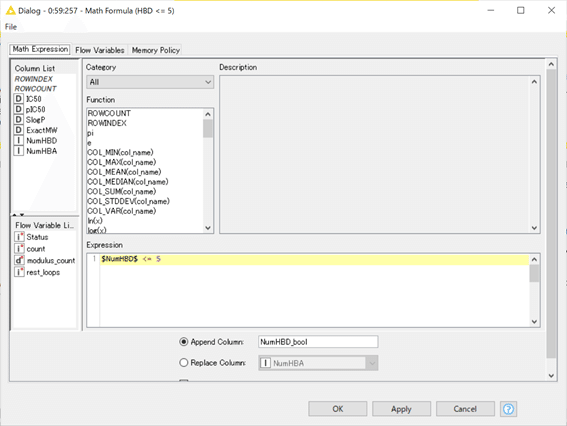
Math Formulaノードを使い、
NumHBD(Number of Hydrogen Bond Donerの略) ≦5
すなわち
• 水素結合ドナーが5以下
であるかどうかの正否判定を行い、
NumHBD_bool
というカラムにboolean(ブール値)すなわち
「0」(否,False)または「1」(正,True)
のどちらかを出力しています。

Rule-based Row Filter
とかで絞ってしまうのではなく、4つのルールの判定結果を出して後で4条件のうち、3つを満たせばいいという絞り方なのでこういう設計になっています。
【Math Formula】 (HBA <=10)
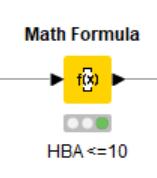
• 水素結合アクセプターが10以下
を同様に判定しています。
次へ進みます。
【Math Formula】 (HBA <=10)

・分子量が500ダルトン以下
を同様に判定しています。
次へ進みます。
…と行きたかったです。本当に。
(補足:フリー体分子量について)
ここでふとちゃんと分子量ってフリー体分子量を計算してるかな?
と確認して見たところ、塩分子もまとめて算出していますね。
workflowを編集しながら説明すると長くなりすぎるので、別途独立して扱います。
デモデータを使ってKNIMEでのケモインフォマティクス体験をする目的で進めているので、ごく一部のデータ処理に議論の余地があっても大勢には影響ないと祈りつつこのまま進めますこと容赦ください。
【Math Formula】 (logP <=5)

• LogP (オクタノールー水 分配係数) <= 5
を同様に判定しています。
ここまでの結果を振り返ると、下図の通り
4つのカラムが増えて「1」または「0」が入力されています。

次へ進みます。
【Column Aggregator】

上記4つのノードで
• 分子量が500ダルトン以下
• 水素結合アクセプターが10以下
• 水素結合ドナーが5以下
• LogP (オクタノールー水 分配係数) <= 5
の合否判定をして「1」または「0」が入力されています。この4カラムの集計を行います。4カラムのうち、いくつが「1」すなわち「合格」だったかを算出します。
<SettingsタブのColumnsウィンドウ>集計したい4つのカラムを選び、

<SettingsタブのOptionsウィンドウ>4つの行を加算する処理を設定、

Column Nameには処理した結果の出力カラム名が”Sum”と入力されています。変更も可能ですが、このまま行きます。
このノード、複数カラムのデータをまとめるには便利そうなんですが、正直いって初めて知りました。
まっきーさんですら扱っておらず、英語の情報しか見つけられませんでした。
意訳しようかとGoogle翻訳を最初に見てみたら、翻訳精度はかなりのものでした。
出る幕なし。

Missing valueは「欠損値」と訳してほしかったですが、上記でも意味は分かります。
結果は例えば下図の通り、Sumが算出されていて、いくつかの化合物だと3より小さい値になっています。

【Numeric Row Splitter】

ノードにコメントされている通り、Ro5の4ルールのうち、3つ以上を満たす化合物だけを残します。
<参考> Row Splitterやその類縁ノードが説明されています。
Numeric Row Splitterは値選択式に機能特化されています。
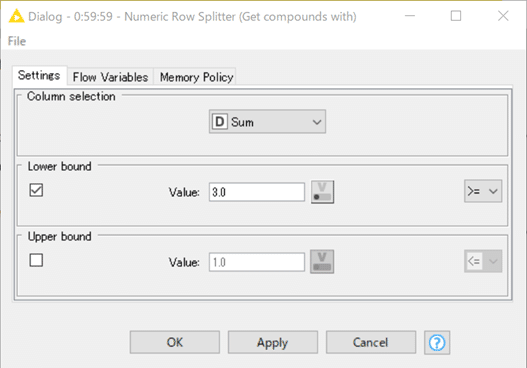
結果は以下の通りです。
ノードを右クリックして下のから二番目のData acceptedを選択します。
4511化合物へ絞られました。

【Ro5でフィルタリングされる化合物の例】
じゃあどんな化合物が除去されたかも見てみます。
ノードを右クリックして一番下のData discardedを選択します。
904化合物を分子量順でソートして表示してみますと、903化合物は分子量500.0を上回って減点されていますね。分子量のクライテリアは比較的よく引っかかる重要な指標と考えられます。

だからこそ、先ほどは判断基準のもととなるExactMWの算出条件が気になったのです。詳しくはまた別の機会に。
次はStep3のデータ可視化へ進めます。
いいなと思ったら応援しよう!

