
【W3】部分構造による化合物フィルタリング_04_Step2_カスタム編
【本パート(W3)の目的】
いくつか私たちのスクリーニングライブラリーに含めたくない部分構造があります。このトークトリアルでは、そのような好ましくない部分構造の様々なタイプを学び、そしてRDKitを使ってそれらの部分構造を見つけ、ハイライトする方法を学びます。
上記はPython版のT3の説明ですが、W3の目的も同じです。
前回までにPAINSとBRENKでのフィルタリングを体験しました。
今回は忌避構造リストをカスタマイズして化合物選抜する手法を学びます。
【Step2の仕様確認】
Step2の上部では既存のRDKit Molecule Catalog Filterノードに収載されているBrenkらの忌避構造リストを用いてフィルタリングしました。
下部はBy handすなわち自分の手で忌避構造リストを作成して部分構造検索します。

【忌避構造リストの作り方】
デモサンプルとして、BRENKらのcsv形式のリストを利用しています。
出典はオリジナルの文献
Brenk et al.: 好ましくない構造のSMARTS定義 (Chem. Med. Chem. (2008), 3,435-444 のSupporting InformationのTable 1
です。
上記の内容をcsv形式に直して利用しています。
もし自分でもカスタマイズしたい場合は、下記のようにcsvファイルを作成してください(形式は name-space-SMARTS です)。
左にname(忌避構造の名前)
半角スペース
右にそのSMARTS表記
の並びです。下図ではSMARTS部分に私が紫色に着色しました。

ところで皆さんはSMARTSでサクサクと検索式をかける方ですか?私は無理です。
SMILES Arbitrary Target Specification (SMARTS)というものがあります。Daylight社によりルールが定められており、Substructure Searchなどの構造検索に利用されます。
本格派の方には本家のサイトを見ていただくとして、
教材としてはやはりmagattacaさんのブログかと。
すっごく楽しそう!でもこちらも本格派だった。初学者向けがあったら教えてください。
【SMARTSのリストを前処理】

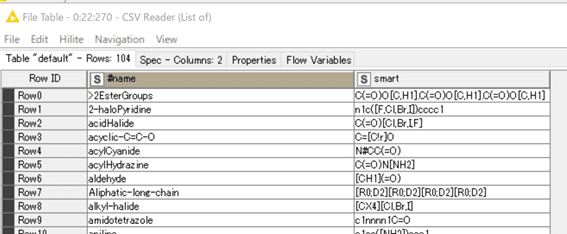
【CSV Reader】
まっきーさんの説明も参考にしつつ、今回のcsvファイルを読み込みます。
注意点としては今回はスペース区切りなので、

Column delimiter欄に半角スペース” “が入っています。分かりにくいです。
結果:104のルール(忌避構造の検索式)が文字列データとして読み込まれました。
続いての【Molecule Type Cast】でSMARTS形式に変換します。

結果は

構造クエリーなので、構造式で表示されていてわかりやすくなりました。
そして【RDKit from Molecule】でRDKit形式に変換しています。

結果は

一部描画は出来てないですが、RDKit形式のカラムが追加されました。
ここまでがRDKitノードでの構造検索前のデータ前処理となります。
ちょっと大変でしたね。次回はおまけでSMARTSを自分では書かず、構造式を列挙して検索する方法も紹介します。
【RDKit Molecule Substructure Filter】

設定:
RDKit形式の化学構造データである“SmilesValue(RDKit Mol)”をSMART形式の検索式群”smart”で検索し、少なくとも1つ検索式にヒットしたら”Matched Substructs”というカラムにどの検索式にヒットしたかをRowIDのリストで出力すると言う設定です。

結果:
2つある出力ポートの上が、今回の場合は忌避構造検索に引っかかった化合物群です。

2348化合物がフィルターに引っ掛かったとわかります。
自分でcsvファイルを使ってBRENKフィルターを設定してフィルタリングできたわけです。
本来はここで完了です。ただし、今回非常に細かいことですがStep2の上部と比べて1件足りないんです。本質的ではないんですが、カスタムの仕方をより深く知るにはいい例かもと思ったので次々回にデバッグをしてみることにしました。
後半へ続く。
いいなと思ったら応援しよう!

