
【KNIME】化合物の構造データを前処理してみよう
前回は構造データを前処理することの大切さなど情報共有しました。
今回は実際にTeachOpenCADDのデモデータで塩、溶媒の除去すなわちフリー体への変換(フリー化、脱塩処理ともいいます)を体験してみます。
<参考>
【TeachOpenCADDのデモデータ】
対象として使うのはW1で収集してきたデータです。
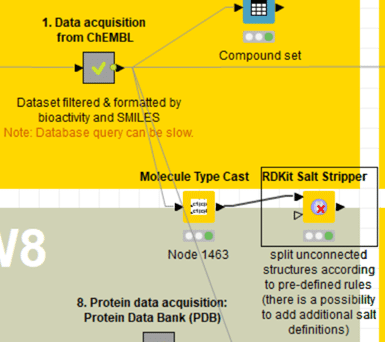
Molecule Type Castノードの次に上図のようにRDKit Salt Stripperノードを繋ぎます。
【RDKit Salt Stripper】
化合物の前処理用にノード群の一つです。
設定:
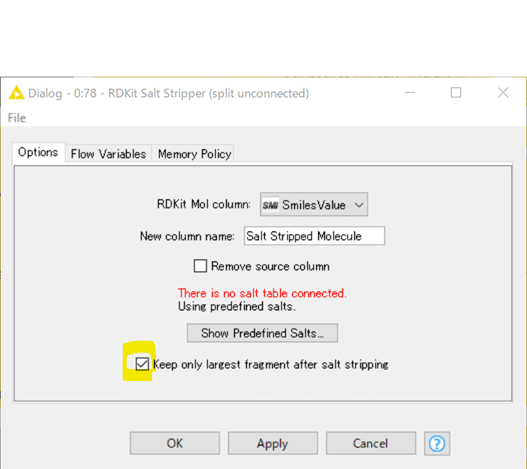
もし複数のフラグメントが含まれるデータがあったら、最大のフラグメント1つのみにする設定にチェックを入れました。
結果:
以下は変換された分子の一例です。5415化合物中169化合物が処理されました。
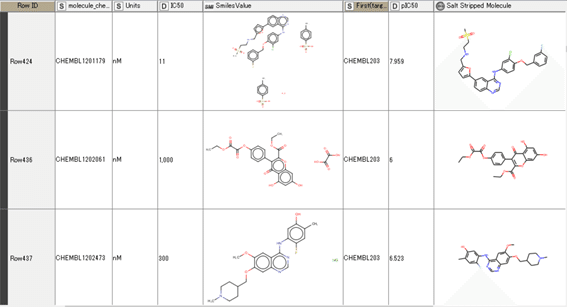
塩酸塩だけではなくて、トシル酸塩、シュウ酸塩などもフリー化されているのがわかります。フリー化処理をしないで算出した分子量でフィルターすることの問題点が分かっていただけるかと思います。
【初期設定の確認】
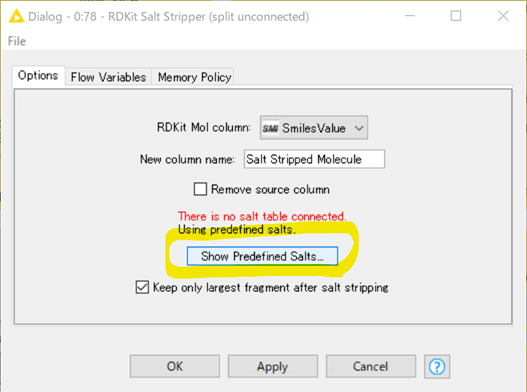
どんな分子を塩と認識するかの詳しい初期設定は設定の時にShow Predefined Salts..ボタンを押すと下記のように表示されます。
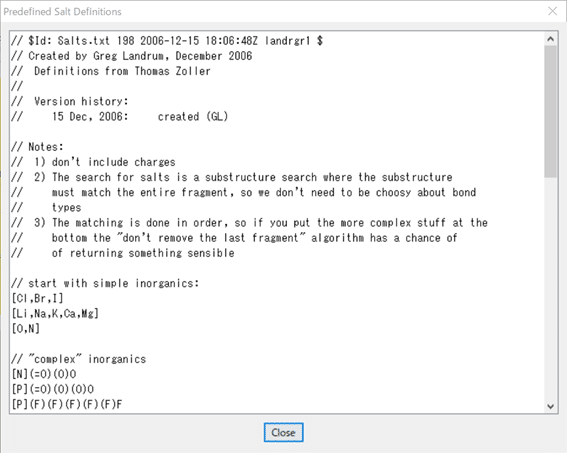
Gergが2006年に下記の通り設定してくれています。
// start with simple inorganics:
[Cl,Br,I]
[Li,Na,K,Ca,Mg]
[O,N]
// "complex" inorganics
[N](=O)(O)O
[P](=O)(O)(O)O
[P](F)(F)(F)(F)(F)F
[S](=O)(=O)(O)O
[CH3][S](=O)(=O)(O)
c1cc([CH3])ccc1[S](=O)(=O)(O) p-Toluene sulfonate
// organics
[CH3]C(=O)O Acetic acid
FC(F)(F)C(=O)O TFA
OC(=O)C=CC(=O)O Fumarate/Maleate
OC(=O)C(=O)O Oxalate
OC(=O)C(O)C(O)C(=O)O Tartrate
C1CCCCC1[NH]C1CCCCC1 Dicylcohexylammonium
医薬品の塩形成によく使われる酸や塩基はかなり網羅されています。さすが抜かりないなぁと感心しました。
<参考>「塩選択の現状分析」
余談ですが私が一番驚いたデータは下記でした
元データは側鎖が一炭素異なる2分子の混合物でした。
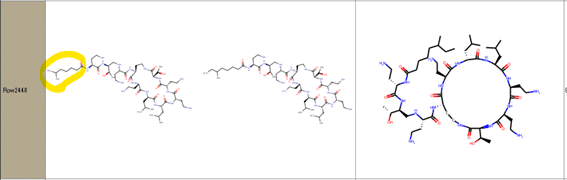
Ro5でのフィルタリングに関していえば、このデータは前処理があってもなくても結果に影響はなかったです。
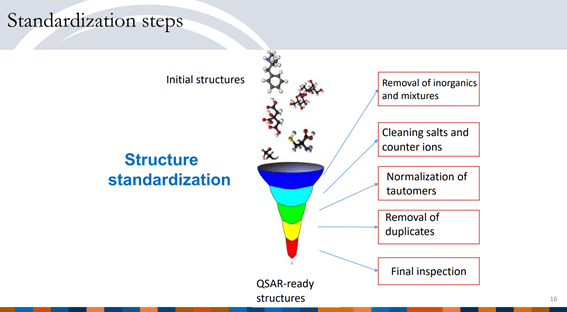
【化学構造式データのKNIMEでの前処理技術紹介】
さて、フリー化だけしか体験しませんでしたが、本当のところ
複数のソフトウェアによって生成されたバーチャルライブラリーや、外部の組織より入手した構造情報に関してとりわけ構造式表記の違いに起因する問題 が起こりがちです。また、ケミストによって塩の表記や短縮表記の使い方が異なる場合があります。
突き詰めると本当に多様な課題があることは前回から紹介している下記のサイトに記載の通りです。
JChemとJChem Extensionsを使えば、KNIMEで完全実装できるんですが、有償ですし素人が手を出せるものではないかとは思います。
<参考>
塩、溶媒の除去だけなら無償トライアル可能なMolConverterノードでもできます。
設定例:
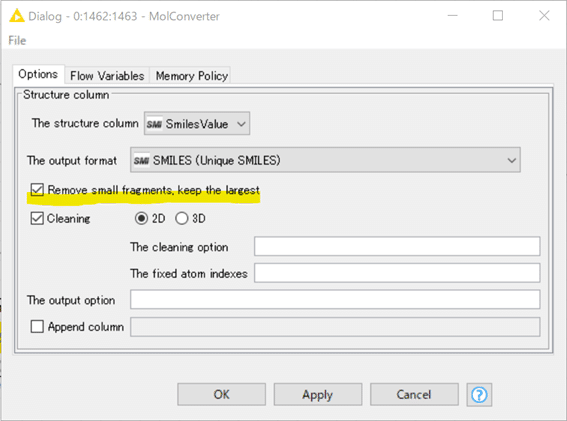
簡単ですね。
Cleaning機能もカスタマイズするともう少し正規化できるかもしれないですが今日はここまで。
【猛者紹介】
実はKNIMEの無償ノード群でかなりのレベルで実装に挑まれている猛者がいました。その熱意を賞賛しつつ紹介しておきますね。
以下公開されているPDFから抜粋させていただきます。

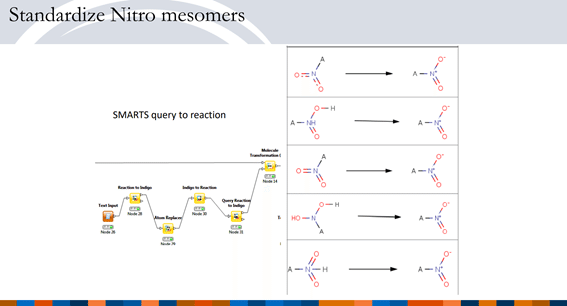
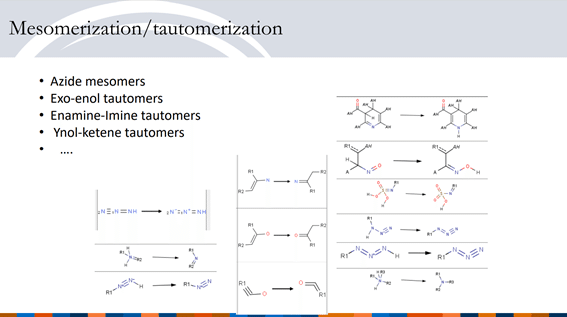
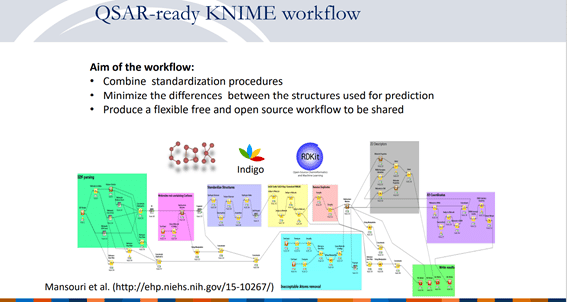
更にもう一報、こちらのデータインテグレーションもかなりのものでした。
まだ眺めただけでコメントできそうにないですが、RDKitノード群も大いに活用されています。
よっしゃ、今日はこれくらいにしといたるわ!
いいなと思ったら応援しよう!

