
【W2】指標による化合物フィルタリング_02_Step1
【本パート(W2)の目的】
W1でChEMBLから取得した化合物群をルールオブファイブ(Ro5)のクライテリアに基づきChEMBLから集めた化合物をフィルタリングします。
薬らしくない分子を取り除く手法の一例を学ぶことが目的です。
(原典)
【W2の中身を見てみよう】
今回扱うパートを具体的に見ていきます。
「2. Molecular filtering: ADME and lead-likeness criteria」メタノード
を開きます。下記の3stepsのうち、
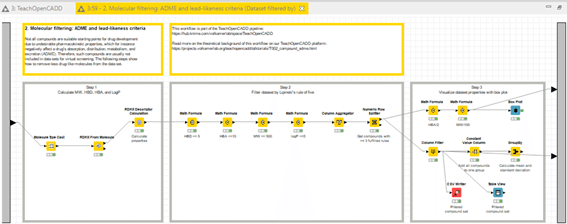
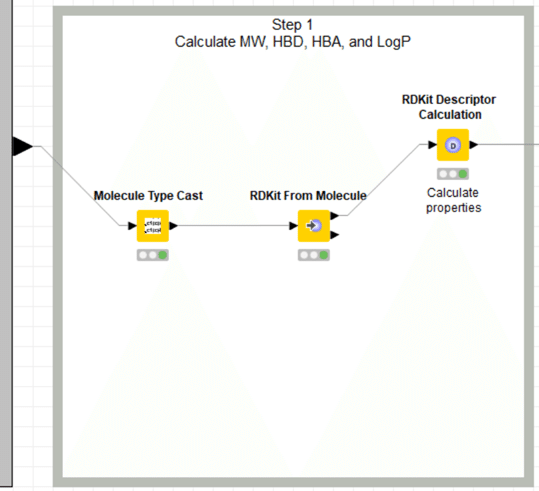
Step1: Calculate MW, HBD, HBA, and LogP
を今回扱います。
入力されるデータに関しては過去の記事を見てください。
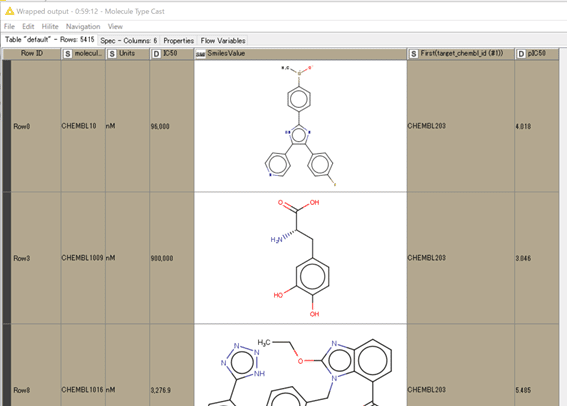
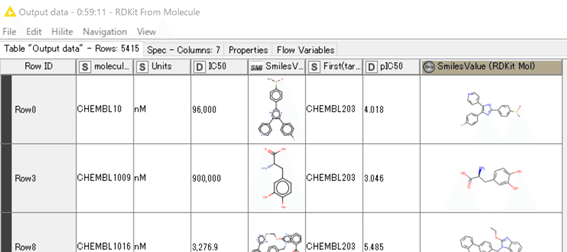
今回は下記のデモデータを用います。一部表示しておきます。
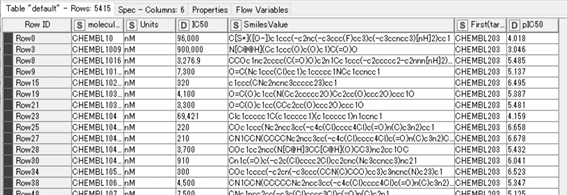
EGFR(target_chembl_id = 203)に関してのIC50値(nM換算)とpIC50値の5415化合物データです。
【RDKitについて】
先達の優れた記事やマニュアルがありますので、あらためて解説する言葉があるわけではないです。
もちろん皆さんご存じのPy4Chemoinformaticsから引用いたします。
開発者のGreg Landrum氏いわく
RDKitはケモインフォマティクスにおけるSwiss Army Knifeであり、様々な機能ピースの集合体である
化合物情報の読み込み、書き込みに始まり、構造の描画、3次元構造配座発生、Rグループ分解、記述子、フィンガープリント計算、ファーマコフォア算出などなど、挙げればきりがないほどの機能が実装されておる。解析から可視化まで幅広い範囲をカバーできるのだ。
語尾が気になった方などぜひ原本を参照ください。
KNIMEではどれだけノードがあるかを見てもらった方が早いと思ったので、以下にスクショを貼り付けておきます。
<参考>
英語のサイトしか見つかりませんでした。
本格派の方はこういったサイトを熟読されることをお勧めしたいです。
「KNIMEでRDKitをはじめよう」コーナーは今後もつくられないですよね、きっと。
【Molecule Type Cast】
このノードは一度紹介していますのですさんの記事を再引用しておきます。
このノードを通すと、stringになっている構造式データを"構造として扱える"形式に変換してくれます。
結果:
SmartsValueカラムは「S」(文字列)ではなく「SMI」(Smiles)と認識されるように変わったので、構造式として表示できるようになりました。
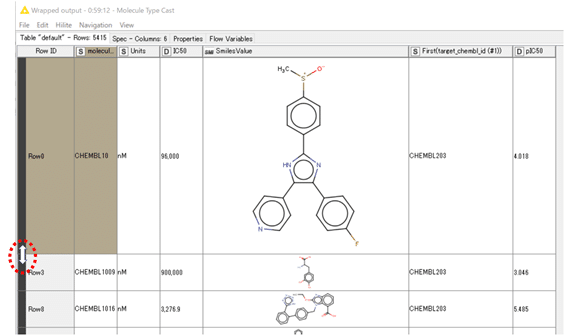
ほとんどの場合、セルが小さく表示されて見にくいので、図で赤丸で囲ったあたりをドラッグして上下を拡げるとか、左右も適度に拡げるとかしてください。
意外に最初は皆さんまごつかれるのですが、全ての行を選択しておいてShiftキーを押しながらどれか一つの行の上下幅を変えると、その高さ設定が全行に反映されます(わからん!という方、twitterにDMいただけたらもう少し頑張って説明します)。
SMILESとして認識させたら、いよいよRDKitノード群でデータ処理していきましょう。
【RDKit from Molecule】
分子の読み込みをします。
それだけのはずなんですが、
ノードディスクリプションを見ると思った以上に多機能です。
KNIME AP ver.4.4系は日本語化パッチを準備中だそうなので、自分で意訳します。
以前より私はインフォコムの日本語化プロジェクトを応援しています!(利己的な気持ち混じりですが)ver.4.4系対応を楽しみにしております。
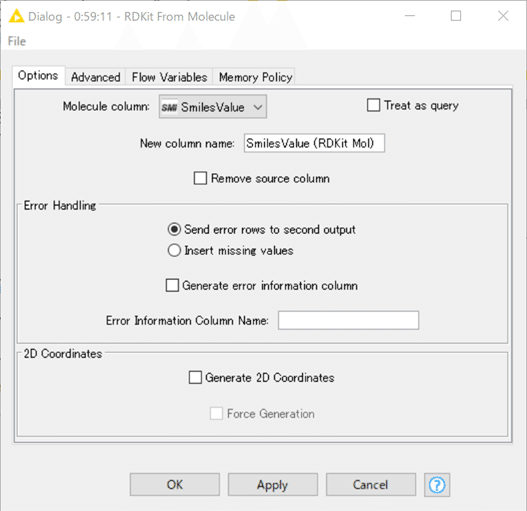
String型の化合物構造データ、つまりSMILESやMOLやSMARTSの形式のデータを変換して
RDKit moleculeというデータ型のカラムをデータテーブルへ新たに追加します。
グレーアウトしてるオプションメニューはチェックを入れても適用されないです。
Optionsを見ると私が使ったことのない機能がいっぱいありました。割愛しますすみません。まっきーさんケモインフォにも興味持ってくれないかなぁ。
さて今回の設定はデフォルトのままでした。
結果は一番右にRDKit molecule カラムが加わっています。
【RDKit Descriptor Calculation】
RDKitでの記述子計算をするノードです。記述子計算に関しての説明と言えばこちらがオススメです。
既にケモインフォマティクスにおける記述子をまとめた分厚い本ができあがるほどに多くの種類がありますので,開発された記述子の全てを覚えることは不可能ですし,意味もないでしょう.合成化学者としては,LogPやTPSAなどのよく使うものについて理解を深めておくことが大切だと思います
と合成化学者向けにやさしく書いて下さりつつも、がっつりPython使うところが素敵です。
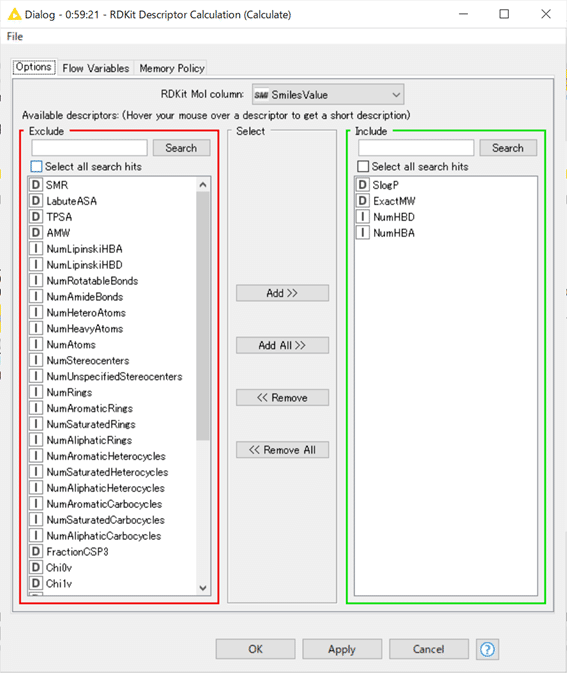
実際にRDKit Descriptor Calculationノードで、45種の記述子計算を選択できるのですが、各項目についてまで解説されたサイトは見つけられませんでした。
"Getting Started with the RDKit in Python"
の下記リンク先に情報はあるものの、網羅的な説明は断念します。参考文献なども掲載されていますので深く知りたい方はどうぞ。
今回の設定は
結果として
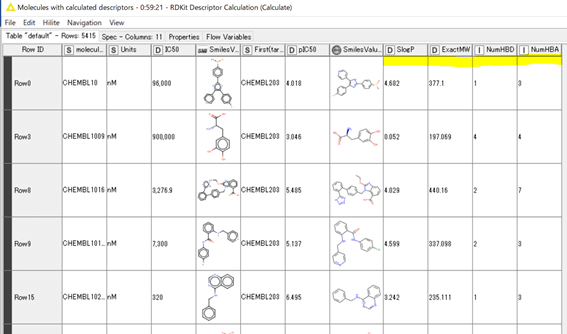
Ro5に必要な4つの化学計算の結果が新たなカラムとして追加されました。
• SlogP: LogP (オクタノールー水 分配係数) 計算値
• ExactMW: 分子量
• NumHBD: 水素結合アクセプターの数
• NumHBA: 水素結合アクセプターの数
次のStep2ではRo5での判定を行います。
いいなと思ったら応援しよう!

