
【JKI】012_Obtaining_a_List_of_Human_Genes_via_REST_02_REST処理
【JKI_012】課題を再確認
課題確認は前回で一旦終えて、自分の解答であるKNIME workflow(WF)の説明をさせていただこうと思います。
後でも話しますが、WFは一ヶ所間違いがあったので今は修正版を上げました。私が課題文をよく見ていなかっただけのケアレスミスです。
【入力ファイルを確認】
ダウンロードした「list_of_hormones_example.csv」ファイルはいつものようにWF内のdataフォルダに格納したのち、相対パスで読み取り設定しました。
上記のデータセットの存在を見落として、自分でTable Creatorで好きなホルモン名を入れられるようにしてしまっていたので、下記のCSV Readerに差し替えたのが修正版です。
設定:

結果:

上記の8種について検索すると言う課題です。
【今回のREST利用WF】

検索式(Query)のURLをString Manipulationで作り、GET Requestで検索を実行、得られたJSONデータから必要なデータ種を選んでJSON Pathでリスト形式にしたら、Ungroupでテーブル形式に展開。
と聞くと流れとしてはわかりやすくないですか?
もう一回書くと、検索式を立てて、検索し、結果がJSONって特殊な形なんで変換してます。
でもやっぱり具体的にデータを追いながら見ないとピンと来ないかなと思います。
それぞれの設定と結果は以下の通りです。
【String Manipulation】
設定:

結果:

【GET Request】
設定:

Delayは大事だそうですので気を付けましょう。
大量のリクエストを短時間に送信してしまうと、相手サーバーに負荷がかかってしまい、サイバー攻撃をしているようになってしまいます。
一歩間違えると犯罪になってしまうんです。興味がある方は「Librahack事件」で調べてみてください。
そこで、遅延処理を入れることが必須となっています。単位が「ms」なので、今回は2秒の間隔を意図的に空けるために「2000 ms」にしました。
もう技術説明はまっきーさんにお任せで安心です。
結果:

JSON形式データも冷静に各行追っていくと文法があってあー欲しいデータは入ってるなぁってわかりますよね。
次のデータを変形して取り出す設定はちょっと手間でした。
【JSON Path】
JSON Pathは
JSON オブジェクトの要素をクエリする標準化された方法です。JSONPath はパス式を使用して、JSON ドキュメントの要素、入れ子要素、配列に移動します。
とのことです。
と言っても私はクエリを自分で正しく書ける自信はないのでこのノードを利用してクエリを作ります。方法は後述するので設定と結果を先に。
設定:

結果:

【JSON Pathをノーコードで発生させる】
設定の参考記事はこちらです。
今回だったら例えばtaxidを取ってきたいとしたら

Taxidのデータ部分をクリックして選んだ状態で「Add Colum query」を押した結果を上図に示しています。Output欄にTaxidのJSONPath一つ増えていますね。
ノーコードツールは便利です。
【Ungroup】
いきなりデータテーブルすなわち一覧表みたいな形式にする方法は私は知らないので、上記のように各カラムがリストになっているものをUngroupで展開しています。
_idsを例にすると
[8660, 3643, 3630, 3667, 10580, 6517, 3486, 2888, 3651, 64599]
を展開したら
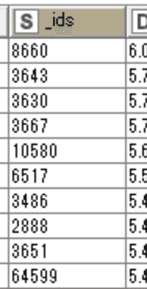
みたいになるわけです。
設定:

結果:

ここまでで今回のREST利用WF部分の説明を終えました。
【データを選んで出力】
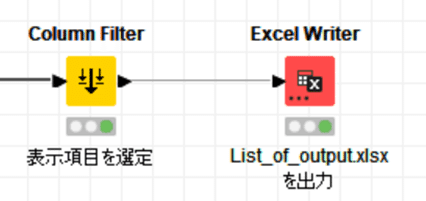
必要なカラムを選んでみんな大好きなExcelに書き出す仕様にしました。
一応設定画面は以下の通り。


【最後の問題の解き方で迷う】
また、リクエストがJSONではなくXMLで応答を返すこともあります。XMLレスポンスもパースする方法を含めるにはどうしたらよいでしょうか?
の正しい解き方がわからなくてひねり出したWFは下図の通り。
JSONで出たレスポンスをXMLに翻訳して、仮のXMLでの応答とみなし、XPathでパースしてみました。
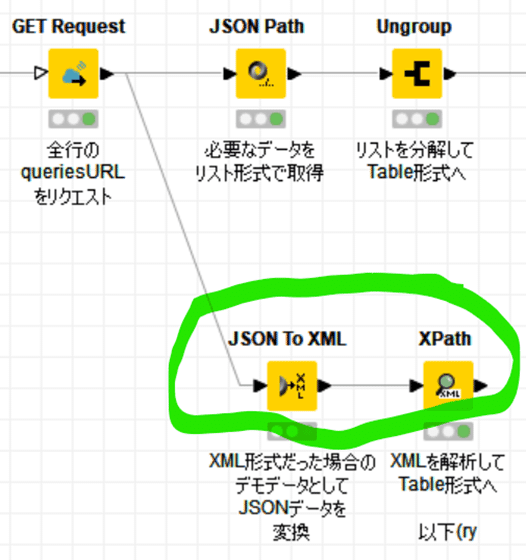
前回にたくさん紹介したブログでも多用されていますよね。
XPathの説明はこちらで決まりでしょう。
設定と結果は下記の通り。
JSON To XML設定:

結果:

XPath設定:

結果:
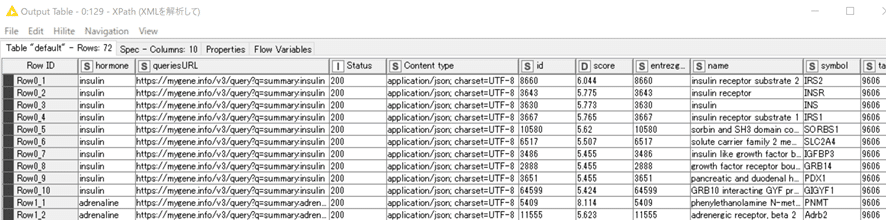
以下はJSONからのパースと同様なので略しました。以上で解答は完了とします。

おまけ:
【覆水盆に返らず】
WFは文頭にある通り提出してあります。今日この記事を書いていてWFを訂正したのですが、おひとり先にダウンロードされていた方、「なんじゃこりゃぁ」って思いましたよね。すみません。
てっきり好きなホルモンを入力していいのかと
個人的に思い入れのある
コルチゾール
と
マキシマム ザ ホルモン
を仮入力していました。
この場を借りてお詫びしておきます。
次回:
記事を読んでいただきありがとうございます。 先人の智慧をお借りしつつ、みなさんに役立つ情報が届けられたらと願っています。 もしサポートいただけるなら、そのお金はKNIMEの無料勉強会の開催資金に充てようと思います。
