
【W8】タンパク質データの取得_10_Step2_07_最大リガンド
【W8の目的】
(i) PDBデータベースからEGFRの全てのPDB IDを取得し、
(ii) X線結晶構造解析による構造で、最も質の良い4つのタンパク質ーリガンド複合体構造を取得して保存します。
Python版はより発展的です。
【Process JSONメタノードまで再確認】
デモデータを使ってW8のStep2を学んでいます。上記の(ii)を実行中。
Process JSONメタノードの中身を見終わりました。

出力は下記のようなデータテーブルです。

今回もETLのTすなわちデータ変換(Transform)の続きですが、リガンド情報に注目してさらにデータを絞り込みます。
【リガンドの選抜条件】
前にも例を挙げて紹介しましたが、1つのPDBデータにリガンドとして登録されている分子は一種とは限らないですし、しかも単なる塩化物イオンなんかもあります。
一方で、私たちが共結晶の構造解析結果を見たいデータはリガンドとして結合している有機化合物でしょう。そこで下記のKNIME workflow(WF)では、分子量100以上でかつ各PDBデータの最大分子量の化合物が目的のリガンドであろうと想定してデータを絞り込んでいます。

しかもこの工程ですが、
This step is needed for next node where each
PDB ID is filtered for largest, i.e. first, ligand.
との注釈が付いています。次の工程 Step3は下図の通り。
「最も質の良い4つのタンパク質ーリガンド複合体構造」すなわち解像度順でTop 4に絞って共結晶のデータを取得するので、そのためのデータ前処理をここまでしているというわけです。

【Step2の最終工程体験】

上記の4ノードはこれまでに何度も出たものですから、もう設定情報と結果だけ見ていきます。
【RowFilter】分子量100以下のデータは除去
設定:
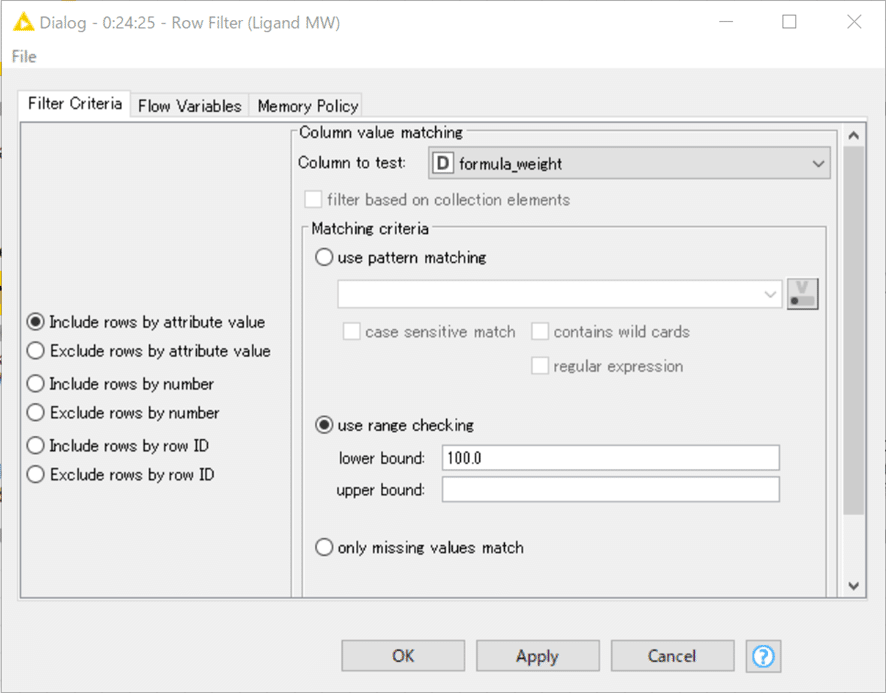
結果:

【Sorter】分子量が大きい順に並べ替え
設定:
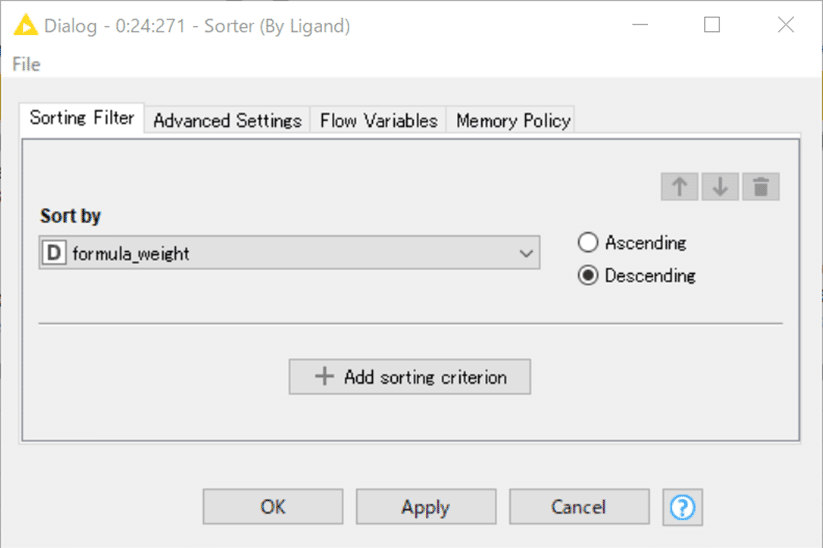
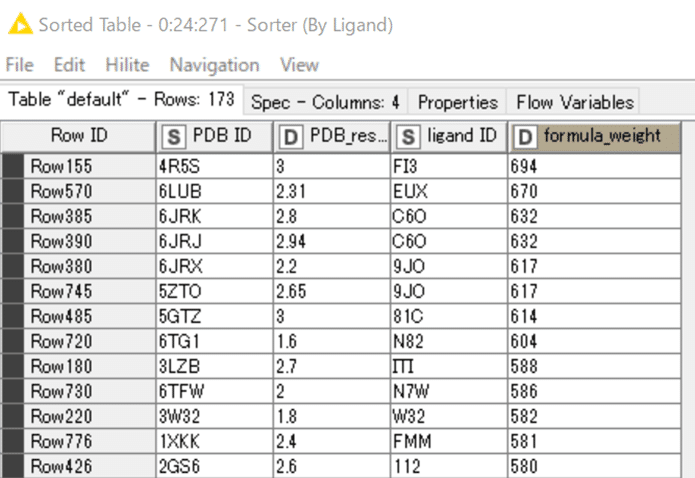
結果:
【GroupBy】各PDB_IDごとに最大分子量の行を残す
設定:


結果:151のPDB_IDそれぞれの最大リガンドのデータが得られる

【Sorter】解像度順に並べ替え
設定:
結果:
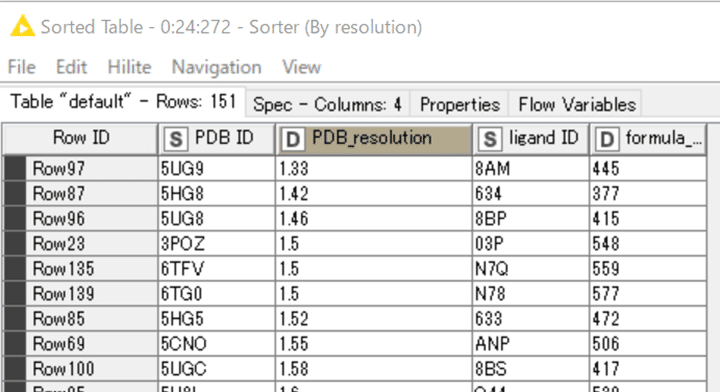
最も解像度が高いPDBデータは5UG9で1.33Åと本当に高い解像度ですね。
この後上位4つに絞るのですが、同スコアで4位が3つあるうち、3POZだけが残りますね。
PDBからデータが入手できた順(Row ID)に並ぶので、PDB_IDの小さいデータすなわち大まかにいえばより古いPDBデータが上に来るのではと推測します。自動で絞り込んで見たいデータを失わないようちょっと注意が必要かもしれません。
ここまででStep2に関しての全てのノードを体験してきました。次回からは最終工程Step3に進みます。
おまけ:
【Step1での不具合情報振り返り】

Step1で実はKNIMEのextensionsのバージョンが古いと使えない機能があると話しました。
そして回避策の一つとしてQueryのうち下図でマークした2つの条件を削除して実行すればいいと説明しました。

Step1では絞り込めていなくても、今回紹介した工程で
Has ligand with MW >= 100
のデータに絞り込めます。
すなわち「Step1でレコード数は増えても、この後体験するStep2ではリガンドのあるなしやその分子量でのフィルタリングを行うので最終結果は同じになります。」と先日紹介した通りになっています。
子曰く、過って改めざる。これを過ちという。
ちゃんと改められて一安心しました。
いいなと思ったら応援しよう!

