
【W3】部分構造による化合物フィルタリング_01_概要
【本パート(W3)の目的】
いくつか私たちのスクリーニングライブラリーに含めたくない部分構造があります。このトークトリアルでは、そのような好ましくない部分構造の様々なタイプを学び、そしてRDKitを使ってそれらの部分構造を見つけ、ハイライトする方法を学びます。
上記はPython版のT3の説明ですが、W3の目的も同じです。
【好ましくない部分構造】
部分構造には好ましくないものがあります。例えば毒性あるいは反応性があるといった理由や、薬物動態学的特性が好ましくないという理由、あるいは特定のアッセイに干渉する可能性が高いという理由です。 今日の医薬品探索ではよくハイスループットスクリーニング (HTS wikipedia) を実施します。好ましくない部分構造をフィルタリングすることで、より望ましいスクリーニングライブラリを構築することができます。これにより、スクリーニングの前にライブラリの数を減らすことができ、時間と資源の節約につながります。
今回はBrenkならびにPAINSを化合物リストから除去します。
BrenkとPAINSについて、私が新たにコメントすべきことはないです。
上記で引用したmagattacaさんのブログを参照ください。わかりやすい説明で、素晴らしいと思います。Dominique SydowさんをはじめとしたVolkamer研究所の方々とmagattacaさんのご尽力に感謝いたします。
RDKitのコミュニティにも感謝したいです。今後見ていきますが、BrenkとPAINSについて、私たちがKNIMEを利用してノーコードで化合物フィルタリングできる仕様となっています。
PAINSを部分構造検索で実装することもできます。また、BrenkらによるリストもすでにRDKitに実装されています。
正直な感想を書いたら、冒頭から謝辞のような内容となりました。
【今回用いる入力データ】
W1-2でChEMBLから入手し、Ro5に注目してフィルタリングした化合物群を対象とします。
TeachOpenCADD-KNIMEのデモデータをそのまま用いますので、下記のような4510化合物です。

【3. Moleclar filtering: unwanted substructursメタノード】

W3の全ての化合物フィルタリングはメタノード内で実行されます。
ダブルクリックして中身を見ていきましょう。
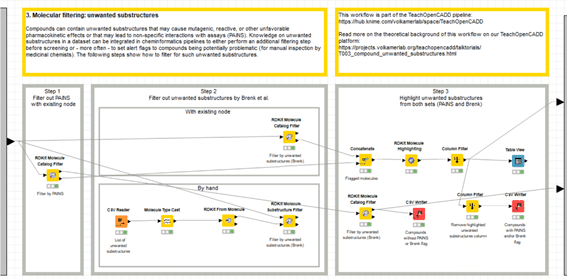
workflowが分岐、交錯しているので一見すると戸惑うかもしれないので、これまでと同様、Step毎などに分けて紹介していきます。
Step1: Filter out PAINS with existing node
既存ノードでPAINSフィルターをかける

Step2: Filter out unwanted substructures by Brenk et al.
Brenkフィルターをかける
上部: with existing node 既存ノードでのフィルタリング
下部: By hand独自のカスタマイズの例

Step3: Highlight unwanted substructures from both sets (PAINS and Brenk)
上部: PAINSとBrenkに抵触した化合物群を集計し、部分構造をハイライトする
下部: PAINSとBrenkに抵触しない化合物群(good molecules)

以上です。では次回はStep1から始めます。
いいなと思ったら応援しよう!

