
【JChem】ChemAxon/Infocomノード群を使ってみよう_05_JChemノード群logP編
【ChemAxonのlogP予測モデル】
JChemノード群、「JChem Extensions」を使えるようになって最初にしたかったのが、logP予測値算出です。
なぜなら下記の記事を読んだからです。
「logP予測精度の限界に挑む。SAMPL 6ブラインドチャレンジの結果」
このブラインドチャレンジにおけるCXNの結果は,選択されたリファレンスを上回るものであった。CXNは低い平均誤差(-0.025)と高いR2 (0.825)を示し,予測値にバイアスやオフセットが含まれていないことを示しています。
どのように精度の検証を行ったか詳しくは上記の記事をご覧いただくとして、CXNすなわちChemAxonのlogP予測値計算をつかってみたくなると思いませんか?
ChemAxon logP法は,Viswanadhanらが発表したatomic log P increments法に独自の大幅な拡張を加えて改良したものです。
<参考>
の
原子ごとの寄与を計算するアプローチ
分子を個々の原子にまで分解して,各々の寄与度の合計を計算することでLogPを推定するのがこのアプローチになります.
にあたると思います。
それにしても「化学の新しいカタチ」は素晴らしい。多くの方がブログなどでも引用されていますね。Dr Tomさんありがとうございます。
【JChem ExtensionsでのlogP予測値算出】
デモデータはTeachOpenCADD-KNIMEで入手します。
対象として使うのはW1で収集してきたデータです。
Molecule Type Castノードの次に下記のlogPノードを繋ぎます。

Workflowは下図のとおり

設定はちょっと悩みました。
ChemAxonでは、logP計算に1)Consensus、2)ChemAxonのモデルを選択することができ、それぞれいわゆるClogP, AlogPと見なせます。
Consensus : this method uses a consensus model built on the ChemAxon and Klopman et al. models and the PhysProp database.
ChemAxon : this method is based on ChemAxon's own logP model, which is based on the VG method (derived from Viswanadhan et al.).
SAMPL 6の結果に触発されて、我々は最近、この比較的小さいが正確に測定されたデータセットを用いてChemAxonのlog P予測ツールの精度を確認しました。ChemAxon logP法は,Viswanadhanらが発表したatomic log P increments法に独自の大幅な拡張を加えて改良したものです。
と先述の記事にあるので、今回は” ChemAxon”を選択してみました。

また、logP予測値以外に、Incrementsも表示する設定にしました。

結果:

Incrementsにもチェックを入れた結果、Molecule(logP)カラムも表示されています。
例としてCHEMBL10のIncrementsを見てみます。
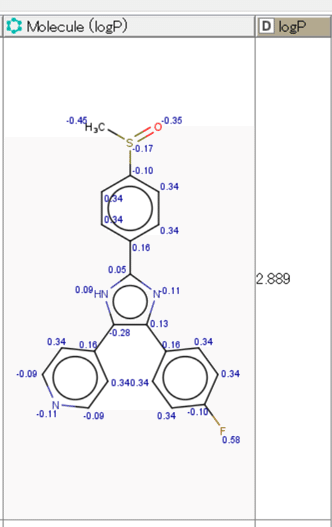
原子単位で、どの原子がlogPにどれだけ寄与したかを見ることができます。
ベンゼン環は脂溶性が上がりますね。
たまにBioisostereとなりうる
4-Pyridine基と4-F-Phenyl基がlogP的には大きく異なるのがよくわかります。
より具体的に言えば
4-Pyridine基: 0.16+0.34+(-0.09)+(-0.11)+(-0.09)+0.34=0.55
4-F-Phenyl基: {0.16+0.34+0.34+(-0.10)+0.34+0.34}+0.58=2.0
とlogPへの寄与度が1.45異なります。
<参考> Bioisostere
良く知られるBioisostere同士を比較して、脂溶性の変化を見るのもいいかな。
JChem Extensionsを使えばこういったデータを一気に発生して利用できます。
脂溶性を調節する化合物デザインをするときの参考になるかもしれませんね。
このように、精度よくlogP値を予測するだけでなく、その算出根拠まで一気に可視化して見せてくれると言うのが私は楽しいと思いました。
次回へ続く。
いいなと思ったら応援しよう!

