
Pythonライブラリ(AutoML):PyCaret
1.概要
PyCaretは多数のライブラリ・フレームワーク(機械学習:scikit-learn、XGBoostなど、パラメータ調整:Optunaなど、可視化:SHAPなど)のPythonラッパーです。
PyCaretを用いることで複数モデルでの学習・推論をまとめて比較でき最適な機械学習モデルの選定・学習が可能となります(AUTOML)。
【Documents】
●公式ページ:https://pycaret.org/
●API Reference:https://pycaret.readthedocs.io/en/latest/
2.環境構築
「PyCaretの難しさ=環境構築」と言っても過言ではないと思います。エラー防止のため本記事ではGoogle Colabを使用していきます。
ローカルPCで使用するなら仮想環境の作成は必須のため合わせて記事を追加しておきます。
2-1.全般
環境構築は公式を参照しており、注意点は下記の通りです。
【PyCaretの環境構築時の注意点】
●「pip install pycaret」でライブラリをインストールできますが、同時に複数の機械学習ライブラリをインストールします。よって自分のPCに直接インストールするとVersion違いによるエラーが出る可能性があります。
●フルバージョンのインポートは後ろに[full]をつける。
●公式は仮想環境下(Docker, venvなど)での実行を推奨している。
[通常]
pip install pycaret
[フルバージョン ※記事では不使用]
pip install pycaret[full]
2-2.Colab使用時の注意事項1:エラー対応
2022年5月では直接pip install pycaretするとsetup時にエラーが出ます。解決策としては①旧Versionの使用、②pycaretの前に所定のライブラリをversion指定でimportすることです。今回は②で対応しました。
またinterpret_model()用にshapやハイパーパラメー調整ライブラリも別途インストールが必要です(無くても特定処理以外は動きます)。
[Jupyterセル]
!pip install pandas-profiling==3.1.0
!pip install pycaret
!pip install shap
!pip install optuna
!pip install scikit-optimize
【未対応ライブラリ】
ハイパーパラメータ調整向けライブラリとしてtune-sklearnがありますがこちらは最新バージョンだとエラーが発生したため放置します。
[IN]
!pip install tune-sklearn ray[tune]
#上記後に処理
[OUT]
AttributeError: partially initialized module 'ray' has no attribute '_private' (most likely due to a circular import)2-3.Colab使用時の注意事項2:GPU利用時ライブラリ
公式より「LightGBMのようなライブラリでGPUを使用する場合は一度アンインストールして下記コマンドで再インストールせよ」とあります。
今回は特にGPUは使用しないため下記は実行しませんがご参考までに。
2-4.参考:YAMLファイル
私が普段使用している仮想環境のYAMLファイルは下記の通りです。Anacondaの記事に従って対応すれば環境の追加が可能です。
3.PyCaretの基礎メソッド
3-1.Version確認
PyCaretのVersion確認方法は下記の通りです。
[IN]
import pycaret
pycaret.__version__
[OUT]
2.3.103-2.Google Colabでの事前設定:enable_colab()
Google Colab使用時にenable_colab()を実行することでインタラクティブな表示をすることが可能になります。
[IN]
from pycaret.utils import enable_colab
enable_colab()
[OUT]
Colab mode enabled.3-3.モジュールインポート:from pycaret.X import *
機械学習は学習に応じて適したモデルがあります。PyCaretでは用途に応じて適した学習モデルをインポートする必要があります。

【機械学習一覧とモジュールインポート】
<教師あり学習>
●分類 :from pycaret.classification import *
●回帰 :from pycaret.regression import *
<教師なし学習>
●クラスタリング:from pycaret.clustering import *
●異常検出 :from pycaret.anomaly import *
●自然言語処理:from pycaret.nlp import *
●アソシエーション分析:from pycaret.arules import *
<時系列分析>※現在beta版らしいがそもそもインポートできなかった
●時系列分析:from pycaret.time_series import *
3-4.モデルの確認:models()
各モジュールのモデル(アルゴリズム)の確認はmodels()を実行します。なお5-1.の前処理(setup)前にmodels()を実行するとエラーが出ます。
<setup実行前>
[IN]
from pycaret.classification import * #分類
models()
[OUT]
NameError: name 'logger' is not defined<setup実行後>
[IN]
from pycaret.classification import * #分類
models()
[OUT]
下表参照
「A Complete Guide to PyCaret!!!」のそれぞれ下記の通りです。
<Classification:分類>
+------------+---------------------------------+
| ID | Name |
+------------+---------------------------------+
| ‘lr’ | Logistic Regression |
| ‘knn’ | K Nearest Neighbour |
| ‘nb’ | Naives Bayes |
| ‘dt’ | Decision Tree Classifier |
| ‘svm’ | SVM – Linear Kernel |
| ‘rbfsvm’ | SVM – Radial Kernel |
| ‘gpc’ | Gaussian Process Classifier |
| ‘mlp’ | Multi Level Perceptron |
| ‘ridge’ | Ridge Classifier |
| ‘rf’ | Random Forest Classifier |
| ‘qda’ | Quadratic Discriminant Analysis |
| ‘ada’ | Ada Boost Classifier |
| ‘gbc’ | Gradient Boosting Classifier |
| ‘lda’ | Linear Discriminant Analysis |
| ‘et’ | Extra Trees Classifier |
| ‘xgboost’ | Extreme Gradient Boosting |
| ‘lightgbm’ | Light Gradient Boosting |
| ‘catboost’ | CatBoost Classifier |
+------------+---------------------------------+
<Regression:回帰>
+------------+-----------------------------------+
| ID | Name |
+------------+-----------------------------------+
| ‘lr’ | Linear Regression |
| ‘lasso’ | Lasso Regression |
| ‘ridge’ | Ridge Regression |
| ‘en’ | Elastic Net |
| ‘lar’ | Least Angle Regression |
| ‘llar’ | Lasso Least Angle Regression |
| ‘omp’ | Orthogonal Matching Pursuit |
| ‘br’ | Bayesian Ridge |
| ‘ard’ | Automatic Relevance Determination |
| ‘par’ | Passive Aggressive Regressor |
| ‘ransac’ | Random Sample Consensus |
| ‘tr’ | TheilSen Regressor |
| ‘huber’ | Huber Regressor |
| ‘kr’ | Kernel Ridge |
| ‘svm’ | Support Vector Machine |
| ‘knn’ | K Neighbors Regressor |
| ‘dt’ | Decision Tree |
| ‘rf’ | Random Forest |
| ‘et’ | Extra Trees Regressor |
| ‘ada’ | AdaBoost Regressor |
| ‘gbr’ | Gradient Boosting Regressor |
| ‘mlp’ | Multi Level Perceptron |
| ‘xgboost’ | Extreme Gradient Boosting |
| ‘lightgbm’ | Light Gradient Boosting |
| ‘catboost’ | CatBoost Regressor |
+------------+-----------------------------------+
<Clustering:クラスタリング>
+-------------+----------------------------------+
| ID | Name |
+-------------+----------------------------------+
| ‘kmeans’ | K-Means Clustering |
| ‘ap’ | Affinity Propagation |
| ‘meanshift’ | Mean shift Clustering |
| ‘sc’ | Spectral Clustering |
| ‘hclust’ | Agglomerative Clustering |
| ‘dbscan’ | Density-Based Spatial Clustering |
| ‘optics’ | OPTICS Clustering |
| ‘birch’ | Birch Clustering |
| ‘kmodes’ | K-Modes Clustering |
+-------------+----------------------------------+
<Anoaly Detection:異常検出>
+-------------+-----------------------------------+
| ID | Name |
+-------------+-----------------------------------+
| ‘abod’ | Angle-base Outlier Detection |
| ‘iforest’ | Isolation Forest |
| ‘cluster’ | Clustering-Based Local Outlier |
| ‘cof’ | Connectivity-Based Outlier Factor |
| ‘histogram’ | Histogram-based Outlier Detection |
| ‘knn’ | k-Nearest Neighbors Detector |
| ‘lof’ | Local Outlier Factor |
| ‘svm’ | One-class SVM detector |
| ‘pca’ | Principal Component Analysis |
| ‘mcd’ | Minimum Covariance Determinant |
| ‘sod’ | Subspace Outlier Detection |
| ‘sos | Stochastic Outlier Selection |
+-------------+-----------------------------------+
<NLP:自然言語処理>
+-------+-----------------------------------+
| ID | Model |
+-------+-----------------------------------+
| ‘lda’ | Latent Dirichlet Allocation |
| ‘lsi’ | Latent Semantic Indexing |
| ‘hdp’ | Hierarchical Dirichlet Process |
| ‘rp’ | Random Projections |
| ‘nmf’ | Non-Negative Matrix Factorization |
+-------+-----------------------------------+3-5.指標の確認・追加:get_metrics()/add_metric()
学習時の評価指標を確認するにはget_metrics()を使用します(※setup後)。
[IN]
get_metrics()
[OUT]
また評価指標を追加したい場合はadd_metric()を使用します。
4.サンプルデータの読み込み:get_data()
PyCaretでは簡易のデータセットをget_data()で取得できます。
[IN]
from pycaret.datasets import get_data
print(get_data().shape) #データ形状
get_data() #データ型:pandas.core.frame.DataFrame
[OUT]
(56, 8)
引数としてDataset名を入れると指定したデータセットが取得できます。(PyCaretが前処理するためエンコーディングなどは不要)
[IN]
from pycaret.datasets import get_data
dataset = get_data('iris') #irisデータセットを取得
print(type(dataset))
print(dataset.shape)
[OUT]
<class 'pandas.core.frame.DataFrame'>
(150, 5)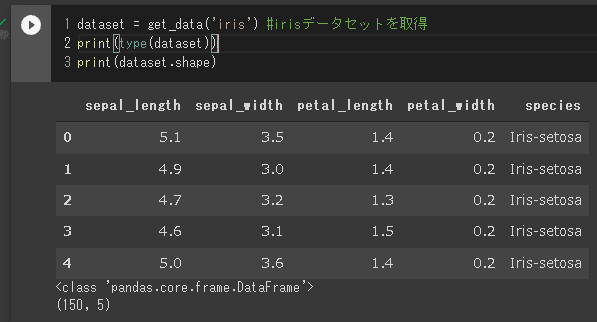
5.PyCaretによる機械学習
機械学習でのモデル訓練の大まかな流れは下記の通りです。PyCaretは複数モデルに関して2-5を一括で処理することが可能です。
【訓練の流れ】
1.データセットの準備
2.AIモデルの選定
3.目的関数(損失関数)の選定
4.最適化手法の選定
5.モデルの学習
5-1.前処理:setup()
機械学習では一般的に下記のような前処理を実施します。
【前処理の一例】
●欠損値処理:欠損値の削除、欠損値の穴埋め(平均値・代表値)
●カテゴリ変数の変換:エンコーディング(数値化)
●数値変数の変換:標準化・正規化、非線形変換
●次元削減・特徴量作成:PCA、クラスタリングなど
●学習・検証データの分割
5-1-1.setup()のパラメータ
PyCaretではsetup()を使用することで前処理を自動化できます。詳細は公式Docs参照のこと。
【必須パラメータ】
●data:学習・検証用データセット
●target:ラベル(教師データ)のカラム名
【任意パラメータ ※とりあえず使いそうなやつだけ記載】
●silent{default:False}:Trueだとデータ確認+Enterを押す作業を無くせます
●train_size {default:0.7}:学習用データの割合
●ignore_features:学習に使用しない特徴量(カラム名)を指定
●fix_imbalance:ラベルデータの数に偏りがある(例:正常データ1000に対して異常データ1)場合に、データセットのバランスを調整する
●use_gpu {default:False}:特定のライブラリでGPUの使用が可能となる
●log_experiment{default:False}:Trueにするとget_logs()でsetupの処理情報を取得できる
【注意点:fix_imbalance】
データ数が少ない時にパラメータの”fix_imbalance”を使用するとエラーは発生しないのですが次のcompare_models()実行時に出力がでません。また個別のモデルを実行すると下記エラーが発生します。
対策としてはパラメータを除外するかデータ数を増やす必要があります。
ValueError: Expected n_neighbors <= n_samples, but n_samples = 5, n_neighbors = 65-1-2.setup()の実行
コード実行後に下図画面が出るため、”Data Type”のデータ型を確認後「Enter」キーを押すと前処理が実行されます。
【setup()のOUTPUTから確認できること】
●Original Data(150, 5)に対してTransformed Train Set /Test Set が(104, 4), (46, 4)であり、データセットが「学習:テスト=70:30」に分割されている。
●Target(ラべル)のデータは文字列だったが自動でエンコーディングされている(Label Encoded):エンコーディング処理は不要。
●GPUの設定はしていないためUse GPU=False である
●一般的な前処理(データ分割、K-分割交差検証、エンコーディング、欠損値処理)はしてくれていそう。
●細かい前処理(正規化・標準化、特徴量作成、主成分分析など)は設定が必要そうである。
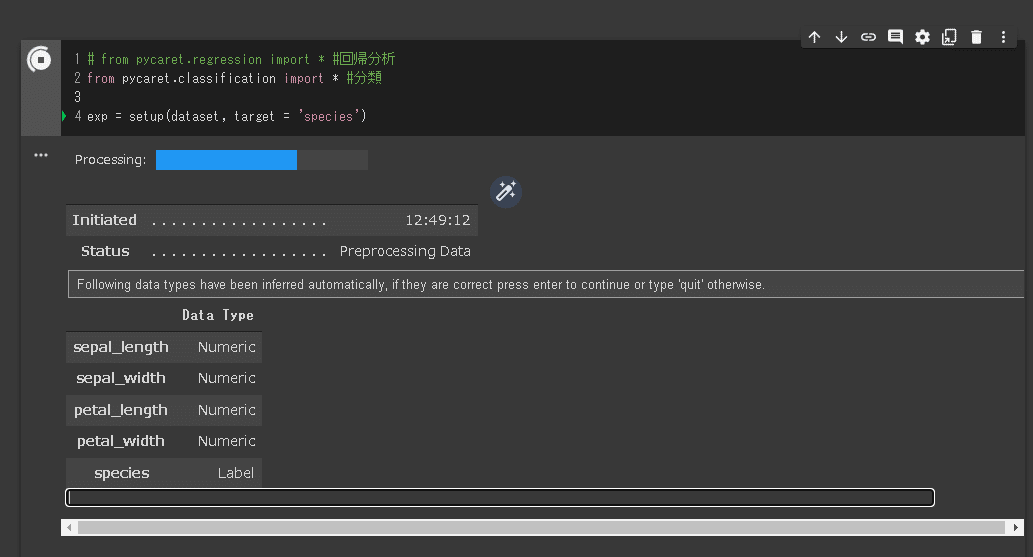
[IN]
# from pycaret.regression import * #回帰分析
from pycaret.classification import * #分類
exp = setup(dataset, target = 'species') #データの前処理
[OUT]
下表が自動で出力
なおexpの中にはTupleで様々な処理結果が入っております。
【expの中身確認 ※条件次第でなぜか変わるため参考までに】
●exp[18]:前処理後テストデータ
●exp[26]:前処理後学習データ
●exp[37]:前処理後データ(全データ=学習+検証)
●exp[39]:前処理後学習ラベル
5-2.モデルの学習:compare_models()
compare_models()で交差検証による複数モデルの性能比較をします。
[IN]
compare_models() #モデルの比較
[OUT]
実行後のcompare_models()には最高性能(精度)のモデルが入っており中身の確認もできます。
なおコンペや実際のデータで使用するとわかりますがランダム値があるため毎回ベストモデルは変わることがあるので1位だけでなく上位陣の複数モデルを検討する必要があります。
[IN]
best = compare_models() #モデルの比較
print(type(best))
print(best)
[OUT]
<class 'sklearn.discriminant_analysis.QuadraticDiscriminantAnalysis'>
QuadraticDiscriminantAnalysis(priors=None, reg_param=0.0,
store_covariance=False, tol=0.0001)5-3.モデルの評価:evaluate_model(best)
モデルの評価指標を確認するにはevaluate_model()を使用します。Jupyterで実行するとインタラクティブに指標を確認できます。
(※6章で紹介するplot_model(object, plot = '評価指標')でも確認できます)
[IN]
evaluate_model(best) #compare_models()での最高性能モデルを確認
[OUT]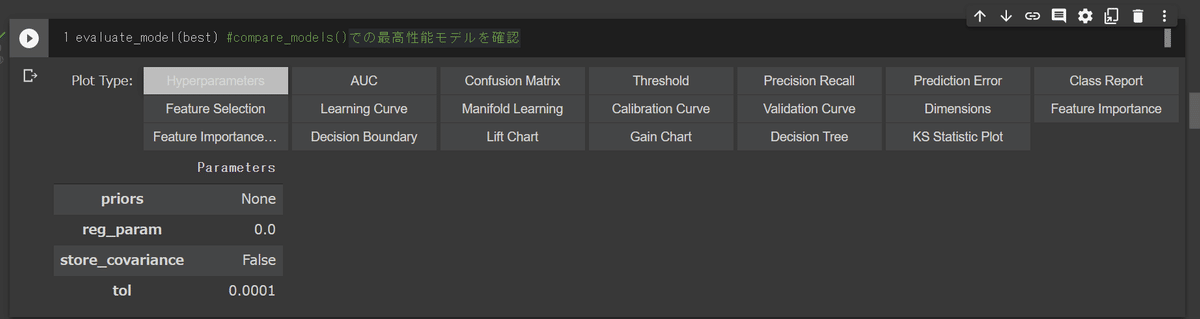

5-4.推論:predict_model()
学習済みモデルを用いて求めたいデータの予測(推論)を実施する場合はpredict_model() を実行します。今回はcompare_models()実行後に取得した最高性能モデルbestを使用します。
【predict_model()の引数】
●data:推論用データを渡す引数である。data=Noneの場合はsetup()時に作成されたテストデータを使用する。
●raw_score{defalut:False}:ラベル(教師データ)の各値の確率(Probability)を確認したい場合はTrueを渡す。
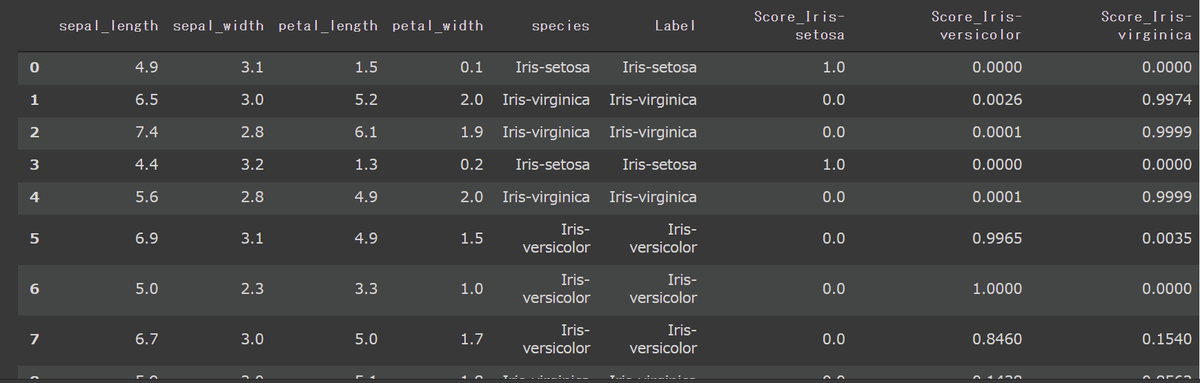
【predict_model()の要点】
●Scoreは教師データ(Labelカラム)の確率を示す
ー>「score=1は正しい正解を予想している」とは意味が違うので注意
ー>決定木の様に確率ではなく一意に正解を決めるモデルはScore=1となる(※上記の通り予想値が正解であることを保証はしていない)
●data=datasetの場合は処理時に表示されるAccuracyなどの評価指標が0となり異常である->テストデータでないと正しくでない??
[IN ※data=None]
df_pred = predict_model(best) #data=Noneのためsetup時のtestデータを使用
print(df_pred.shape)
display(df_pred)
df_pred_raw = predict_model(best, raw_score=True) #data=Noneのためsetup時のtestデータを使用
print(df_pred_raw.shape) #(46, 7)->テストデータと同じ数
display(df_pred_raw)
[OUT]
Model Accuracy AUC Recall Prec. F1 Kappa MCC
0 Quadratic Discriminant Analysis 1.0 1.0 1.0 1.0 1.0 1.0 1.0
(46, 7) #df_pred.shape
(46, 9) #df_pred_raw.shape
[IN ※dataを渡したとき]
df_pred = predict_model(best, data=dataset) #data=Noneのためえsetup時のtestデータを使用
print(df_pred.shape) #(150, 7)->datasetと同じ数
display(df_pred)
[OUT]
Model Accuracy AUC Recall Prec. F1 Kappa MCC
0 Quadratic Discriminant Analysis 0.0 0.9995 0 0 0 0 0
(150, 7)
6.モデルの最適化
compare_models()では自動で最高スコアのモデルが選択されますが、あくまで現状の前処理での結果となるため特徴量作成やハイパラ調整で性能が逆転することは十分にあり得ます。
本章では更に性能を追求するためにモデルの選択およびハイパーパラメーターの調整を説明します。
6-1.モデルの選択:create_model()
使用したいモデルを選定する場合は"create_model('compare_models()のindex')"を使用します。
今回は性能より説明しやすさを考慮して決定木'dt'を選択しました。
[IN]
dtree = create_model('dt') #'dt'=決定木
print(type(dtree)) #データ型
print(dtree) #モデルのハイパーパラメータ確認
[OUT]
<class 'sklearn.tree._classes.DecisionTreeClassifier'>
DecisionTreeClassifier(ccp_alpha=0.0, class_weight=None, criterion='gini',
max_depth=None, max_features=None, max_leaf_nodes=None,
min_impurity_decrease=0.0, min_impurity_split=None,
min_samples_leaf=1, min_samples_split=2,
min_weight_fraction_leaf=0.0, presort='deprecated',
random_state=635, splitter='best')
今まで学習したメソッドも同様の形で使用できます。
[IN]
evaluate_model(dtree)
df_pred = predict_model(dtree)
# df_pred = predict_model(dtree, raw_score=True)
df_pred
[OUT]
省略6-2.ハイパーパラメータ調整:tune_model(model)
学習モデル(compare_model()で取得したbestも使用可)のハイパーパラメータを調整するメソッドとしてtune_model()があります。
【ハイパーパラメータ調整】
●n_iter:学習時のイテレーションの回数設定
●optimize:評価指標の選択
●custom_grid:自分でハイパーパラメータの項目・値範囲を指定
●search_library/search_algorithm{default:RandomGridSearch}:最適化アルゴリズムの変更(scikit-optimize、optunaなど)
●return_tuner{defalut:False}:最適化アルゴリズムを確認できるよう変数出力
●choose_better{defalut:False}:tuned_modelで性能が改善されなかった場合は調整前の結果を選択する
引数としては各モデルのハイパーパラメータを調整できますが、各モデルの知識が必要となるため今回は引数無しで処理します。
[IN]
tuned_dtree = tune_model(dtree)
print(tuned_dtree) #モデルの確認
print(tuned_dtree.get_params) #ハイパーパラメータ確認
[OUT]
DecisionTreeClassifier(ccp_alpha=0.0, class_weight=None, criterion='entropy',
max_depth=8, max_features=1.0, max_leaf_nodes=None,
min_impurity_decrease=0.3, min_impurity_split=None,
min_samples_leaf=3, min_samples_split=5,
min_weight_fraction_leaf=0.0, presort='deprecated',
random_state=635, splitter='best')
<bound method BaseEstimator.get_params of DecisionTreeClassifier(ccp_alpha=0.0, class_weight=None, criterion='entropy',
max_depth=8, max_features=1.0, max_leaf_nodes=None,
min_impurity_decrease=0.3, min_impurity_split=None,
min_samples_leaf=3, min_samples_split=5,
min_weight_fraction_leaf=0.0, presort='deprecated',
random_state=635, splitter='best')>

結果を比較するために可視化します。次章のplot_model()でもできますが、今回はevaluate_model()を実行して混同行列(Confusion Matrix)で比較しました。
[IN]
evaluate_model(dtree)
evaluate_model(tuned_dtree)
[OUT]
結果として1データだけ正解できたデータが増えました。
7.モデルの可視化(Analyze)
機械学習はブラックボックス化しやすいため、説明のためには可視化が重要になります。PyCaretにはモデルの可視化メソッドがあります。
7-1.モデルのプロット:plot_model()
モデルの結果を可視化するためにplot_model(model, plot='評価指標')を実行します。plotの引数はモデルごとに異なるため公式Docsから確認できます。


plotが空の場合はdefalutの指標が出力されます(分類はauc)。
[IN]
plot_model(tuned_dtree) #plot_model(tuned_dtree, plot = 'auc')と同じ
[OUT]
【AUC(Area Under The ROC Curve)】
AUCは真陽性率(TPR)と偽陽性率(FPR)をプロットした曲線であるROC曲線の面積です。
$$
真陽性率(正例(Positive)データ中でのTPの割合)TPR = \frac{TP}{(TP + FN)}
$$
$$
偽陽性率(偽例(Negative)データ中でのFPの割合)FPR= \frac{FP}{(FP + TN)}
$$
【AUCの補足】
●ROC曲線はモデルの閾値(Probability)を移動させたときのTPRとFPRの推移を現したグラフである。
●真陽性率TPR=1.0の時はすべてのPositiveデータを正しく予想できている。(FPは完全に無視のためAccuracyが高いかは関係なし)
●偽陽性率FPR=0の時はすべてのNegativeデータを正しく予想できている。
(TPは完全に無視のためAccuracyが高いかは関係なし)
●モデルの精度が100%の時ヒストグラムはきれいに分離される(下図で赤と青色バーが重ならない)ため、モデル閾値(Probalitity)を移動させてもNegativeには影響しない(PとFが完全分離されているためFPR=0の時にTPRのみが動く)ため、AUCは(0, 1.0)の点を通るグラフとなる。


7-2.特徴量分析:plot_model(plot='feature')
特徴量はplot_model(model, plot='feature')で出力します。
[IN]
plot_model(tuned_dtree, plot='feature')
[OUT]
【参考:決定木の可視化との比較】
参考までにsklearnやdtreevizでの可視化も可能であることを確認します。
[IN]
import matplotlib.pyplot as plt
x = ['sepal length (cm)','sepal width (cm)','petal length (cm)','petal width (cm)']
y = tuned_dtree.feature_importances_ #特徴量の重要度
plt.barh(x, y)
[OUT]
dtreevizの可視化は「!pip install dtreeviz」でインポート後に下記コードで出力可能です。
[IN]
data = dataset[['sepal_length', 'sepal_width', 'petal_length', 'petal_width']]
target = dataset['species']
#下記エンコーディングは不要
id2col_label = {id:name for id, name in enumerate(target.unique())} #{0: 'Iris-setosa', 1: 'Iris-versicolor', 2: 'Iris-virginica'}
col2id_label = {name:id for id, name in id2col_label.items()} #{'Iris-setosa': 0, 'Iris-versicolor': 1, 'Iris-virginica': 2}
target = target.map(col2id_label) #speciesカラムのデータを数値に変換
from dtreeviz.trees import dtreeviz
viz = dtreeviz(
tuned_dtree, # 決定木モデル
data.values, #データ
target.values, #データラベル
target_name='species', #正解値のラベル
feature_names=['sepal_length', 'sepal_width', 'petal_length', 'petal_width'], #特徴量名
class_names= ['setosa', 'versicolor', 'virginica']
)
# viz.view() #ブラウザ上で表示
viz #Jupyter Notebook上で表示
[OUT]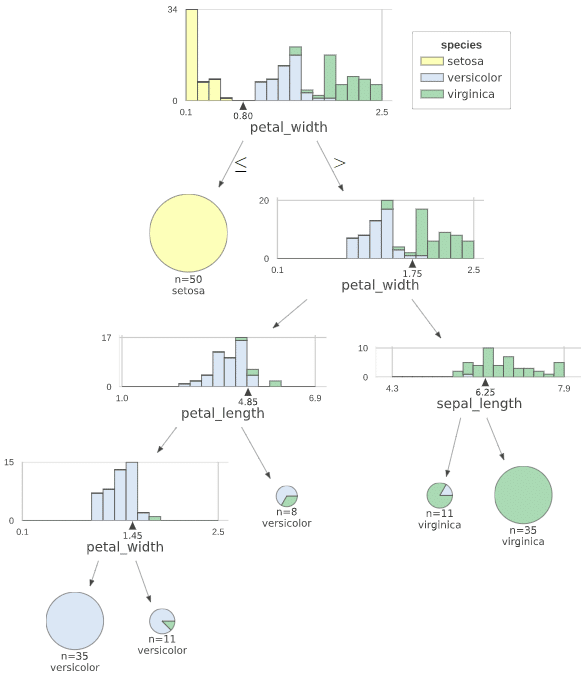
7-3.混同行列:plot_model(plot='confusion_matrix')
混合行列表示は下記の通りです。
[IN]
plot_model(tuned_dtree, plot='confusion_matrix')
[OUT]
7-4.モデル解釈:interpret_model()
機械学習モデルの可視化をするPythonライブラリのSHAPを用いてモデルの解釈図を出力できます(2-2節で説明の通り事前に要インポート)。

使用方法はinterpret_model(model)となります。
[IN]
interpret_model(tuned_dtree) #interpret_model(tuned_dtree, plot='summary')と同じ
[OUT]
引数のplotに値を渡すと異なる図を出力できます(公式Docs)。

[IN]
interpret_model(tuned_dtree, plot='reason')
interpret_model(tuned_dtree, plot='correlation')
[OUT]

8.モデルの保存・読み込み
8-1.モデルの保存:save_model(best, 'name')
作成したモデルを保存する場合はsave_model(model, 'filename')を実行すると名前に応じたPickleファイルが作成されます。
※Google Colabはランタイム切断時(Colab終了時)にクリアされるため保存したい場合はファイルを手動でダウンロードする必要があります。
[IN]
save_model(best, 'my_best_pipeline')
[OUT]
Transformation Pipeline and Model Successfully Saved
(Pipeline(memory=None,
steps=[('dtypes',
DataTypes_Auto_infer(categorical_features=[],
display_types=True, features_todrop=[],
id_columns=[],
ml_usecase='classification',
numerical_features=[], target='species',
time_features=[])),
('imputer',
Simple_Imputer(categorical_strategy='not_available',
fill_value_categorical=None,
fill_value_numerical=None,
numeric_stra...
('cluster_all', 'passthrough'),
('dummy', Dummify(target='species')),
('fix_perfect', Remove_100(target='species')),
('clean_names', Clean_Colum_Names()),
('feature_select', 'passthrough'), ('fix_multi', 'passthrough'),
('dfs', 'passthrough'), ('pca', 'passthrough'),
['trained_model',
QuadraticDiscriminantAnalysis(priors=None, reg_param=0.0,
store_covariance=False,
tol=0.0001)]],
verbose=False), 'my_best_pipeline.pkl')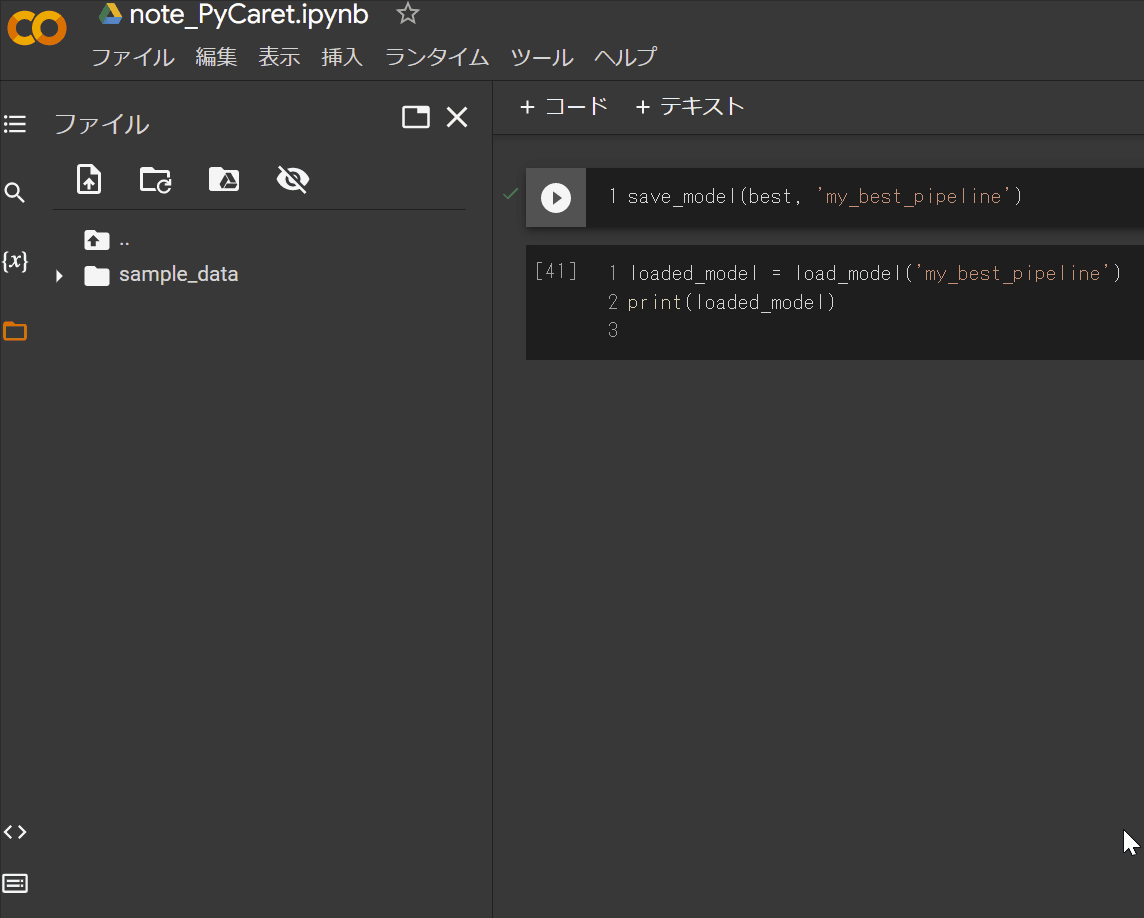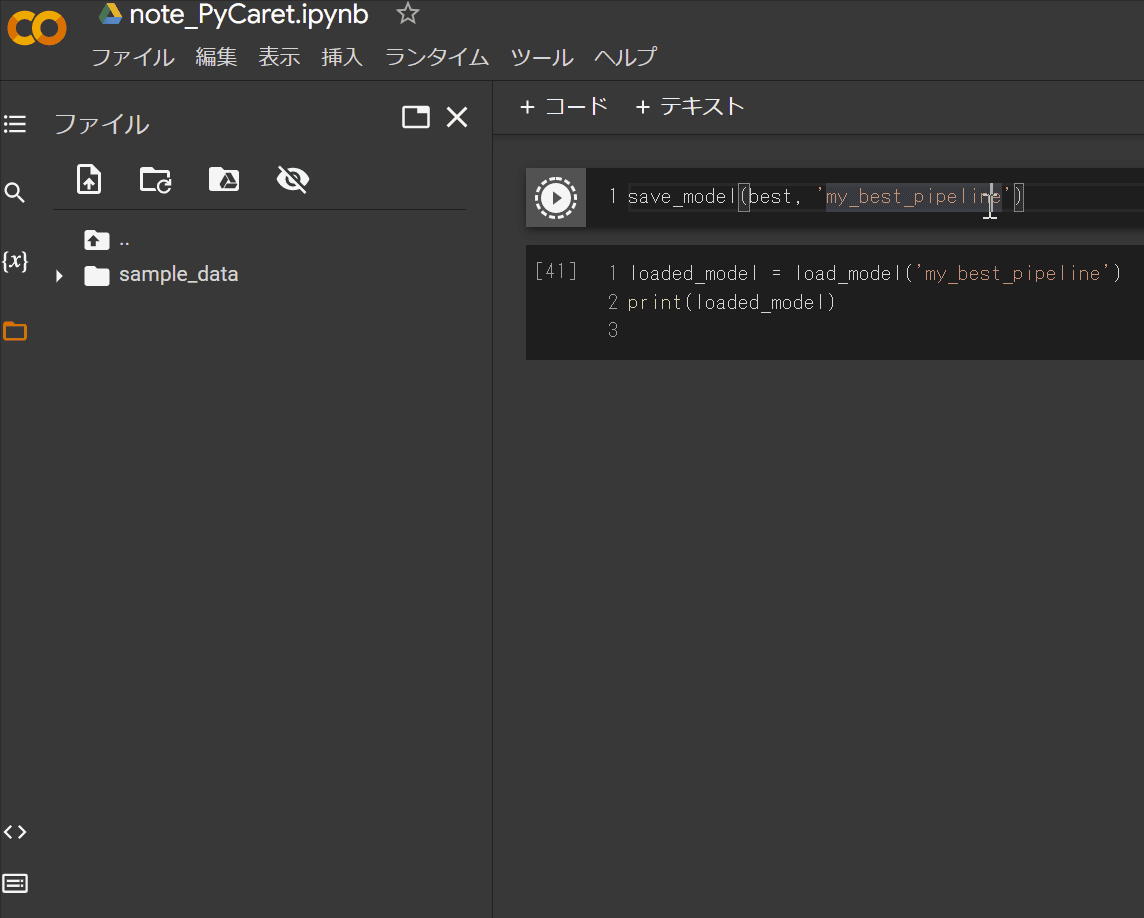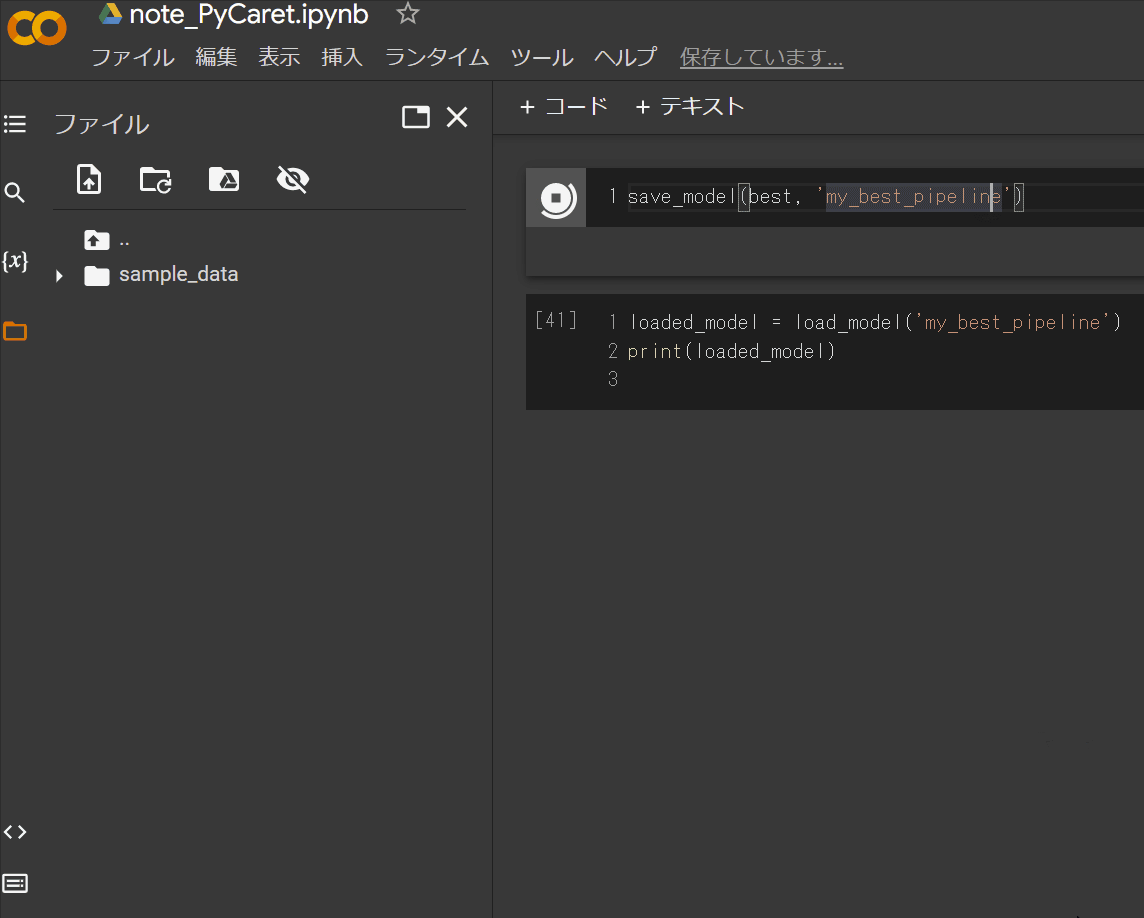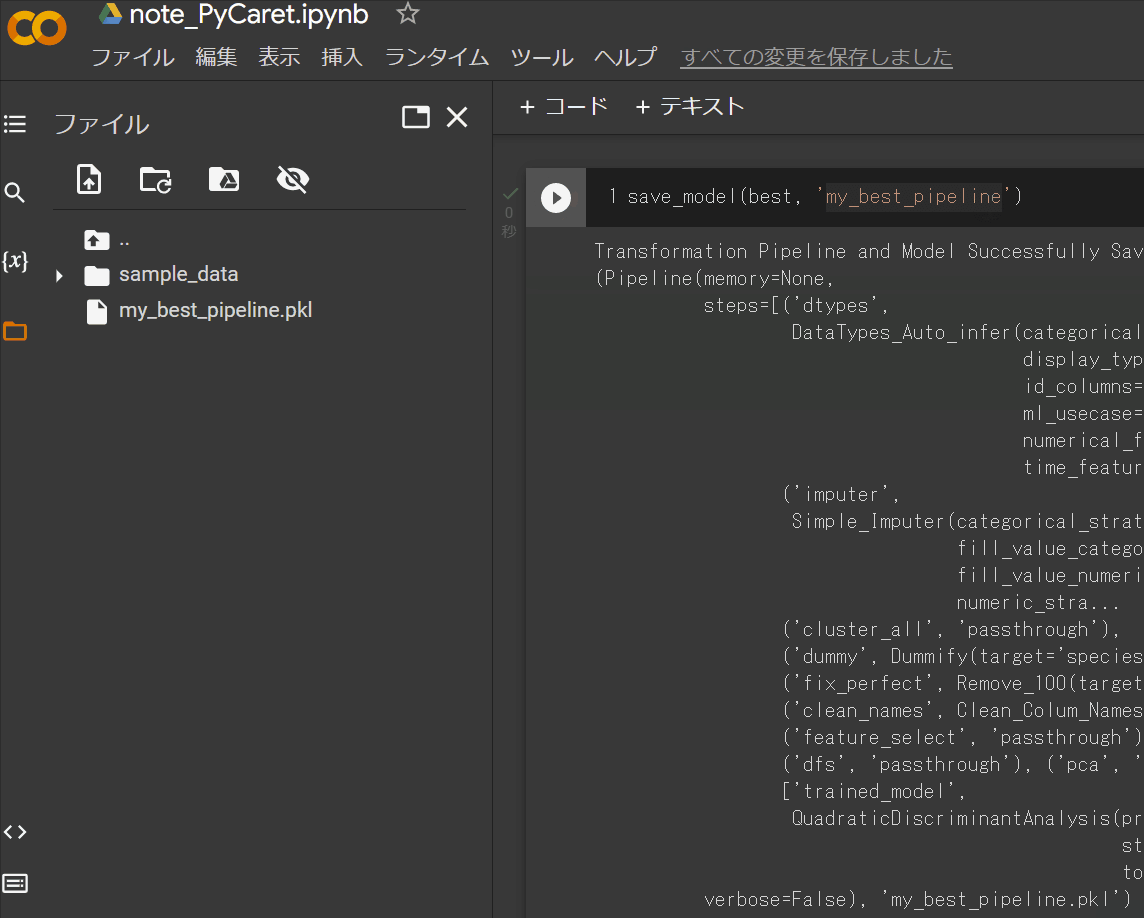
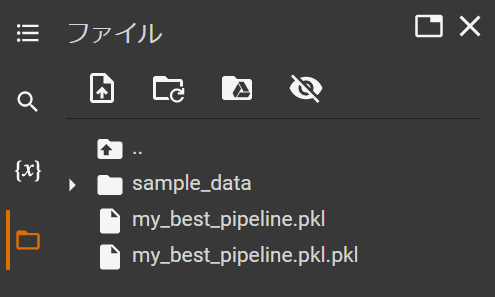
注意点としてPickleファイルを作成することを意識して拡張子.pklを記載すると下記の通り変なファイルが作成されます。
8-2.モデルの読み込み:load_model('name')
save_model()で作成したPickleファイルを読み込む場合はload_model()を使用します。
[IN]
loaded_model = load_model('my_best_pipeline')
print(loaded_model)
[OUT]
Transformation Pipeline and Model Successfully Loaded
Pipeline(memory=None,
steps=[('dtypes',
DataTypes_Auto_infer(categorical_features=[],
display_types=True, features_todrop=[],
id_columns=[],
ml_usecase='classification',
numerical_features=[], target='species',
time_features=[])),
('imputer',
Simple_Imputer(categorical_strategy='not_available',
fill_value_categorical=None,
fill_value_numerical=None,
numeric_stra...
('cluster_all', 'passthrough'),
('dummy', Dummify(target='species')),
('fix_perfect', Remove_100(target='species')),
('clean_names', Clean_Colum_Names()),
('feature_select', 'passthrough'), ('fix_multi', 'passthrough'),
('dfs', 'passthrough'), ('pca', 'passthrough'),
['trained_model',
QuadraticDiscriminantAnalysis(priors=None, reg_param=0.0,
store_covariance=False,
tol=0.0001)]],
verbose=False)Pickleファイルを読み込むため拡張子込みで記載するとエラーが出ます。

9.アンサンブル
複数モデルの結果を多数決で予想させる手法をアンサンブルといいます。アンサンプルは①平均をとるシンプルなアンサンブル:blend_models()と②スタッキング:stack_models()があります。
※ensembleは別の引数で使用されているため注意
9-1.アンサンブルの手動作成:blend_models()
create_model()で作成したオブジェクトをリストでblend_models()に渡してアンサンブルを作成します。
【blend_models()の引数】
●fold{defalut:10}:交差検証(K-Fold)の回数を変更
●method{defalut:auto}※分類のみ:soft:probability(確率->predict_probaメソッドなどで使用)、hard:それ以外
●weights:アンサンブルのモデルに重み(加重平均)をつけてモデル選択に有意差をつける
●choose_better{default:False}:Trueにするとアンサンブルより単一モデルの方が性能が高い場合アンサンブルではなく最高性能モデルを選択する
[IN]
logistic = create_model('lr') #Logistic Regression
dtree = create_model('dt') #決定木
lightgbm = create_model('lightgbm') #LigthGBM
ridge = create_model('ridge') #LigthGBM
#アンサンブル
blender = blend_models([logistic, dtree, lightgbm, ridge])
type(blender)
print(blender)
[OUT]
sklearn.ensemble._voting.VotingClassifier
VotingClassifier(estimators=[('lr',
LogisticRegression(C=1.0, class_weight=None,
dual=False, fit_intercept=True,
intercept_scaling=1,
l1_ratio=None, max_iter=1000,
multi_class='auto',
n_jobs=None, penalty='l2',
random_state=5549,
solver='lbfgs', tol=0.0001,
verbose=0, warm_start=False)),
('dt',
DecisionTreeClassifier(ccp_alpha=0.0,
class_weight=None,
criterion='gini'...
random_state=5549, reg_alpha=0.0,
reg_lambda=0.0, silent='warn',
subsample=1.0,
subsample_for_bin=200000,
subsample_freq=0)),
('ridge',
RidgeClassifier(alpha=1.0, class_weight=None,
copy_X=True, fit_intercept=True,
max_iter=None, normalize=False,
random_state=5549, solver='auto',
tol=0.001))],
flatten_transform=True, n_jobs=-1, verbose=False,
voting='hard', weights=None)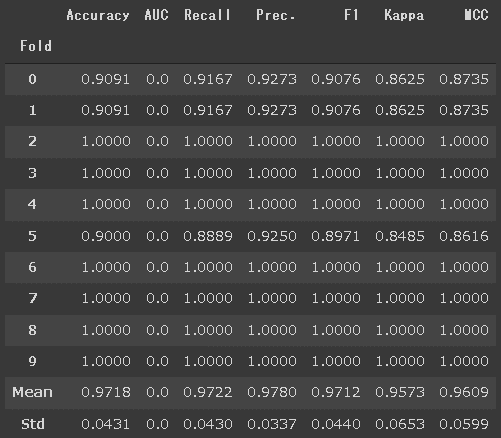
9-2.アンサンブル加重平均:blend_models(weights)
アンサンブルに加重平均を加える場合はweightsに荷重割合を渡します。またアンサンブルOjbをtune_model()に渡すとweightが自動調整されます。
[IN]
blender_weighted = blend_models([logistic, dtree, lightgbm, ridge],
weights = [0.4, 0.3, 0.25, 0.05])
tuned_blender = tune_model(blender_weighted)
tuned_blender
[OUT ※最終行のweightsが入力値から変更されているのに注目]
VotingClassifier(estimators=[('lr',
LogisticRegression(C=1.0, class_weight=None,
dual=False, fit_intercept=True,
intercept_scaling=1,
l1_ratio=None, max_iter=1000,
multi_class='auto',
n_jobs=None, penalty='l2',
random_state=5549,
solver='lbfgs', tol=0.0001,
verbose=0, warm_start=False)),
('dt',
DecisionTreeClassifier(ccp_alpha=0.0,
class_weight=None,
criterion='gini'...
random_state=5549, reg_alpha=0.0,
reg_lambda=0.0, silent='warn',
subsample=1.0,
subsample_for_bin=200000,
subsample_freq=0)),
('ridge',
RidgeClassifier(alpha=1.0, class_weight=None,
copy_X=True, fit_intercept=True,
max_iter=None, normalize=False,
random_state=5549, solver='auto',
tol=0.001))],
flatten_transform=True, n_jobs=-1, verbose=False,
voting='hard', weights=[0.05, 0.76, 0.2, 0.27])9-3.アンサンブルの自動作成:compare_models(n_select=3)
compare_models()で取得できる上位モデルを自動でアンサンブル化する場合はcompare_models(n_select=3)をblend_models()に渡します。
[IN]
blender = blend_models(compare_models(n_select=3))
blender
[OUT]
VotingClassifier(estimators=[('gbc',
GradientBoostingClassifier(ccp_alpha=0.0,
criterion='friedman_mse',
init=None,
learning_rate=0.1,
loss='deviance',
max_depth=3,
max_features=None,
max_leaf_nodes=None,
min_impurity_decrease=0.0,
min_impurity_split=None,
min_samples_leaf=1,
min_samples_split=2,
min_weight_fraction_leaf=0.0,
n_estimators=100,
n_iter_no_change=No...
DecisionTreeClassifier(ccp_alpha=0.0,
class_weight=None,
criterion='gini',
max_depth=None,
max_features=None,
max_leaf_nodes=None,
min_impurity_decrease=0.0,
min_impurity_split=None,
min_samples_leaf=1,
min_samples_split=2,
min_weight_fraction_leaf=0.0,
presort='deprecated',
random_state=5549,
splitter='best'))],
flatten_transform=True, n_jobs=-1, verbose=False,
voting='soft', weights=None)
9-4.スタッキング:stack_models()
create_model()で作成したオブジェクトをリストでstack_models()に渡してスタッキングを作成します。
【stack_models()の引数】
●fold{defalut:10}:交差検証(K-Fold)の回数を変更
●method{defalut:auto}:predict_proba, decision_function, predictから任意に選択可能
●meta_model:スタッキングに使用するメタモデルを選択
ー>分類{defalult:LogisticRegression}、回帰{defalult:LinearRegression}
●restack:予想値だけ使用するか学習データも使用するかを選定
※スタッキングを十分に理解できていないため紹介のみ
[IN]
logistic = create_model('lr') #Logistic Regression
dtree = create_model('dt') #決定木
lightgbm = create_model('lightgbm') #LigthGBM
ridge = create_model('ridge') #LigthGBM
#スタッキング
stacker = stack_models([logistic, dtree, lightgbm, ridge])
print(type(stacker))
print(stacker)
[OUT]
<class 'sklearn.ensemble._voting.VotingClassifier'>
VotingClassifier(estimators=[('lr',
LogisticRegression(C=1.0, class_weight=None,
dual=False, fit_intercept=True,
intercept_scaling=1,
l1_ratio=None, max_iter=1000,
multi_class='auto',
n_jobs=None, penalty='l2',
random_state=5549,
solver='lbfgs', tol=0.0001,
verbose=0, warm_start=False)),
('dt',
DecisionTreeClassifier(ccp_alpha=0.0,
class_weight=None,
criterion='gini'...
random_state=5549, reg_alpha=0.0,
reg_lambda=0.0, silent='warn',
subsample=1.0,
subsample_for_bin=200000,
subsample_freq=0)),
('ridge',
RidgeClassifier(alpha=1.0, class_weight=None,
copy_X=True, fit_intercept=True,
max_iter=None, normalize=False,
random_state=5549, solver='auto',
tol=0.001))],
flatten_transform=True, n_jobs=-1, verbose=False,
voting='hard', weights=None)スタッキングの自動作成もアンサンブルと同様の形で実施可能です。
[IN]
stacker = stack_models(compare_models(n_select=3))
stacker
[OUT]
VotingClassifier(estimators=[('gbc',
GradientBoostingClassifier(ccp_alpha=0.0,
criterion='friedman_mse',
init=None,
learning_rate=0.1,
loss='deviance',
max_depth=3,
max_features=None,
max_leaf_nodes=None,
min_impurity_decrease=0.0,
min_impurity_split=None,
min_samples_leaf=1,
min_samples_split=2,
min_weight_fraction_leaf=0.0,
n_estimators=100,
n_iter_no_change=No...
DecisionTreeClassifier(ccp_alpha=0.0,
class_weight=None,
criterion='gini',
max_depth=None,
max_features=None,
max_leaf_nodes=None,
min_impurity_decrease=0.0,
min_impurity_split=None,
min_samples_leaf=1,
min_samples_split=2,
min_weight_fraction_leaf=0.0,
presort='deprecated',
random_state=5549,
splitter='best'))],
flatten_transform=True, n_jobs=-1, verbose=False,
voting='soft', weights=None)参考記事
あとがき
ライブラリの使い方より環境構築のスキルが必要すぎて初心者ご法度感が否めない感じはするけど、Dockerのスキルつけたらかなり有益かも。
ただなんか最新情報が少ない気がする・・・・
