
市販のオリゴには無関係な配列のオリゴが混入している: Nucleic Acids Researchに掲載された論文から
2023年上旬、アメリカのKevin McKernan先生はmRNAコロナワクチンがDNAで汚染されている事を発見し、それ以降プレプリントやSNS等で関連情報を精力的に発信されてきました。コロナワクチンへのDNA汚染問題については私のブログ内でも何度か取り上げてきましたが、当初から私がこの問題に高い関心を持った理由の一つには、実は私自身もかねてより「別のDNA汚染」の研究をしていたという事情があるのです。科学実験の場における汚染はコンタミネーション (contamination) 、略してコンタミと呼ばれます。
もう一つのDNA汚染
今回の記事は、2024年1月にNucleic Acids Research誌に掲載された私自身の論文の紹介になります。トピックとしてはコロナワクチンから若干離れるのですが、ある意味DNA汚染問題とオーバーラップする内容と言えますので、改めてここで紹介させていただく事にしました。
「オリゴ」はもともとギリシャ語の「少ない」を語源としています。例えば「オリゴ糖」とは単糖が数個結合した糖類の事ですが、「オリゴヌクレオチド」とは短いヌクレオチド (DNAまたはRNA) の配列を意味します。このオリゴヌクレオチドは分子生物学の分野において不可欠な素材であり、しばしば単に「オリゴ」と呼ばれます。オリゴの応用範囲はたいへん広く、生命科学のほとんどの分野で使用されていると言っても過言ではありません。また、コロナのPCR検査においてもプライマーやプローブとして使用され、今やオリゴは医療従事者だけではなく一般の人々の身の回りにもあふれています。
今回の研究の発端は、私自身がかねてより別の研究の中で使用していた市販のオリゴDNAに無数の種類の無関係のオリゴが混入していた事を偶然発見した事に始まります。オリゴという材料が関係する分野が広範囲な事からも、この事実は重要かつ公表する意義があると考え、論文として発表しました。
研究を進めて行く中で予想外の問題に直面した場合、私はできる限りその根本原因を突き止めるように努めます。基本的な理由としては、同じ失敗を繰り返さないためなのですが、その検証が思いがけない発見につながる事もあります。一般的に、研究者はネガティブな研究結果に関しては積極的に発表しようとはしません。何故なら、ネガティブな発見、特に「製薬業界に不利益を与える」ような発見や見解はそもそも雑誌社としても論文を採用したがらず、また「業界に反発」するような内容の研究は場合によってはそれ自体が自分の評判すら落としかねないからです。つまり、研究者にとってその研究結果を発表する事のベネフィットがコストに釣り合わないと判断されがちなのです。しかしながら、他の研究者が同様の問題に悩まずに済むように、また研究時間やリソースを無駄にしないためにも、ネガティブな情報を共有する事も大切であると私は考えます。そして今回の私の論文はそうした研究に該当するものと言えます。
以下が論文のリンクです⬇︎
https://academic.oup.com/nar/article/52/6/3137/7602849?login=false
Arakawa et al. (2024)
Cross-contamination of CRISPR guides and other unrelated nucleotide sequences among commercial oligonucleotides
Nucleic Acids Res
市販のオリゴヌクレオチドにおけるCRISPRガイドとその他の無関係なヌクレオチド配列の交差汚染
CRISPRと遺伝子の「お宝探し」
私が最初にこの問題に気付いたのはCRISPRによるゲノム編集の実験をしている時でした。CRISPR (clustered regularly interspaced short palindromic repeat、クリスパー) の本来の機能は細菌の獲得免疫です。細菌がファージ (ウイルス) やプラスミドに感染すると、細菌は感染源のDNAの断片を「DNA上の感染の記憶」としてCRISPR遺伝子座に組み込みます。そして細菌が再び同じ病原体に感染すると、記憶していた遺伝情報から短いRNAを合成してCas9に組み込み、Cas9が配列特異的エンドヌクレアーゼとして働き、感染源のゲノムを切断して排除します。
特異的な短いRNA (ガイドRNA) をデザインしてCas9と共に発現させると標的DNAを切断でき、この仕組みがゲノム編集にも応用されています。CRISPRによって多数の細胞で並列して遺伝子ノックアウトを行い、細胞の表現系から遺伝子を探索する事もできます。この実験手法はライブラリと呼ばれる技術であり、遺伝子の「お宝探し」に応用できるのです。
CRISPRの発見以前にもゲノム編集が容易なモデル系が一つ存在していました。それがニワトリB細胞株DT40です。DT40では相同組換えを利用して高い効率で任意の遺伝子座を加工する事ができるため、DT40はまさに唯一無二のゲノム編集のモデル細胞と言えます。また、ニワトリB細胞にはもう一つの特徴があります。抗体遺伝子の多様化をV(D)J組換えではなく、遺伝子変換によって行うという性質です。遺伝子変換は単純な組換えではなく、コピー&ペーストの仕組みで遺伝情報を多様化しますが、その分子機構の全容は未だ解明されていません。そしてこれらの現象が私の本来の専門分野なのです。ちなみにB細胞はもともとニワトリで発見された細胞であり、B細胞の「B」とは鳥類特有のB細胞器官「ファブリキウス嚢 (the Bursa of Fabricius)」に由来します。遺伝子変換と遺伝子標的組換えを制御する酵素など、ニワトリから始まったB細胞免疫には今も大きな謎が残っています。私には学生の頃から探し続けている遺伝子があるのです。
さて、私は「どの生物からでもmRNAをガイド配列に変換できる技術」を発明し、2016年にScience Advances誌に論文として発表しました。
以下が論文のリンクです⬇︎
https://www.science.org/doi/10.1126/sciadv.1600699
この技術を使っての未知の遺伝子探索を私は「遺伝学的お宝探しプロジェクト」と呼んでいます。
PCR産物のディープシークエンシングには工夫が必要
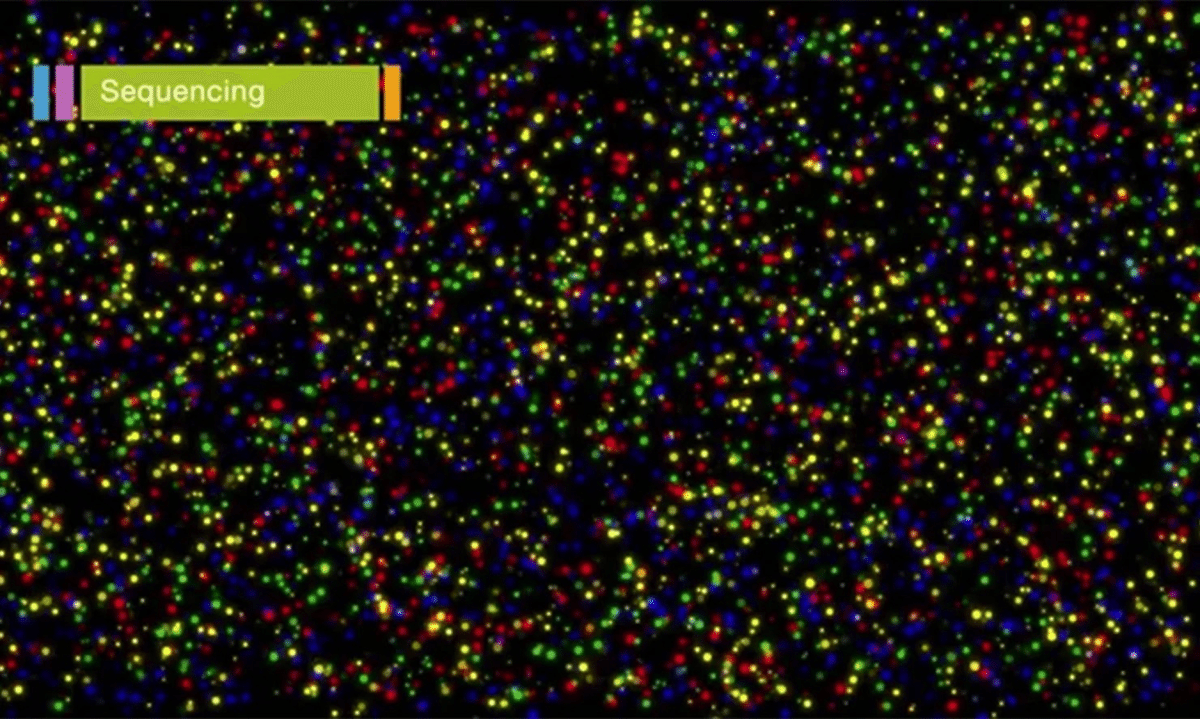
https://www.uclftr.com/post/how-dna-sequencing-became-cheap-and-changed-biotech-once-and-for-all
ここでディープシークエンシングの技術について少しお話しします。イルミナのディープシークエンシングは光学技術の応用でもあり、DNAの塩基配列は顕微鏡による蛍光シグナルの画像から解析されます。図1はイルミナによるディープシークエンシングの画像の一例です。点滅する光の点 (クラスター) は検体DNAを示し、色の違いが配列を意味します。経時的に変化する色の違いを読み取る事でそれぞれのクラスターの配列 (リード) が決定されます。ここで注意すべき点は、隣りあったクラスターが同じ色だと解析の際に両者が混同される恐れがある事です。
実はイルミナは末端に同一の配列を持つDNAの解析を苦手とします。イルミナは最初の11塩基の違いによってそれぞれのリードを認識しますが、そのためPCR産物のように最初が同じ配列の場合、複数のリードを単一であると誤認識しがちであり、途中から別の配列に切り替わり配列が混ざってしまう事があるのです。そうなるとPCR産物のディープシークエンシングの解析は精度が極端に下がってしまうため、シークエンス自体をやり直す羽目になるという経験が私にもあります。
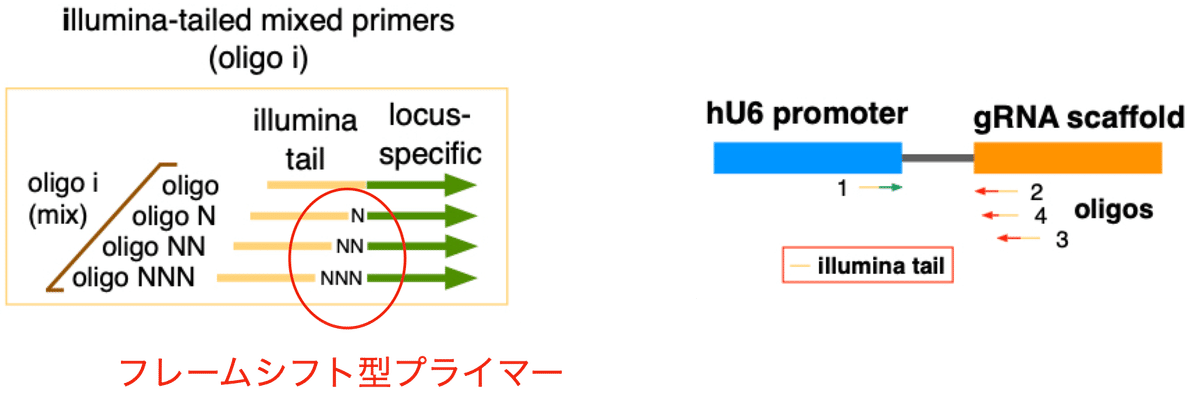
今回のディープシークエンシング解析では検体の末端に多様性を持たせるために、私はあえて「フレームシフト変異」を導入したPCRプライマーをデザインしました(図2)。
遺伝子座特異的配列とイルミナテールの間に0~3塩基のランダム配列 (N) を挿入し、配列の多様性を生み出しました。そして追加ヌクレオチドの数により、オリゴ、オリゴN、オリゴNN、オリゴNNNと、イルミナの4つのプライマーの混合物をオリゴ i (イルミナ) と名付けました。また、順向きプライマーと逆向きプライマーのそれぞれについて4つのオリゴを混合し、それぞれのPCRに使用しました。
鋳型無しでPCR産物が生じた?
通常のPCRには一対 (2つ) のプライマーを用いますが、今回のフレームシフトPCR法では多様性を生じさせるために、4+4の8つのプライマーを用いました。
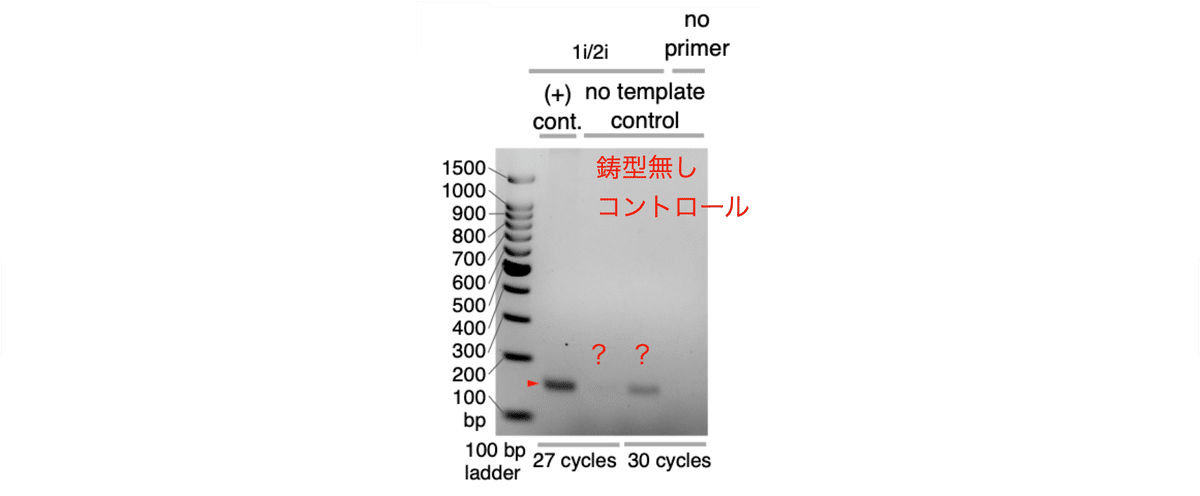
ゲノムDNAに組み込まれたガイド配列は、27サイクルのPCR後に検出されました (図3)。しかし、鋳型無しコントロール (Non-Template Control, NTC) でもかすかにバンドが現われました。PCRを30サイクルまで延長すると、NTCでより明確にPCR産物が確認でき、PCR成分中にガイド配列が混入していたように見えました。
こうした場合、大抵は自分自身のミスによる事がほとんどです。そこで水、PCRバッファー、ポリメラーゼなど、PCRの成分を別ロットのものに変更し改めて検証してみたところ、それでもやはりPCR産物のサイズや量は変わらなかったのです。つまり、どうもこれは単純なラボ内でのコンタミなどではなさそうでした。
高品質なグレードのオリゴにもコンタミが見られた
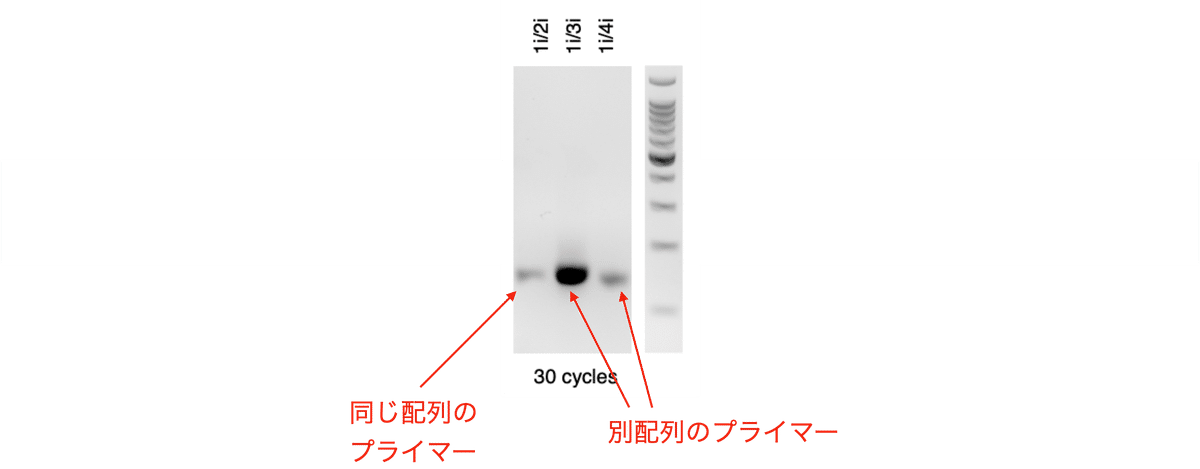
まず1iと2iのオリゴが汚染 (コンタミ) されている可能性が考えられましたので、これらのオリゴの別ロットを再注文しました。また、配列自体に問題がある可能性もありましたので、配列を変更したプライマーも設計し直しました (3iと4i)。
けれども、再注文した同一のプライマーでも再設計したプライマーでもやはり鋳型が無い状態でPCR産物が生成されました (図4)。しかも1i/3iの組み合わせではより多量の非特異的増幅を生じたのです。
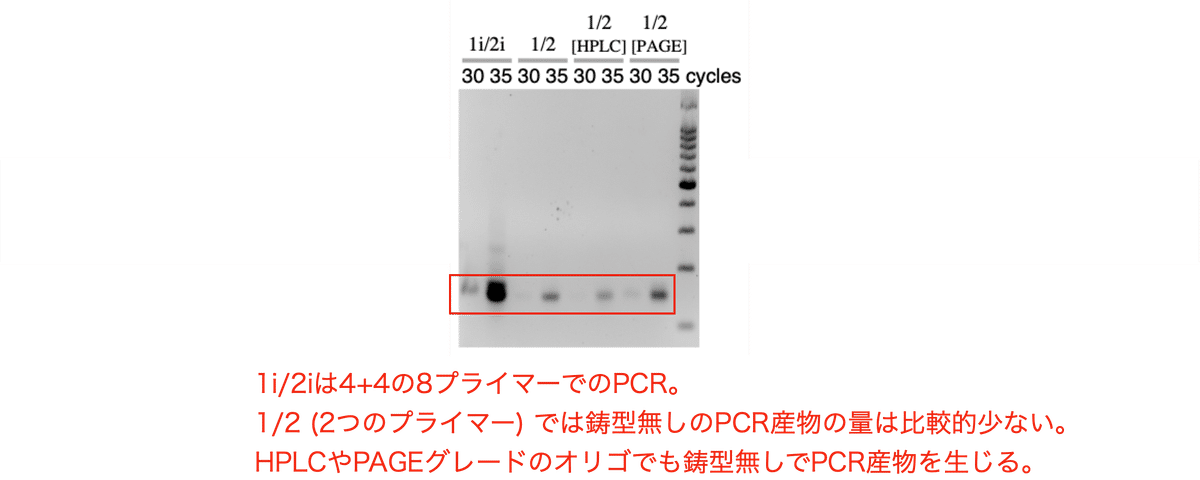
次にオリゴの精製度に問題があるのかもしれないと考え、精製度の高いHPLCやPAGEグレードのオリゴを発注しました。しかしHPLCやPAGEグレードでも、やはりPCR産物が検出されました (図5)。それどころか、PAGEグレードのオリゴでは安価な通常のオリゴ (脱塩グレード) よりもさらに多くのPCR産物を生じました。

この時点でプライマー1、2、3と同じ配列特異性を持つ異なるプライマーのコレクションが既に私の手元にありましたので、それらを用いて12種類のフォワードと7種類のリバースからなる84種類のプライマーの組み合わせで鋳型を用いずに35サイクルのPCRを行いました (図6)。すると、鋳型無しで84種類のプライマーの組み合わせの全てにおいてPCR産物を生じました。
また興味深い事に、プライマーの組み合わせによってもPCR産物の量が異なりました。例えば1NN/3N、1NN/3NN、1NN/3NNNのような組み合わせではPCR産物の生成量が少なかったのです (図6の*印)。このようにNTC PCR産物のサイズや量が微妙に異なる事からも、それぞれのオリゴストックには様々な物質がコンタミとして含まれている可能性が強く疑われました。
オリゴのコンタミは特定の企業だけの問題ではない
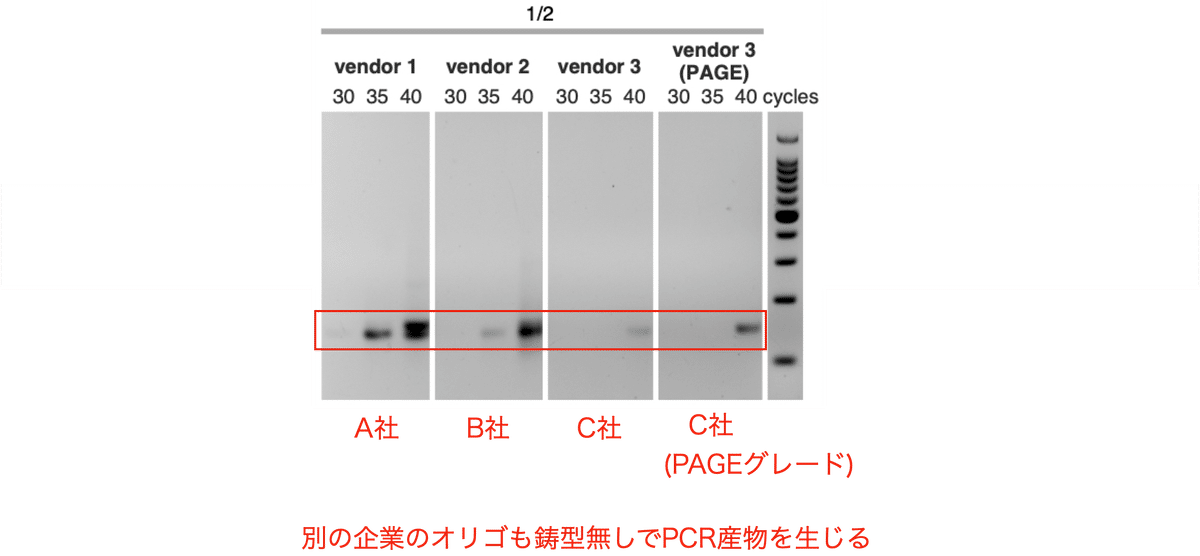
ここまでの実験で使ったオリゴはA社のものでしたが、コンタミが他の企業のオリゴでも見られるのかを調べるために、さらに別の2つの企業 (B社、C社) からオリゴ1とオリゴ2を取り寄せました。ちなみに論文中で各企業名を匿名にしている理由は、いずれも業界を代表すると言って差し支えない大企業ではあるのですが、この研究は特定の企業を糾弾する目的で行われたわけではないからです。
結果、B社とC社のオリゴでもNTCでPCR産物を生じました (図7)。比較するとA社のものよりもPCR産物の量が少なく、程度はA社のものよりは低いのですが、やはりコンタミが見られました。
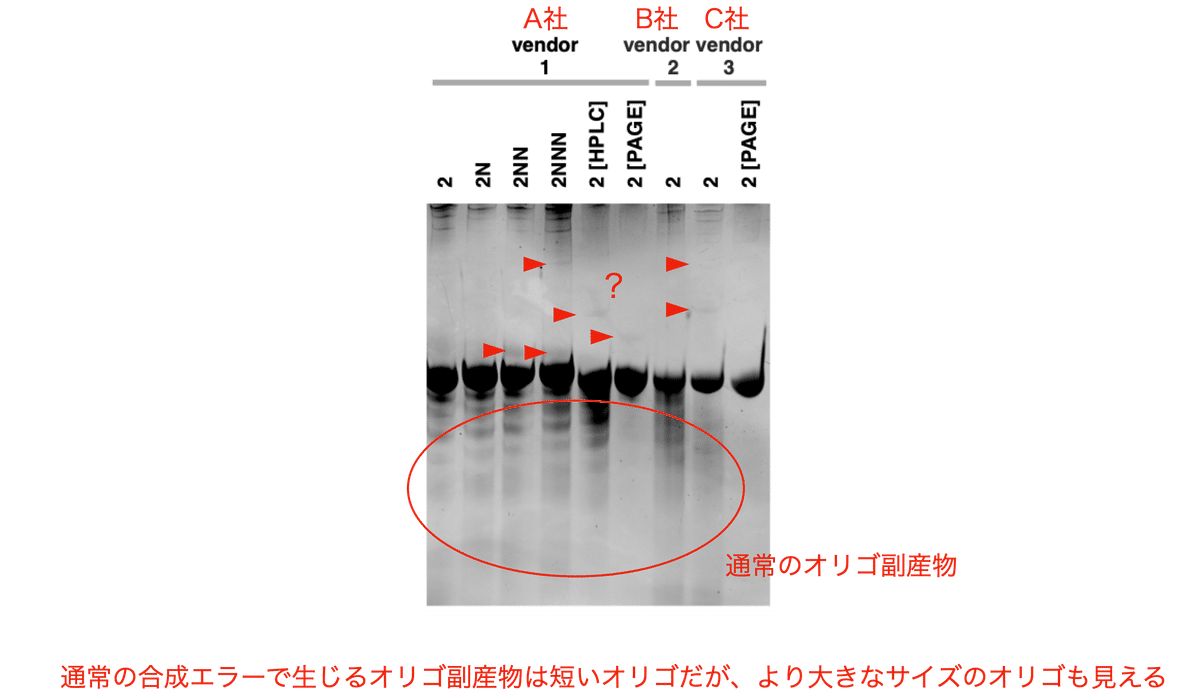
2iのオリゴを電気泳動すると (尿素を含む変性PAGE)、メインのDNAバンドに加え、想定外のバンドがいくつも見つかりました (図8)。通常のオリゴにおいても「失敗オリゴ」が含まれる事自体は正常であり、想定の範囲内です。また、オリゴ合成反応のエラーではオリゴ配列内に欠失が生じますので、不良オリゴはメインのバンドよりも小さなはしご状のバンドを形成します。
しかし、ほとんどの脱塩グレードのオリゴで、由来不明の大きなサイズのバンドがいくつか観察されました。これらの余分で大きなバンドは、A社の高純度HPLCおよびPAGEグレードオリゴでも検出されましたが、C社のPAGEグレードオリゴでは検出されませんでした。
CRISPRガイド配列がオリゴにコンタミしていた
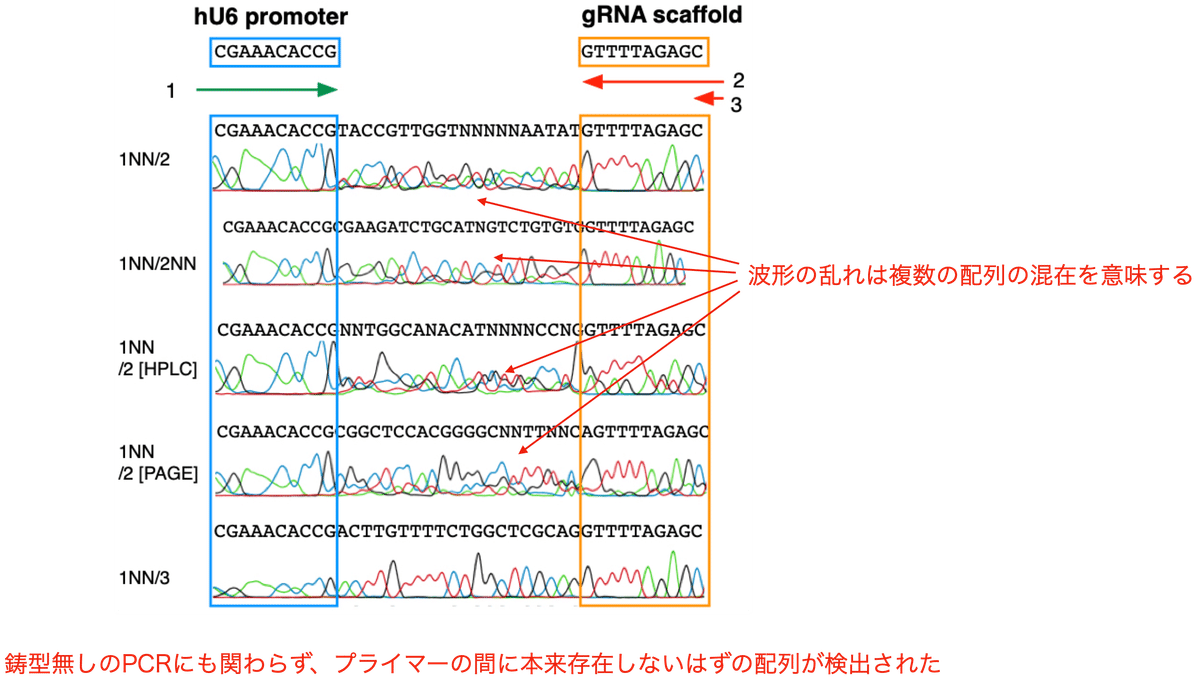
では、コンタミしている配列は一体何なのでしょうか? 確認のためPCR産物のいくつかの塩基配列をサンガーシークエンシングで解析しました (図6の矢印のPCR産物)。すると、PCRに用いたプライマー対の間に「本来存在しないはず」の約20bpの配列が予期せず観察されました (図9)。4つのNTC PCR産物では2つ以上の配列が混在していましたが、1NN/3では単一の特異的配列を生じました。
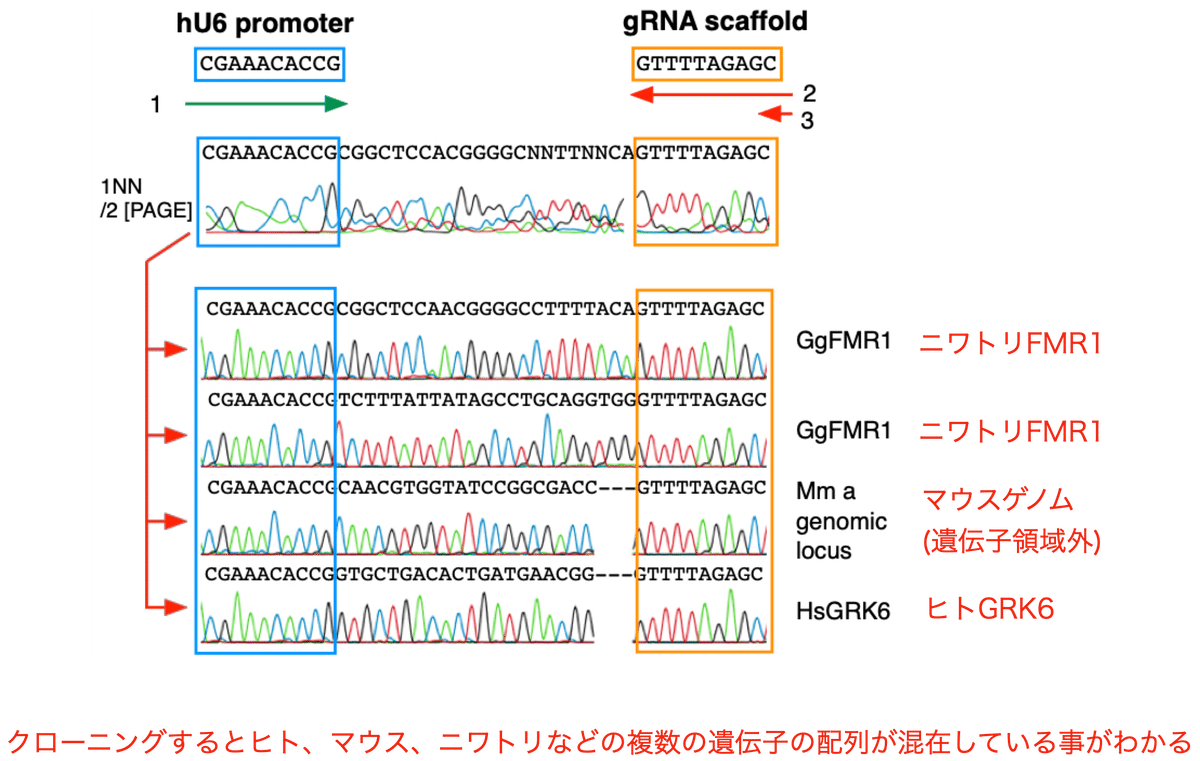
さらに詳しく調べるためにPCR産物をプラスミドベクターにクローニングし、それぞれから6つの独立したプラスミドクローンの塩基配列を決定しました。その解析の一例が図10です。PCR産物クローン全てにCRISPRガイド配列が検出されました。
2つからはニワトリの遺伝子、1つからは遺伝子領域外のマウスゲノム、1つからはヒト遺伝子、他の4つのNTC PCR産物からも様々な生物種由来の多くの異なるガイド配列が検出されました。またその他にもアナホリフクロウ、ゲラダヒヒ、オキナワハツカネズミ、オナガコウモリのなどの遺伝子も見つかりました。検出されたこれらの遺伝子の多様性を考えると、コンタミが私の研究室由来とは到底考えにくいのです。
CRISPRガイド配列のオリゴへのコンタミは国境を越えて普遍的である
この頃、ちょうど別件の共同研究をしていたゲノム編集専門のアメリカの研究者と話す機会があり、私は彼に「自分の解析したオリゴに無関係なガイド配列がランダムに混入していたのだが、これについてどう考えるか?」と聞いてみました。それに対して彼は「まさかそんなはずはないだろう、私には信じられない!」と答えました。もっともな反応です。そこで「では実際に試してみてはどうか?」と提案したところ、彼から「せっかくなので別の日本の知人の研究者にも一緒に試してもらって良いか?」と聞かれ、「是非お願いします。」という話になりました。
その後彼らに私のプロトコルを試してもらったところ、日本からも「出た」、アメリカからも「こちらでも出た」との報告が続き、結果的に私の調べた欧州の3社に加え、日本の企業2社、アメリカの企業3社のいずれのオリゴにもやはり無関係なガイド配列がコンタミしていた事が判明しました。そして、混入している配列はオリゴによって異なり、オリゴへのDNA混入は予想以上に普遍的な問題だと分かってきました。
私のPCRデザインで検出できるコンタミDNAはCRISPRガイド配列のみですが、混入しているのは本当にガイド配列だけなのでしょうか?
CRISPRガイド配列以外の多様なコンタミ
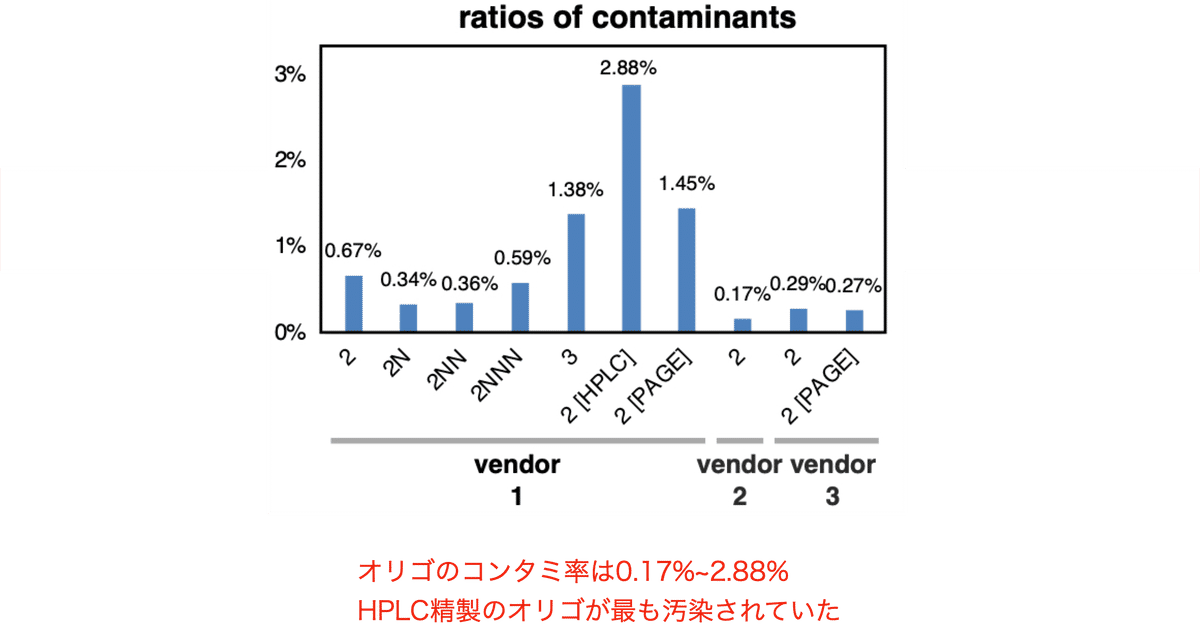
オリゴに混入している遺伝子配列の全体像を知る解明するために、10個のオリゴ (図8の9個のオリゴに加えて、ガイド配列の大量混入が予測されたオリゴ3) をイルミナMiSeqでディープシークエンス解析を行いました。
その結果、分析した10個のオリゴはどれも全て様々なコンタミを含有しており、合計15154のリードがコンタミオリゴ由来でした。コンタミはオリゴによって0.17%から2.88%の範囲でした (図11)。
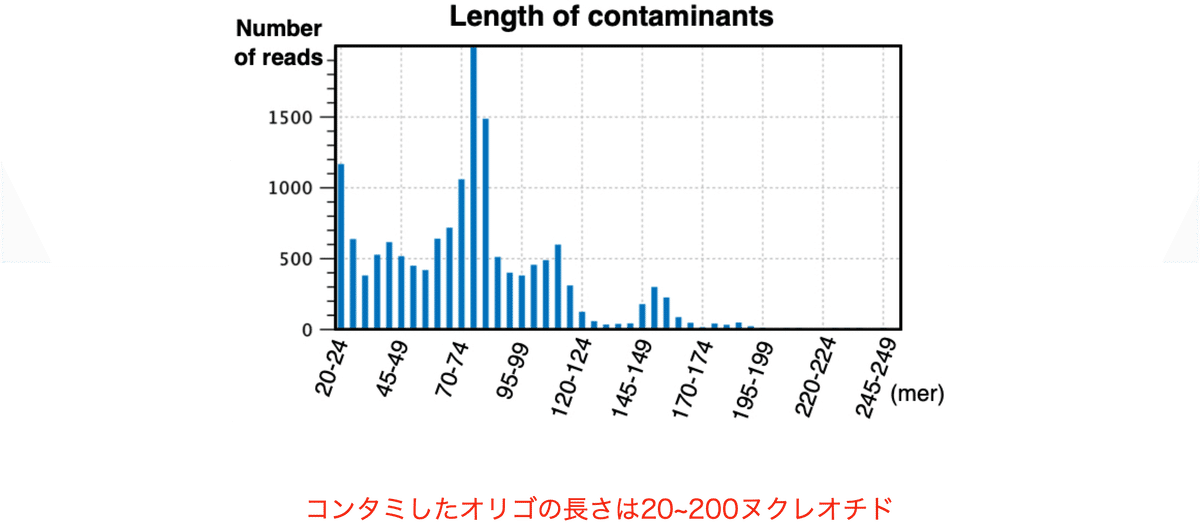
「オリゴ」がもともとギリシャ語で「少ない」を意味するように、以前はオリゴとは20塩基対かそれ以下の短いヌクレオチドを指していたのですが、自動合成技術の進歩によって200塩基ほどのヌクレオチドを合成する事も可能になった現在では、そうした長さのヌクレオチドであっても同様にオリゴと呼ばれています。
今回解析したコンタミオリゴの長さは20~200ヌクレオチド (図12) であり、一般的にオーダーメイドで合成可能なオリゴの範囲と一致しました。

ディープシークエンスリード から、コンタミガイド配列DNAの塩基配列を同定する事に成功しました (図13)。このように市販のオリゴにはPCRの鋳型となる「CRISPRガイド配列オリゴ」がコンタミしていたのです。
ここで少しCRISPRガイド配列についてお話しします。ガイドRNAを発現させるためには適切なプロモーターが必要ですが、最も汎用されているものがヒトU6プロモーターです。またガイドRNAとして機能するためには専用の「足場」が必要であり、それはgRNAスカフォールドと呼ばれています。ガイド配列の鋳型となるオリゴに使われるプロモーターとスカフォールドは共通ですが、ガイド配列自体は遺伝子ごとに様々です。
ディープシークエンシングで同定された配列は、ヒトU6プロモーターとgRNAスカフォールドの一部と共にガイド配列を含んでおり、サンガー配列で決定された配列と一致しました。
オリゴは長いほど合成のコストがかかりますので、コストを節約するためにもオリゴに含まれるプロモーターとスカフォールドの長さは限定的です。今回私はディープシークエンシングのコストを節約するために、できるだけガイド配列の近くにPCRプロモーターが来るように実験をデザインしました。このためコンタミDNAと私のプライマーで重複する部分が長くなり、コンタミDNAを検出する感度が高くなったのです。
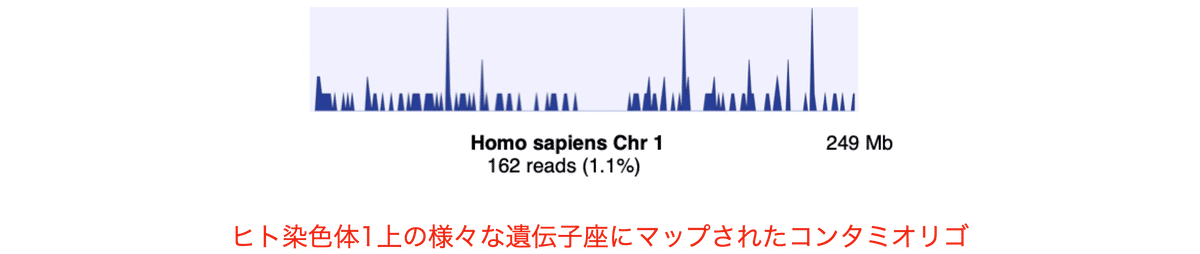
さらに残りのコンタミの起源を探るため、まずコンタミをヒトゲノム上にマッピングしました。例えば162のリードがヒト1番染色体上に同定できました (図14) が、これは1つの例であり、コンタミは他の染色体にも分布していました。つまり、コンタミDNAの中にはヒトの様々な遺伝子配列が含まれていました。
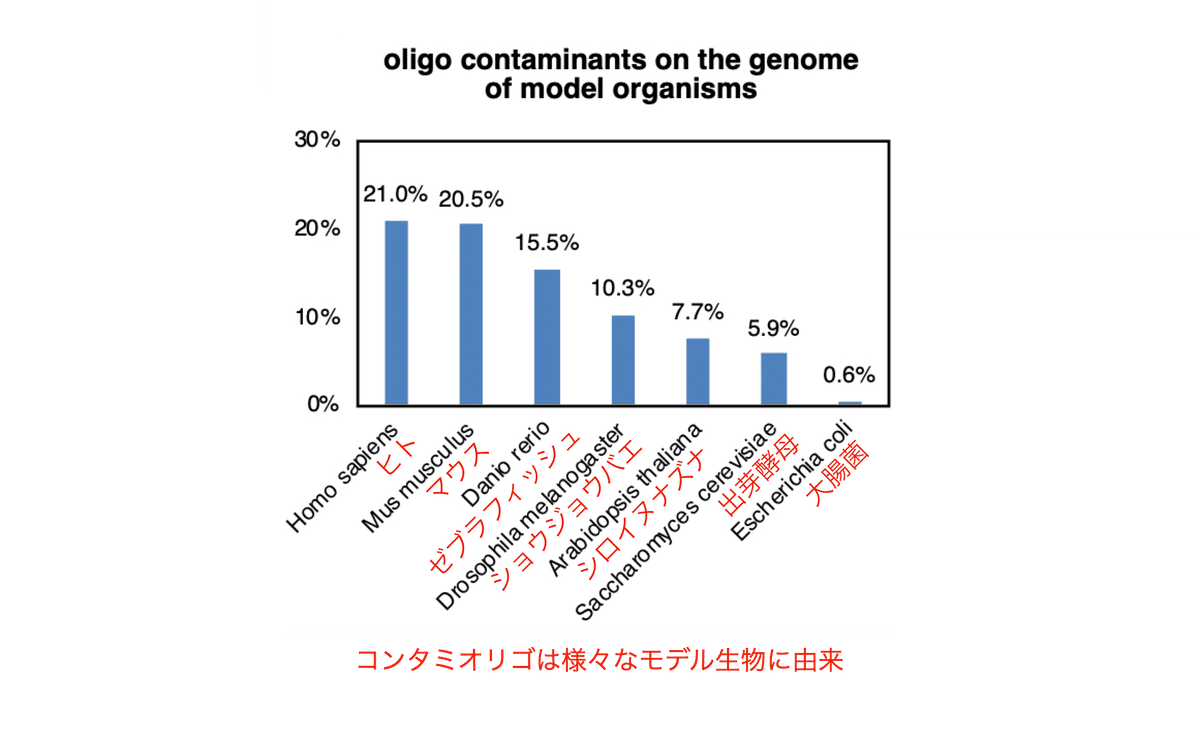
次にコンタミDNA配列を色々なモデル生物のゲノムと比較しました。マウス、ゼブラフィッシュ、ハエ、酵母、植物(シロイヌナズナ)、大腸菌などのモデル生物のゲノム上にもコンタミオリゴはマッピングできました (図15)。このようにコンタミオリゴの由来は実に様々だと分かります。

また、コンタミオリゴはプラスミドベクターにも由来していました (図16)。

他にもコンタミオリゴはウイルスゲノム上にも見つかりました。そして10のリードはHIV上に見つかりました (図17)。
2018年に発注したオリゴに新型コロナウイルスのオリゴがコンタミしていた
さて、ここで大変興味深い事実があるのです。実はオリゴ中のコンタミには「新型コロナウイルス(SARS-CoV-2)」に由来しているものも見つかったのですが、私がそのオリゴを発注したのは「2018年12月」でした。つまり新型コロナウイルスの発見前であり、コロナ騒動の始まる前の事なのです。では、これは一体何を意味するのでしょうか? 私はこの事も新型コロナウイルスが人工ウイルスであり、人為的に作成されたものである状況証拠の1つと考えています。ちなみにこの論文中にもデータは入っているのですが、その意味を考察した文章については査読の際に「論文の主旨とは直接関係しない上に証拠が不十分だ」という理由で削除するように要請されたという経緯があります。その後、私はこの実験データについて新型コロナウイルスが人工物であるという趣旨の私自身の論文内で改めて引用しました。
オリゴは生命科学の分野で昔から実験材料として汎用されており、最近ではコロナのPCR検査によっても広く普及しました。世界中に膨大な数のユーザーがいるにも関わらず、なぜオリゴのコンタミが今までずっと気付かれなかったのでしょうか? 私が今回この問題に気付いたのには偶然いくつかの要因が重なりました。
まず一つ目には、私の実験系が偶然にもコンタミDNAの検出のための感度が高かった事です。私はディープシークエンシングのコストを節約するために、ガイド配列に近い高感度PCRプライマーを使用していました。さらに8種類のプライマーを混合する事で、コンタミを検出する感度をさらに高めていました。
二つ目にはCRISPRが世界的に広く扱われるようになり、ガイド配列がコンタミする機会も増えていた事です。ガイド配列のPCR鋳型は長さが短いためオリゴとして設計できますし、両脇には特定の配列が存在し、中央が異なる多様な遺伝子配列となります。このためガイド配列だけでも多様なコンタミが生じ得るのです。
実際、ここまで普遍的に使用されてきた市販のオリゴ中に無関係なオリゴが混入している事など今まで全く知られていませんでした。例えば、ある研究者がヒトの遺伝子ライブラリから遺伝子を探索する中で、新発見したと思った遺伝子が実はコンタミを拾ったものだったとします。そしてそれに気付かずその遺伝子を解析した場合、解析結果はどう解釈される事になるでしょうか?
オリゴの用途は多岐にわたります。わずかな量の混入DNAの存在は目的によっては問題にならない場合もあるかもしれませんが、目的次第では混入DNAのために結果の解釈を大きく間違える可能性も出てくるのです。腸内細菌や環境中のウイルスなどを単離せずに「ごちゃ混ぜ」で配列を決定し、コンピューター解析によって分類する手法がメタゲノム解析です。遠い昔に絶滅したネアンデルタール人のゲノム解析すらも可能な現在のゲノム解析技術ですが、材料自体に全く関係の無い遺伝子配列が混入していた場合、間違った解析結果が出るかもしれません。また最近では「病気の治療用」のオリゴも盛んに研究されており、これも遺伝子製剤の一端なのです。仮にそういった製剤を人体に使用した場合、短期的には問題が無く見えたとしても、その後時間を経て問題が顕在化する恐れがあるのです。
今回の記事はDNA汚染、人工ウイルス説とも関わりのある私自身のサイドプロジェクトの研究を紹介させていただきましたが、次回からはまたmRNAワクチンの危険性を中心にお話ししていこうと思います。
#コロナワクチン
#ワクチン
#コロナ
*記事は個人の見解であり、所属組織を代表するものではありません。
