
qiime2Rを用いてα多様性解析を美しく描画
この記事では、バイオインフォマティクス解析ツールQiime2で得られたデータから、統計計算環境Rを用いたα多様性解析のデザインについて説明します。
実行環境とソフト
実行環境:Ubuntu 22.04.2 LTS (GNU/Linux 5.15.146.1-microsoft-standard-WSL2 x86_64)
実行ソフト:QIIME 2 2023.9 Amplicon Distribution上のR version 4.2.2
※WSL上でQiime2をactivateし、RでR環境を立ち上げています。
conda activate qiime2-amplicon-2023.9
Rデータの読み込み
tidyverseとqiime2Rをインストール。
Rでメタデータとα多様性データを読み込みます。
install.packages("tidyverse")
library(tidyverse)
if (!requireNamespace("devtools", quietly = TRUE)){install.packages("devtools")}
devtools::install_github("jbisanz/qiime2R")
library(qiime2R)
metadata <- read_q2metadata("sample-metadata.tsv")メタデータ
> head(metadata)
SampleID 3groups 4groups days-since-experiment-start date-taken
1 imm2rpt imm imm 0 231116
2 imm3rpt imm imm 0 231116
3 imm4rpt imm imm 0 231116
4 1day3 1day 1day 1 231030
5 1day4 1day 1day 1 231030
6 1dayrpt 1day 1day 1 231116"qiime diversity core-metrics-phylogenetic"で得られるα多様性指数を読み込み、metadataと結合。
observed_features <- read_qza("alpha-diversity/Sample_depth_10000/core-metrics-results/observed_features_vector.qza")
observed_features <- observed_features$data %>% rownames_to_column("SampleID")
metadata <- metadata %>% left_join(observed_features)結合後
> head(metadata)
SampleID 3groups 4groups days-since-experiment-start date-taken
1 imm2rpt imm imm 0 231116
2 imm3rpt imm imm 0 231116
3 imm4rpt imm imm 0 231116
4 1day3 1day 1day 1 231030
5 1day4 1day 1day 1 231030
6 1dayrpt 1day 1day 1 231116
observed_features
1 1241
2 1222
3 1083
4 648
5 630
6 745注意点
指標faith_pdのときの"faith_pd_vector.qza"ファイルのように、データの形が異なる場合があります。
> head(observed_features$data)
observed_features
1day3 648
1day4 630
1dayrpt 745
7dayrpt 854
9day1 605
9day2 546
> head(faith_pd$data)
V1 V2
1 1day3 52.85961
2 1day4 52.46804
3 1dayrpt 65.10177
4 7dayrpt 66.06848
5 9day1 47.87399
6 9day2 43.22798列名などを変換して調整する必要があります。
> colnames(faith_pd$data) <-c("SampleID", "faith_pd")
> metadata<-
metadata %>%
left_join(faith_pd$data)
Joining with `by = join_by(SampleID, faith_pd)`
> head(metadata)
SampleID 3groups 4groups days-since-experiment-start date-taken faith_pd
1 imm2rpt imm imm 0 231116 95.94016
2 imm3rpt imm imm 0 231116 97.95470
3 imm4rpt imm imm 0 231116 84.21200
4 1day3 1day 1day 1 231030 52.85961
5 1day4 1day 1day 1 231030 52.46804
6 1dayrpt 1day 1day 1 231116 65.10177α多様性の可視化
Rの可視化ライブラリggplot2を用いて可視化します。
#順番の変更
preferred_order <- c("imm", "1day", "long")
#箱ひげ図
#'3groups'はmetadataでグループを指定している列の名前に変えてください
metadata %>%
filter(!is.na(observed_features)) %>%
mutate(`3groups` = factor(`3groups`, levels = preferred_order)) %>%
ggplot(aes(x = `3groups`, y = observed_features, fill = `3groups`)) +
geom_boxplot() +
stat_summary(geom = "errorbar", fun.data = mean_se, width = 0) +
stat_summary(geom = "line", fun.data = mean_se, aes(group = 1)) +
stat_summary(geom = "point", fun.data = mean_se) +
xlab("Groups") +
ylab("Observed Features Diversity") +
scale_y_continuous(limits = c(0, NA)) +
theme_q2r() +
ggsave("alpha-compare_observed_features.png", height = 3, width = 4, device = "png")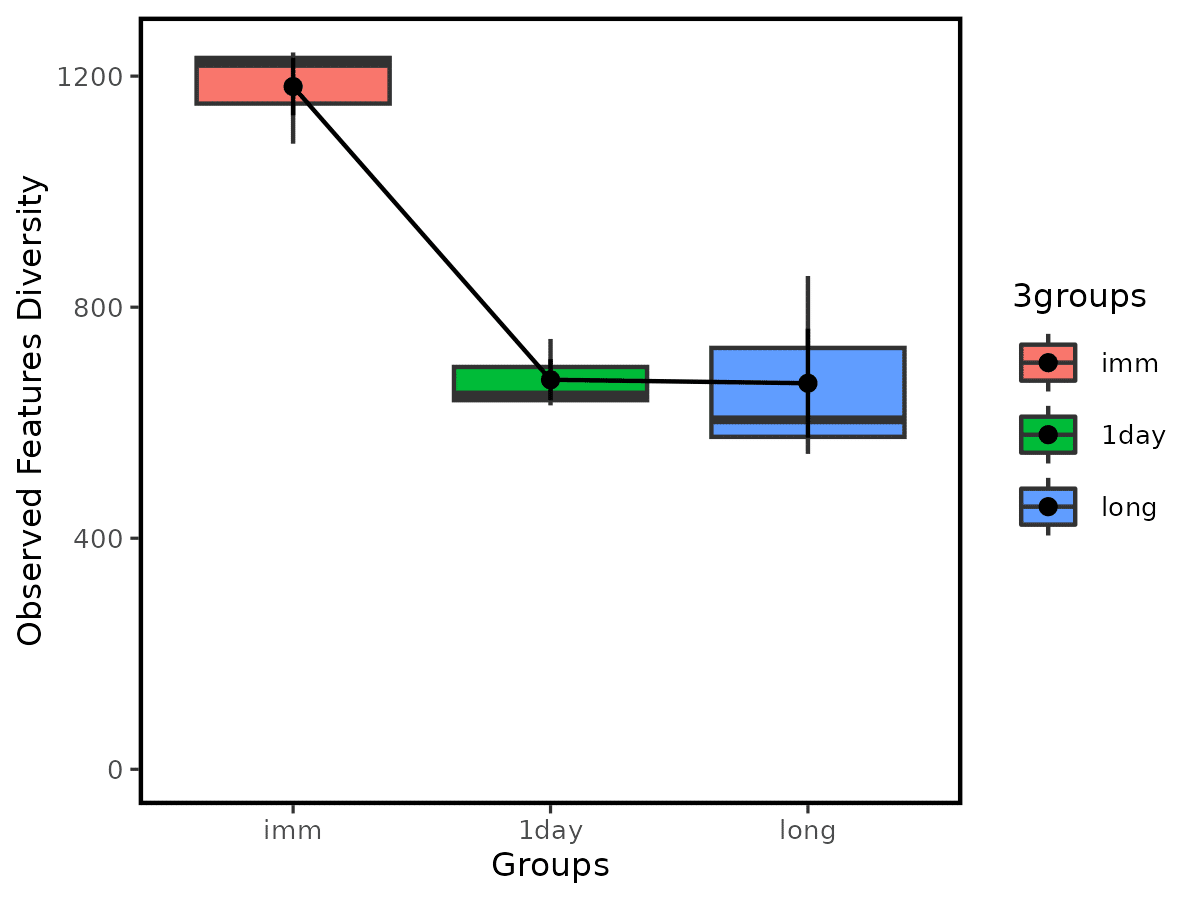
まとめ
この記事では、Rを用いたα多様性の可視化方法を説明しました。
異なるグループ間で観察された特徴の多様性を可視化することで、微生物群集組成に関する貴重な洞察を得ることができます。
次回はβ多様性の可視化をします
