オレ流系統解析 アライメント
野球の話ではありません、前回紹介した系統解析とは別の解析を紹介します。完全な自己流ですので、どこまで参考になるかわかりませんが、使う事があれば流用して下さい。
オレ流とは?
私は、指示されたことだけをしっかりとこなすダメな大学院生でした。そのため、研究室の准教授が魚類のDNA解析を行なっていたにも関わらず、私はその技術を学んで寄生虫に活かすことをしませんでした。分子系統解析は、就職してから業務の傍ら京大の院生に教えてもらいながら身につけて行きました。教科書は少し読みましたが、講義は受けたことはなく、ウェブ上のプロトコルをもとに自分でやり方を身につけていったことから“オレ流”と表現しました。
今回の解析法の紹介をしようと考えたのは、知人(教え子ですが)から、字しかないウェブ上のプロトコルは分かりにくいので、写真付きで説明して欲しいと言われたのがきっかけです。前回は、MEGAという高校の教科書にも出ており、大学の実習でも用いられる有名で使いやすい解析ソフトを紹介しました。ただ、今回紹介する解析ソフトは少々専門性が高いのでこの記事の内容は面白くないだけでなく、役にも立たないかもしれません。本当に、必要としている人が見てくれたらと思っています。
真似するのが一番!
系統樹の作製方法を習得するためには、論文の方法に従って同じものが作れるか試してみるのが一番です。他の人の論文のデータが使えるのか疑問に感じる方もいると思いますが、塩基配列の解析を伴う研究を行った場合、判明した塩基配列は番号を付けてもらってGenbankに登録します。論文には解析で使用した塩基配列のリストや解析法がのせられており、他の人が再現することが可能になっています。ここでは、昨年出版された私の論文をもとに前回とは違った系統解析を紹介します。
ここからの内容を試してみたいという方がおりましたら、以下のリンクから私の論文をダウンロードしてご利用してください。
まず、Table 1にあるリストをもとに塩基配列をダウンロードして、塩基配列のリストのファイルを作成してください(28S rRNAの方をお願いします)。リストの作成法は以下の記事を参考にしてください。
次に、GoogleでMAFFTで検索してください。トップページにあるOnline versionをクリックしてください。

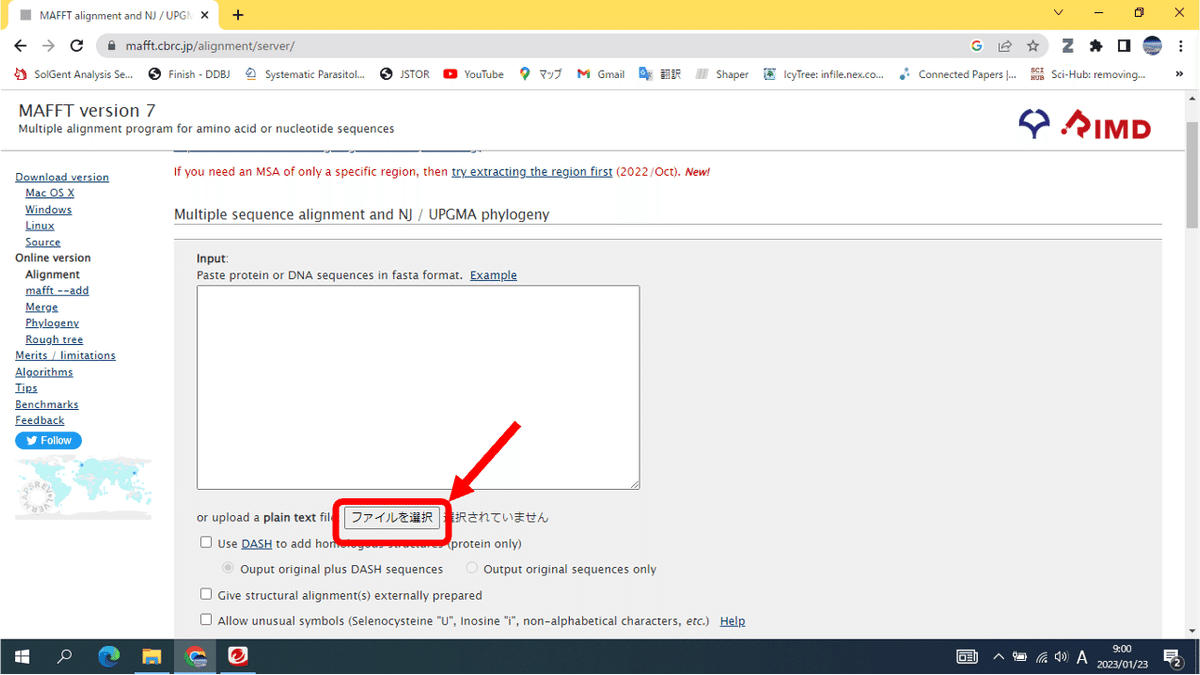
この状態で、一番下のSubmitをクリックしてもよいのですが、論文のMaterial and Methodsにあるように設定を行います。論文には、“The sequences of 28S were aligned using MAFFT version 7 (Katoh et al. 2019) using the “unalignlevel: 0.8” and “Leave gappy regions” options under the G-INS-i strategy.”とあります。この方法を画像でお見せすると。




次に、Gblocksというサイトで、“Ambiguous sites in the aligned datasets were removed with Gblocks ver. 0.91b (Castresana 2002) using the “Allow smaller final blocks” option.”とおいう処理を行います。


参考文献
Kamio, Y., & Nitta, M. (2022). New records of Gemmaecaputia corrugata (Monogenea: Chauhaneidae) from Sphyraena forsteri (Sphyraenidae) off Yomitan Town, Okinawa-jima island, southern Japan. Species Diversity, 27, 83–90.
この記事が気に入ったらサポートをしてみませんか?