Pfizer BioNTech COVID-19 mRNA Vaccine BNT162b2 in vitro ヒト肝細胞株における細胞内逆転写
by Markus Aldén ,Francisko Olofsson ,Daowei Yang ,Mohammad Barghouth 1,Cheng Luan ,Magnus Rasmussen Yang De Marinis
1 Department of Clinical Sciences, Lund University, 20502 Malmö, Sweden
2 Infection Medicine, Department of Clinical Sciences, Lund University, 22362 Lund, Sweden
*
Author to whom correspondence should be addressed.
Academic Editor: Stephen Malnick
Curr. Issues Mol. Biol. 2022, 44(3), 1115-1126; https://doi.org/10.3390/cimb44030073 (registering DOI)
Received: 18 January 2022 / Revised: 19 February 2022 / Accepted: 23 February 2022 / Published: 25 February 2022
(This article belongs to the Topic Clinical, Translational and Basic Research on Liver Diseases)
概要
ファイザー社とバイオテック社が開発したCOVID-19 mRNAワクチンBNT162b2の前臨床試験では、BNT162b2の注射を受けた動物に可逆的な肝機能の効果が認められました。さらに、最近の研究では、SARS-CoV-2 RNAが逆転写され、ヒト細胞のゲノムに統合されることが示された。本研究では,BNT162b2がヒト肝細胞株Huh7に及ぼす影響をin vitroで検討した.Huh7細胞をBNT162b2に暴露し,細胞から抽出したRNAに対して定量PCRを行った.その結果、Huh7細胞においてBNT162b2が高レベルで検出され、内因性逆転写酵素であるlong interspersed nuclear element-1(LINE-1)の遺伝子発現が変化することが確認された。BNT162b2 で処理した Huh7 細胞で LINE-1 open reading frame-1 RNA-binding protein (ORFp1) に結合する抗体を用いた免疫組織染色により、LINE-1 の核内分布が増加することが示唆された。BNT162b2で処理したHuh7細胞のゲノムDNAをPCRしたところ、BNT162b2に特異的なDNA配列が増幅された。この結果は、BNT162b2がヒト肝細胞Huh7に速やかに取り込まれ、LINE-1の発現と分布に変化をもたらすことを示している。また、BNT162b2のmRNAは、BNT162b2曝露後、6時間という短時間で細胞内でDNAに逆転写されることも明らかにした。
キーワード COVID-19 mRNAワクチン;BNT162b2;肝臓;逆転写;LINE-1;Huh7
1. はじめに
重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)によるコロナウイルス病2019(COVID-19)は、2020年3月11日に世界保健機関(WHO)によってグローバルパンデミックと発表され、破壊的な健康危機として浮上した。2022年2月現在、COVID-19は世界中で4億3,000万人以上の感染症例と590万人の死者を報告しました[1]。COVID-19に関連する罹患率および死亡率を低減するために、有効かつ安全なワクチンが緊急に必要とされています。
COVID-19用のいくつかのワクチンが開発されており、特にmRNAワクチン(ファイザー・バイオテック社およびモデルナ社)、複製欠損組換えアデノウイルスベクターワクチン(ヤンセン・ジョンソン・アンド・ジョンソン社、アストラゼネカ社、スプートニックV社、カンシノ社)、不活化ワクチン(シノファーム社、バラットバイオテック社、シノヴァック社)に焦点が当てられています。mRNAワクチンは、免疫原の設計や製造が柔軟かつ効率的に行えるという利点があり、現在、数多くのワクチン候補が様々な開発・応用段階にあります。具体的には、Pfizer社とBioNTech社が開発したCOVID-19 mRNAワクチンBNT162b2が臨床試験で評価され[2,3,4]、世界各地のCOVID-19国家予防接種キャンペーンで投与され[5,6,7,8]、成功に至っています。
BNT162b2は、脂質ナノ粒子(LNP)カプセル化された、ヌクレオシド修飾RNAワクチン(modRNA)で、抗原的に最適な融合前コンフォメーションを確保するために2つのプロリン変異で修飾されたSARS-CoV-2スパイク(S)タンパク質の完全長をコードし、ウイルス中和抗体を誘導するために無傷のウイルスに似せています[3]。無作為化臨床試験と一致して、BNT162b2は、実環境におけるCOVID-19関連の幅広い転帰において高い有効性を示しました[5]。とはいえ、ワクチンの長期的な安全性と有効性のモニタリングなど、多くの課題が残っています。このため、さらなる評価と調査が必要です。BNT162b2の安全性プロファイルは、現在、短期間の臨床試験からしか得られていません。BNT162b2のあまり一般的でない副作用として、心膜炎、不整脈、深部静脈血栓症、肺塞栓症、心筋梗塞、頭蓋内出血、血小板減少症などが報告されています[4,9,10,11,12,13,14,15,16,17,18,19,20]。また、他の種類のワクチンで観察された副作用を報告する研究もあります[21,22,23,24]。ワクチンに関連する副作用のメカニズムをよりよく理解するためには、臨床研究だけでなく、細胞や分子レベルでの解析が必要です。
最近の研究では、SARS-CoV-2 RNAが逆転写され、ヒト細胞のゲノムに組み込まれることが示された[25]。このことは、SARS-CoV-2 RNAの一部をコードしているBNT162b2でもこのようなことが起こりうるのではないかという疑問を生じさせる。ファイザー社が欧州医薬品庁(EMA)に提供した薬物動態データでは,BNT162b2の生体内分布が,放射性標識したLNPとルシフェラーゼmodRNAをマウスとラットに筋肉内注射することで調べられた.最初の時間点(0.25 時間)からほとんどの組織で放射能が検出され、その結果、注射部位と肝臓が主な分布部位であり、投与後 8-48 時間で最大濃度が観察された [26].さらに,BNT162b2 の注射を受けた動物では,肝臓の肥大,空胞化,γGT(γglutamyl transferase)値の上昇,AST(aspartate transaminase)値および ALP(Alkaline phosphatase)値の上昇などの可逆的肝障害が観察された[26].LNP送達システムによって引き起こされる一過性の肝障害は以前に報告されています[27,28,29,30]。それにもかかわらず、modRNAを含まない空のLNPだけでは、有意な肝障害が生じないことも示されています[27]。そこで、本研究では、BNT162b2のヒト肝細胞株への影響をin vitroで調べ、BNT162b2が内因性メカニズムによりDNAに逆転写されるかどうかを調べることを目的とする。
2. 材料と方法
2.1. 細胞培養
Huh7細胞(JCRB Cell Bank, Osaka, Japan)は、10%(v/v)牛胎児血清(Sigma-Aldrich, F7524-500ML, Burlington, MA, USA)および1%(v/v)ペニシリン-ストレプトマイシン(HyClone, SV30010, Logan, UT, USA)で補充したDMEM培地(HyClone, HYCLSH30243.01 )で37℃、5%CO2で培養させた。BNT162b2処理には、Huh7細胞を24ウェルプレートに200,000cells/wellの密度で播種した。BNT162b2 mRNAワクチン(Pfizer BioNTech, New York, NY, USA)は、製造者のガイドラインに記載されているように、滅菌0.9%塩化ナトリウム注射液、USPで最終濃度100 μg/mLに希釈した[31]。次に、BNT162b2懸濁液を、最終濃度が0.5、1.0、または2.0μg/mLとなるように細胞培養液中に添加した。Huh7細胞を、BNT162b2と共に、またはBNT162b2なしで、6、24、および48時間インキュベートした。細胞をPBSで十分に洗浄し、トリプシン化によって採取し、さらなる使用まで-80℃で保存した。
2.2. リアルタイムRT-QPCR
細胞からのRNAは、RNeasy Plus Mini Kit(Qiagen, 74134, Hilden, Germany)を用いて、製造元のプロトコルにしたがって抽出した。RT-PCR は RevertAid First Strand cDNA Synthesis kit (Thermo Fisher Scientific, K1622, Waltham, MA, USA) を用いてメーカーのプロトコルにしたがって行った。リアルタイムqPCRは、BNT162b2、LINE-1、ハウスキーピング遺伝子ACTBおよびGAPDHのプライマーを用いて、Maxima SYBR Green/ROX qPCR Master Mix (Thermo Fisher Scientific, K0222, Waltham, MA, USA) を使用して行った(表1)。
Table 1. RT-qPCRとPCRのプライマー配列。
2.3. 免疫蛍光染色と共焦点画像化
Huh7細胞を8室スライド(LAB-TEK, 154534, Santa Cruz, CA, USA)で40,000cells/wellの密度で、BNT162b2 (0.5, 1 or 2 µg/mL) とともに6時間、またはBNT162b2を含まない状態で培養した。免疫組織化学は、一次抗体抗LINE-1 ORF1p マウスモノクローナル抗体(Merck, 3574308, Kenilworth, NJ, USA)、二次抗体 Cy3 Donkey anti-mouse (Jackson ImmunoResearch, West Grove, PA, USA) およびHoechst (Life technologies, 34850, Carlsbad, CA, USA) を用いて、サーモフィッシャー(Waltham, MA, USA)からのプロトコルに従って実施された。Zeiss LSM 800と63X油浸対物レンズを用いて1条件につき2枚の画像を撮影し、ImageJ 1.53cにより1画像あたり15細胞の個々の全細胞領域と核領域について染色強度を定量化した。細胞質に対するLINE-1染色強度は、細胞全体の染色強度から核の染色強度を差し引くことで算出した。偏りを防ぐため、細胞のすべての画像に乱数を割り当てた。核(Hoechst染色により決定)と細胞全体(LINE-1蛍光の境界により決定)をマークするために、フリーハンド選択ツールを使用した。その後、これらの領域を測定し、平均強度を用いて、各グループを比較した。
2.4. ゲノムDNA精製、PCR増幅、アガロースゲル精製、サンガーシークエンス
ゲノムDNAは、先に述べたプロトコルに従って、PBNDバッファ(10 mM Tris-HCl pH 8.3, 50 mM KCl, 2.5 mM MgCl2, 0.45% NP-40, 0.45% Tween-20)により細胞ペレットから抽出した[32]。DNA調製物から残留RNAを除去するために、RNase (100 µg/mL, Qiagen, Hilden, Germany) をDNA調製物に加え、37℃で3時間、その後95℃で5分インキュベートした。その後、BNT162b2を標的とするプライマー(配列は表1に示す)を用いて、以下のプログラムでPCRを行った。95℃で5分、95℃30秒、58℃30秒、72℃1分の35サイクル、最後に72℃5分、12℃5分。PCR産物を1.4% (w/v)アガロースゲルで分析した。予想されるサイズ(444 bps)のアンプリコンに対応するバンドを切り出し、QIAquick PCR Purification Kit(Qiagen, 28104, Hilden, Germany)を用いて、メーカーの説明書に従ってDNAを抽出した。DNAアンプリコンの配列は、Sanger sequencing (Eurofins Genomics, Ebersberg, Germany) により確認した。
統計情報
統計的比較は、両側スチューデントのt検定およびANOVAを用いて行った。データは、平均値±SEMまたは±SDで表した。p < 0.05の差は有意であるとみなされる。
2.5. 倫理的記載事項
Huh7 細胞株は、日本リサーチバイオリソースコレクション(JCRB)細胞バンクから入手した。
3. 成果
3.1. BNT162b2はヒト肝細胞株Huh7細胞に高い効率で侵入する
BNT162b2がヒト肝細胞に入るかどうかを調べるために、ヒト肝細胞株Huh7にBNT162b2を曝露した。Huh7細胞におけるLNP送達の取り込み動態に関する以前の研究では,LNPの最大生物学的効果は4-7時間の間に観察された[33].そこで,本研究では,Huh7細胞を,濃度の増加したBNT162b2(0.5,1.0および2.0 µg/mL)とともに,あるいはまったく加えずに6,24および48時間培養し,細胞からRNAを取り出して,図1に示すようにBNT162b2配列を標的とするプライマーを用いてリアルタイム定量逆転写PCR(RT-qPCR)を実行した.BNT162b2の全配列は公開されており[34]、2ヌクレオチドのキャップ;ヒトα-グロビン遺伝子の5′-UTRを組み込んだ5′-非翻訳領域(UTR)を含んでいる。SARS-CoV-2のSタンパク質全長と2つのプロリン変異;ヒトミトコンドリア12S rRNA(mtRNR1)セグメントとヒトAES/TLE5遺伝子セグメントと2つのC→U変異;ポリ(A)テールを含む3′-UTRを含んでいる。BNT162b2のSタンパク質配列を詳細に解析した結果、ヒトゲノム配列と100%一致する配列が124個、19〜26ntsに1塩基(nt)のみミスマッチのある配列が3個見つかった(表S1、補足資料参照)。BNT162b2のRNA量を検出するために、フォワードプライマーがSARS-CoV-2のSタンパク質領域に、リバースプライマーが3′-UTRに位置するようにプライマーを設計し、ヒトゲノム領域と非特異結合せずにBNT162b2特有のPCRアンプリコンを検出することができるように工夫した。
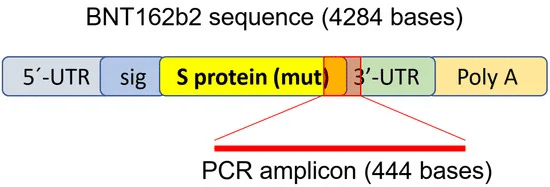
図1. BNT162b2のmRNAレベルの検出と逆転写に使用したPCRプライマーセット。BNT162b2の図解は、既出の文献[34]から引用した。
RT-qPCRの結果、BNT162b2で処理したHuh7細胞は、6、24、および48時間で、ハウスキーピング遺伝子と比較して高レベルのBNT162b2 mRNAを有した(図2、例外的に高いレベルのため、ログ2-ΔCTで示された)。3つのBNT162b2濃度は、1.0と2.0μg/mLの間の有意差が48時間で観察されたことを除いて、異なる時点で同様の細胞内BNT162b2 mRNAレベルを導いた。BNT162b2 mRNAレベルは6時間と比較して24時間で著しく減少し、48時間で再び増加した。

図2. BNT162b2で処理したHuh7細胞におけるBNT162b2 mRNAレベル。Huh7 細胞を、BNT162b2 なし(Ctrl)、または 0.5(V1)、1(V2)、2 µg/mL(V3) で 6 時間(緑の点)、24 時間(オレンジの点)、48 時間(青の 点)処理した。RNAを精製し、BNT162b2を標的としたプライマーを用いてqPCRを実施した。BNT162b2のRNAレベルは、ハウスキーピング遺伝子GAPDHおよびACTBに対する対数2-ΔΔCT値として示される。結果は5回の独立した実験から得たものである(n=5)。それぞれのグループ間の差は、両側スチューデントのt-検定を用いて分析した。データは、平均±SEMとして表される。(* p < 0.05; ** p < 0.01; *** p < 0.001 vs. 各時点におけるそれぞれのコントロール、または示された通り)。
3.2. ヒト内在性逆転写酵素 Long Interspersed Nuclear Element-1 (LINE-1) に対する BNT162b2 の影響
ここでは、LINE-1遺伝子の発現に対するBNT162b2の影響を検討した。BNT162b2 (0, 0.5, 1.0, 2.0 µg/mL) で6, 24, 48時間処理したHuh7細胞から精製したRNAについて、LINE-1を標的とするプライマーを用いてRT-qPCRを実施した。2.0 µg/mL BNT162b2によって6時間後にコントロールと比較して有意に増加したLINE-1発現が観察されたが、より低いBNT162b2濃度はすべての時点でLINE-1発現を減少させた(図3)。

図3. BNT162b2で処理したHuh7細胞におけるLINE-1 mRNAレベル。Huh7細胞を、BNT162b2なしで(Ctrl)、または0.5(V1)、1(V2)、および2μg/mL(V3)で、6時間(緑の点)、24時間(赤い点)、および48時間(青い点)処理しました。RNAを精製し、LINE-1を標的とするプライマーを用いてqPCRを実施した。LINE-1のRNAレベルは、ハウスキーピング遺伝子GAPDHおよびACTBに対する2-ΔΔCT値として示される。結果は、5つの独立した実験からのものである(n=5)。それぞれのグループ間の差は、両側スチューデントのt-検定を用いて分析した。データは、平均±SEMとして表される。(* p < 0.05; ** p < 0.01; *** p < 0.001 vs. 各時点におけるそれぞれのコントロール、または示された通り; † p < 0.05 vs. 6 h-Ctrl).
次に、BNT162b2がLINE-1タンパク質量に及ぼす影響を調べた。全長LINE-1は、5′非翻訳領域(UTR)、2つのオープンリーディングフレーム(ORF)、ORF1およびORF2、3′UTRからなり、そのうちORF1はシャペロン作用を有するRNA結合蛋白質である。LINE-1のレトロトランスポジション活性は,ORF1の核内移行が関与していることが示されている[35].BNT162b2 (0.5, 1.0 and 2.0 µg/mL) で6時間処理したまたは処理していないHuh7細胞を固定し、細胞核を可視化するためにLINE-1 ORF1p に結合する抗体、およびDNA特異的プローブHoechstで染色した(図4a)。免疫蛍光染色強度の定量化により、BNT162b2は、試験したすべての濃度において、細胞全体領域および核の両方においてLINE-1 ORF1pタンパク質レベルを増加させることが示された(図4b〜d)。
図4. BNT162b2で処理したHuh7細胞のLINE-1タンパク質分布に関する免疫組織化学的研究。細胞を固定し、LINE-1 ORF1pに結合する抗体(赤色)および細胞核を可視化するためのDNA特異的プローブHoechst(青色)で染色した。(a)BNT162b2で処理した、またはBNT162b2なしで処理したHuh7細胞におけるLINE-1発現の代表的な画像である。(b-d)全細胞領域(b)、細胞質(c)、および核(d)におけるLINE-1タンパク質の定量化。すべてのデータは、One-Way ANOVAを用いて分析し、グラフは、GraphPad Prism V 9.2を用いて作成した。すべてのデータは平均値±SDで示した(** p < 0.01; *** p < 0.001; **** p < 0.0001 as indicated)。
3.3. Huh7細胞における逆転写されたBNT162b2 DNAの検出
以前の研究で、LINE-1タンパク質の核内への侵入はレトロトランスポジションと関連していることが示されている[35]。上記の免疫蛍光染色実験では、核内のLINE-1レベルの増加は、BNT162b2の最低濃度(0.5 µg/mL)で既に観察された。LINE-1が上昇したときにBNT162b2がDNAに逆転写されるかどうかを調べるために、0.5 µg/mLのBNT162b2で6、24、48時間処理したHuh7細胞からゲノムDNAを精製し、RNaseで処理してRNAを除去し、図1に示すようにBNT162b2標的プライマーでPCR処理を施した。増幅されたDNA断片は、電気泳動によって可視化され、ゲル精製された(図5)。BNT162b2のDNAアンプリコンは、3つのタイムポイント(6、24、48時間)すべてで検出された。サンガーシークエンスにより、このDNAアンプリコンはプライマーに挟まれたBNT162b2の配列と同一であることが確認された(表2)。DNAアンプリコンがBNT162b2 RNAではなくDNAに由来することを確認するために、0.5 µg/mL BNT162b2で6時間処理したHuh7細胞から精製したRNAについても、RNase処理ありまたはなしでPCRを行った(図5のCtrl 5および6)が、PCRにかけたRNA試料からはアンプリコンが検出されなかった。
図5. BNT162b2で処理したHuh7細胞におけるBNT162b2のDNAアンプリコンの検出。Huh7細胞は、BNT162b2なしで(Ctrl)または0.5 µg/mLで6、24、48時間処理した。図1および表1に示すように、BNT162b2を標的とするプライマーを用いて、すべてのサンプルについてPCRを行った。DNAアンプリコン(444 bps)はアガロースゲル上で可視化された。BNT:BNT162b2; L:DNA ladder; Ctrl1:Huh7 培養細胞; Ctrl2: 6時間後に回収したBNT162b2処理なしのHuh7細胞;Ctrl3: 24時間に収集したBNT162b2処理なしのHuh7細胞;Ctrl4:48時間に収集したBNT162b2処理なしのHuh7細胞;Ctrl5:BNT162b2を0.5μg/mLで6時間処理したHuh7細胞からのRNA;Ctrl6:BNT162b2を0.5μg/mLで6時間処理しRNaseで消化されたHuh7細胞からのRNA。
Table 2. BNT162b2アンプリコンのサンガーシークエンス結果。
4. ディスカッション
本研究では、COVID-19 mRNAワクチンBNT162b2がin vitroでヒト肝細胞株Huh7に侵入することができるという証拠を提示した。BNT162b2 mRNAは、BNT162b2曝露後6時間という速さで、細胞内でDNAに逆転写される。逆転写のメカニズムとして、内因性逆転写酵素LINE-1を介する可能性があり、LINE-1の核内タンパク質分布はBNT162b2によって上昇した。
肝細胞におけるLNPの細胞内蓄積はin vivoで証明されている[36].BNT162b2に関する前臨床研究では,BNT162b2がヒト細胞株HEK293T細胞に入り,BNT162b2抗原の強固な発現につながることが示された[37].そこで、本研究では、まず、ヒト肝細胞株Huh7細胞におけるBNT162b2の侵入を検討した。この研究で使用したBNT162b2濃度の選択には説明が必要である。BNT162b2は3週間間隔で2回投与され,各投与量は0.3 mLに30 µgのBNT162b2を含むので,注射部位の局所濃度は最高でも100 µg/mLとなる[31].同様のLNP送達システムを用いたH10N8およびH7N9インフルエンザウイルスに対するmRNAワクチンに関する以前の研究では,mRNAワクチンは,肝臓,脾臓,心臓,腎臓,肺および脳などのいくつかの器官にむしろ非特異的に分布し,肝臓での濃度は,筋肉内注射部位での濃度よりもおよそ100倍低くなることが示されている[38].ファイザーがEMAに提出したBNT162b2の評価報告書では、ラットの薬物動態分布試験で、総投与量の比較的大きな割合(最大18%)が肝臓に分布することが実証された[26]。そのため,肝細胞を用いた実験では,0.5,1,2μg/mLのワクチンを使用することにした.しかし,より広い範囲の低濃度および高濃度のBNT162b2の効果も,今後の研究で検証する必要がある.
今回の研究では、in vitroでの検討にはヒト肝細胞株を採用した。肝細胞にもワクチン由来のSARS-CoV-2スパイクタンパク質が存在し、あらかじめプライミングされたスパイクタンパク質反応性細胞傷害性T細胞のターゲットとなる可能性があるため、検討に値すると思われる。BNT162b2ワクチン接種後に自己免疫性肝炎を発症した人の症例報告[39]がある.BNT162b2の肝機能への潜在的影響についてより良く理解するために、今後の研究ではin vivoモデルが望まれる。
BNT162b2 の毒性報告では,遺伝毒性試験および発がん性試験は行われていない[26].我々の研究では,肝細胞株Huh7においてBNT162b2がDNAに逆転写されることが示されており,BNT162b2由来のDNAが宿主ゲノムに統合されてゲノムDNAの完全性に影響を与え,遺伝毒性副作用を介する可能性があるのではないかという懸念が生じる可能性がある。現段階では、BNT162b2から逆転写されたDNAが細胞ゲノムに統合されるかどうかは分かっていない。BNT162b2がゲノムの完全性に及ぼす影響を実証するためには、BNT162b2に曝露した細胞の全ゲノム配列決定や、BNT162b2のワクチン接種を受けたヒト被験者の組織など、さらなる研究が必要である。
ヒト自律型レトロトランスポゾンLINE-1は、細胞内在性逆転写酵素であり、ヒトに唯一残る活性型トランスポゾンで、自身と他の非自律型要素をレトロトランスポーズすることができ [40,41]、ヒトゲノムの約17%はLINE-1配列で構成されています [42](P. 34)。非自律的なAlu要素,short, interspersed nucleotide element (SINEs), variable-number-of-tandem-repeats (VNTR) や細胞内のmRNA処理された偽遺伝子は,トランスで働くLINE-1逆翻訳タンパク質によって逆翻訳される [43, 44].最近の研究では、内在性のLINE-1がSARS-CoV-2配列の逆転写と感染ヒト細胞のゲノムへの組み込みを仲介していることが示された[25]。さらに、内因性LINE-1の発現は、SARS-CoV-2感染を含むウイルス感染時にしばしば増加する [45,46,47]. これまでの研究から,LINE-1レトロトランスポジション活性は,RNA代謝 [48,49],DNA損傷応答 [50],オートファジー [51] によって制御されていることが分かっている.LINE-1の効率的なレトロトランスポジションは,しばしば細胞周期や有糸分裂中の核膜破壊 [52,53] ,外来レトロウイルス [54,55] と関連しており,LINE-1の核内への侵入を促進する.我々の研究では,試験したすべての濃度(0.5,1,2μg/mL)で,BNT162b2によって核内の免疫組織化学で決定されるLINE-1 ORF1p分布が増加し,LINE-1遺伝子発現の上昇がBNT162b2最高濃度(2μg/mL)で検出された.遺伝子の転写はクロマチン修飾、転写因子制御、RNA分解速度によって制御されるが、タンパク質の翻訳制御には開始コドン上でのリボソーム動員、ペプチド伸長の調節、タンパク質合成の終了、またはリボソーム生合成が含まれることは注目に値する。この2つのプロセスは異なるメカニズムで制御されているため、外的なチャレンジに応答して必ずしも同じ変化パターンを示すとは限らない。BNT162b2に対するLINE-1活性の正確な制御については、さらなる研究が必要である。
本研究で用いた細胞モデルは癌細胞であり、非分裂性の体細胞とは異なり、DNA複製が活発である。また、Huh7細胞は、RNA代謝に関与するタンパク質の発現が増加するなど、大きく異なる遺伝子およびタンパク質発現を示すことが示されている[56]。しかし,骨髄や上皮基底層などのヒト組織や胚発生期においても細胞増殖は活発であり,そのような条件下でのBNT162b2のゲノムインテグリティへの影響を検討する必要がある.さらに、LINE-1の効果的なレトロトランスポジションは、ヒト神経細胞のような非分裂細胞や終末分化細胞においても報告されている[57,58]。
ファイザー社のEMA評価報告書は、BNT162b2が脾臓(1.1%未満)、副腎(0.1%未満)に分布し、さらに卵巣および精巣(0.1%未満)において低く測定できる放射能があることも示しています[26]。さらに、BNT162b2の胎盤移行に関するデータは、ファイザー社のEMA評価報告書からは得られない。その結果,BNT162b2のmRNAは,注射部位濃度の0.5%に相当する濃度(0.5μg/mL)でHuh7細胞に容易に入り,LINE-1遺伝子およびタンパク質の発現変化を誘発し,6時間以内にBNT162b2の逆転写が検出されることが分かった.したがって、in vitroおよびin vivoの両方で、他の細胞タイプや組織に対するBNT162b2の効果をさらに調査することが重要である。
5. 結論
本研究は、COVID-19 mRNAワクチンBNT162b2のヒト肝細胞株への影響に関する初のin vitro研究である。BNT162b2の細胞内への速い進入と、それに続くBNT162b2 mRNAのDNAへの細胞内逆転写に関する証拠を提示するものである。
補足資料
以下のサポート情報 https://www.mdpi.com/article/10.3390/cimb44030073/s1 でダウンロードできます。
著者による寄稿
M.A.、F.O.F.、D.Y.、M.B.、C.L.はin vitroの実験を実施した。M.A.とF.O.F.はデータ解析を行った。M.R.とY.D.M.は研究の実施に貢献し、設計し、研究を監督した。Y.D.M.は全著者の意見を取り入れながら論文を執筆した。すべての著者は、掲載された原稿を読み、同意している。
資金提供
本研究は、Swedish Research Council, Strategic Research Area Exodiab, Dnr 2009-1039, Swedish Government Fund for Clinical Research (ALF) および Skåne University Hospital の財団の支援を受けて実施された。
インスティテューショナル・レビュー・ボード(Institutional Review Board)声明
該当事項はありません。
インフォームド・コンセントに関する声明
該当事項はありません。
データの利用可能性に関する声明
本研究の結果を裏付けるすべてのデータは、論文および添付資料の中で公開されています。
謝辞
Sven Haidl, Maria Josephson, Enming Zhang, Jia-Yi Li, Caroline Haikal, and Pradeep Bompadaに感謝します。
利益相反
著者は利益相反のないことを宣言する。
コロワクは遺伝子組み換えしないもん!と鼻息を荒くしていた人達、ゲームオーバー
— めいこ(極限を超えて自由になる - Freedom Beyond Limits) (@positiverevolu3) February 26, 2022
動物実験でファイザー毒注射が、肝臓の遺伝子組み換えをしたことが証明された
危惧していた通り逆転写deathhttps://t.co/glVtE9lfch