
【完成版】PythonでL-SystemをGAさせてみました。
前回の記事「PythonでL-SystemをGAさせてみました。」で楽しくなってきたので、図書館で本を借り、どんどんPythonの深みにはまってみることにしました。
Head First Python 第2版
――頭とからだで覚えるPythonの基本
Paul Barry 著、嶋田 健志 監訳、木下 哲也 訳
2018年03月 発行
624ページ
ISBN978-4-87311-829-1
読み進めながら、初版に手を加えていきました。
なんとなく書いていたところやC言語の発想で組んでいたところを
少しずつ組み替えていきます。
Pythonの基本データ構造であるリスト、辞書、タプル、セットを理解し、スライス記法ができるようになりました。
また、言語が分かり自力で組めるようになったので、GA部分の改良に着手しました。
染色体構造の変化による突然変異
* 欠失 - 染色体の一部が失われる。
* 例: 白いカラス、オレンジ色のモグラ、黒→白になった犬(ラブラドールレトリバー種)
* 逆位 - 染色体の一部が通常の逆の向きになる。
* 重複 - 染色体の一部が重複する。
* 転座 - 染色体の一部が切れて、別の染色体に繋がる。
このような処理は文字列操作の良い練習となります。上記の操作の一部をPythonで実装してみました。
def mutation(s: str) -> str:
'''一定確率で突然変異を起こす。'''
i = random.randint(0, 9)
if i == 0: #単一遺伝子の変異
p = random.randint(0, len(s)-1)
s = s[:p]+s_random()+s[p+1:]
elif i == 1: #欠失-遺伝子の一部を失う。
p = random.randint(1, len(s)-1)
s = s[:p]+s[p+1:]
elif i == 2: #逆位-遺伝子の一部が逆向きになる。
p1 = random.randint(0, len(s)-2)
p2 = random.randint(p1, len(s)-1)
sInv = s[p1:p2:-1]
s = s[:p1]+sInv+s[p2:]
elif i == 3: #重複-遺伝子の一部が重複する。
p1 = random.randint(0, len(s)-2)
p2 = random.randint(p1, len(s)-1)
sDup = s[p1:p2]
s = s[:p1]+sDup+sDup+s[p2:]
return s
Google Colaboratoryで共有できる形にしました。
実際に動かしてみてください。
Googleアカウントひとつで、無料でクラウド上のPython環境が手に入るよい時代です。
実行例

交配は毎回ランダムで行われるため、どのような形質が次世代に引き継がれるか分かりません。

選択した結果を赤枠で表示しています。

今回は小ぶりに成長しています。

形状が安定してきました。

ここはじっと我慢です。

形状が少し動き始めました。

左流れの樹形を選んでみます。

まだ形状は進化しません。

少し変化の兆しが見えました。
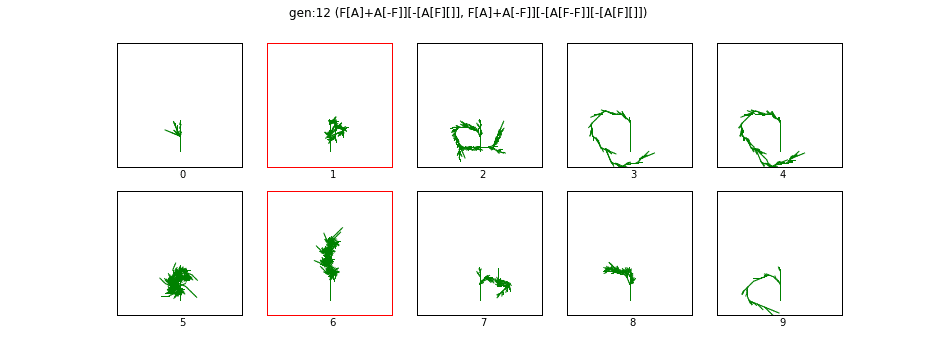
面白い形になかなか発展しません。






このあたりまで根競べです。

ちょっと動き出したかな。

指向を変えて選択してみます。




今回はあまり面白い形には進化しませんでした。
このシステムの面白い所は、「ユーザーが選択を行う」ことが、「評価関数」として機能していることです。ユーザーがよいと思う方向に形状が進化していきます。

ソースは以下のとおりです。
#@title PythonでL-SystemをGAする。2 bbd
#@markdown 選択: の説明
#@markdown
#@markdown ?:現世代の遺伝子を表示します。
#@markdown q:終了
#@markdown 0-9:遺伝子を2つ選択します。 (例:"12")
#@markdown
#@markdown ※注意※ 選択: が表示されない場合はコードの実行を中断し、再度実行してください。
#昔どこかで見たようなシミュレーションを作りたくなりました。
#思い付きだけでなんとかなる時代にITの進化を実感しました。
#参考にした記事
#PythonエンジニアによるPython3学習サイト
#https://www.python.ambitious-engineer.com/
#E.V.ジュニアさん
#PythonでL-Systemsを作る(5) 植物の成長システム
#https://note.com/evjunior/n/nab0229b23b40
#Pythonの勉強をした結果、もう少し書き換えたくなった。
#Pythonの作法に近づける。
import numpy as np
import matplotlib.pyplot as plt
import random
#グローバル変数
repeat = 4
distance = 2*(1/2) ** repeat
angle = 22.5/180*np.pi
nrows, ncols = 2, 5
def forward(st: 'state = [[x, y], theta]', axis: 'Axes') -> 'state = [[x, y], theta]':
'''前進する。'''
th = st[1]
x1, y1 = st[0][0], st[0][1]
x2, y2 = x1 + distance*np.cos(th), y1 + distance*np.sin(th)
axis.plot([x1, x2], [y1, y2], lw = 1, color = 'green')
return [[x2, y2], th]
def rotate(coef: '1:ccw, -1:cw', st: 'state = [[x, y], theta]') -> 'state = [[x, y], theta]':
'''回転する。'''
th = st[1]
th = th + coef*angle
return [st[0], th]
def turtle(command: str, st: 'state = [[x, y], theta]', stack: 'リスト', axis: 'Axes') -> 'state = [[x, y], theta]':
'''コマンドに従いタートル描画を行う。'''
if command == "A" or command == "F":
st = forward(st, axis)
if command == "+":
st = rotate(1, st)
if command == "-":
st = rotate(-1, st)
if command == "[":
stack.append(st)
if command == "]":
#スタックアンダーフローに備える。
if (len(stack) > 0):
st = stack.pop()
return st
def draw_one(commands: str, st: 'state = [[x, y], theta]', generator: '遺伝子', axis: 'Axes'):
'''ひとつ描く。'''
#スタックをクリアする。
stack = []
for i in range(repeat):
commands = commands.translate(str.maketrans(generator))
for command in commands:
st = turtle(command, st, stack, axis)
def s_random() -> str:
'''ランダムでgeneratorの文字を与える。'''
return random.choice(('A', 'F', '+', '-', '[', ']'))
def mutation(s: str) -> str:
'''一定確率で突然変異を起こす。'''
i = random.randint(0, 9)
if i == 0: #単一遺伝子の変異
p = random.randint(0, len(s)-1)
s = s[:p]+s_random()+s[p+1:]
elif i == 1: #欠失-遺伝子の一部を失う。
p = random.randint(1, len(s)-1)
s = s[:p]+s[p+1:]
elif i == 2: #逆位-遺伝子の一部が逆向きになる。
p1 = random.randint(0, len(s)-2)
p2 = random.randint(p1, len(s)-1)
sInv = s[p1:p2:-1]
s = s[:p1]+sInv+s[p2:]
elif i == 3: #重複-遺伝子の一部が重複する。
p1 = random.randint(0, len(s)-2)
p2 = random.randint(p1, len(s)-1)
sDup = s[p1:p2]
s = s[:p1]+sDup+sDup+s[p2:]
return s
def crossover(pa: '[p0, p1]') -> '[p0, p1]':
'''2組のリストpaをランダムな位置pで交叉する。'''
p = random.randint(1, len(pa[0])-2)
res = ["", ""]
res[0] = pa[0][:p]+pa[1][p:]
res[1] = pa[1][:p]+pa[0][p:]
return res
#初期値は既知のパターン
parents0 = "FA[+F[+FA][-F]][-FA[+F][-F]]"
parents1 = "F-[[A]+A]+F[+FA]-A"
parents = [parents0, parents1]
#メインループ
nGen = 1
while True:
#作図領域の初期化を行う。
plt.close()
fig, ax = plt.subplots(nrows, ncols,
figsize = (13, 5),
facecolor='lightgray',
subplot_kw=dict(facecolor='white'))
i = 0
for r in range(nrows):
for c in range(ncols):
ax[r, c].axis([0, 8, 0, 8])
ax[r, c].text(4, -0.7, hex(i)[2:])
ax[r, c].spines["top"].set_linewidth(1)
ax[r, c].spines["left"].set_linewidth(1)
ax[r, c].spines["bottom"].set_linewidth(1)
ax[r, c].spines["right"].set_linewidth(1)
ax[r, c].tick_params('both', length = 0, which = 'both')
ax[r, c].set_xticklabels([])
ax[r, c].set_yticklabels([])
ax[r, c].spines["top"].set_color('black')
ax[r, c].spines["left"].set_color('black')
ax[r, c].spines["bottom"].set_color('black')
ax[r, c].spines["right"].set_color('black')
i = i + 1
#parentよりchildrenを得る。
children = []
while True:
#遺伝子は任意の位置で交叉する。
cv = crossover(parents)
#一定確率で突然変異する。
cv[0], cv[1] = mutation(cv[0]), mutation(cv[1])
#リストに追加する。
children.append(cv[0])
children.append(cv[1])
#childrenを整理し重複をなくす。
children = list(set(children))
#遺伝子が一定数増えるまで繰り返す。
if len(children) >= nrows * ncols:
break
#作図を行う。
i = 0
for cur_row in range(nrows):
for cur_col in range(ncols):
initiator = "A"
state = [[4, 1], np.pi/2]
generator = {"F":"FF", "A":children[i]}
draw_one(initiator, state, generator, ax[cur_row, cur_col])
i = i + 1
fig.suptitle("gen:" + str(nGen) + " (" + parents[0] + ", " + parents[1] + ")")
plt.show()
#次の世代へ残す個体を選択する。例:"01"、"00"など。”?"で現世代のgeneratorを表示する。
sel = ""
while True:
sel = input("選択:")
if sel == "?":
for j in range(len(children)):
print(str(j) + ":" + children[j])
else:
break
if (sel == "q"):
break;
r, c = int(sel[0]) // ncols, int(sel[0]) % ncols
ax[r, c].spines["top"].set_color('red')
ax[r, c].spines["left"].set_color('red')
ax[r, c].spines["bottom"].set_color('red')
ax[r, c].spines["right"].set_color('red')
r, c = int(sel[1]) // ncols, int(sel[1]) % ncols
ax[r, c].spines["top"].set_color('red')
ax[r, c].spines["left"].set_color('red')
ax[r, c].spines["bottom"].set_color('red')
ax[r, c].spines["right"].set_color('red')
fig.savefig("img{:02d}".format(nGen) + ".png")
plt.show()
p0, p1 = int(sel[0]), int(sel[1])
#選択された個体を親にする。
parents[0], parents[1] = children[p0], children[p1]
nGen = nGen + 1
ようやく私もPythonの良さを理解できるようになりました。
直観的にコードが組め、コピペでの再利用もしやすい、お手軽な言語として活用していきたいと思います。
最後までご覧いただき、誠にありがとうございました。
追記
先日、医療関係のベンチャー交流会に参加しました。CRISPR-Cas9などのゲノム編集を用いたビジネスの模索が盛んでした。そういえば、2020年のノーベル化学賞でCRISPR-Cas9の名前だけは知っていましたが、なんとなく繋がってきました。今回私が作ったものは、タートルグラフィックスの作図ルールを遺伝子見立てて、人為選択により進化させるものでしたが、「ゲノム編集」の技術は研究者が求める遺伝子をリアルに作り出す方法であることを知りました。遺伝子はプログラミングできる時代になっていたことに大いに驚きました。
#Python #LSystem #GA #人工生命 #遺伝的アルゴリズム #植物シミュレーション #GoogleColaboratory
