ggplot2 を使って、ボルケーノプロットを作成
ボルケーノプロットは、遺伝子発現の情報を比較した結果によく用いられるプロット図です。これを ggplot2 で作成する方法を紹介します。
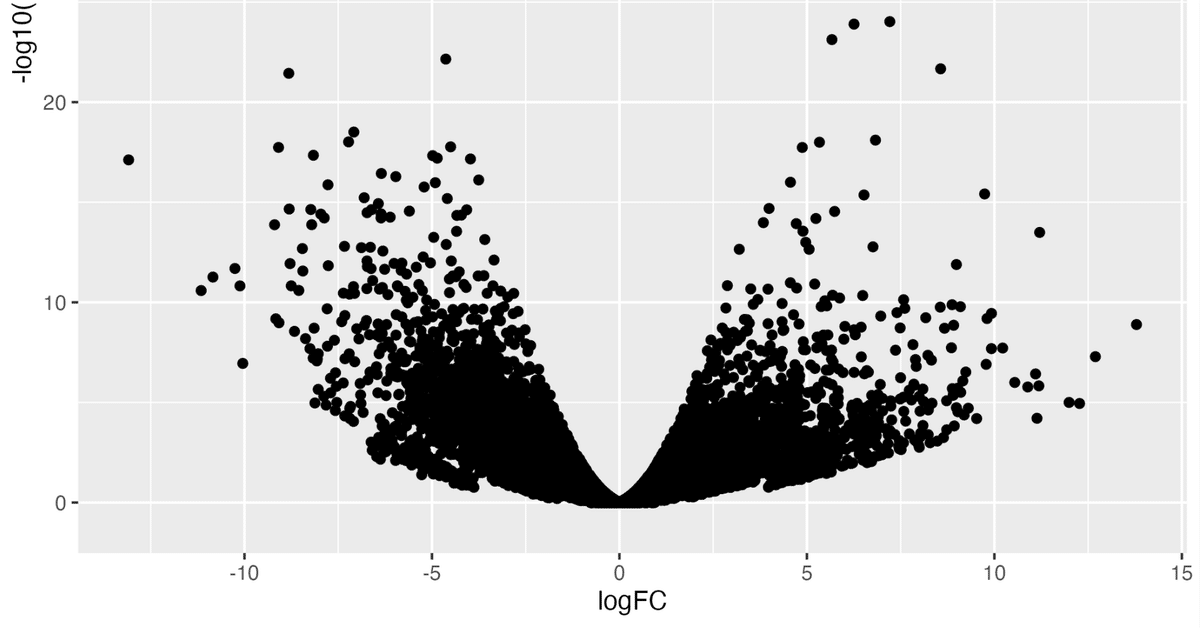
ボルケーノプロットは「散布図」の1種です。 そのため、以前紹介したgeom_point() を使って普通に作成できます。ただ、x軸に logFC 、y軸に log変換された p-value を用いているというのが特徴です。散布図のドットが広がって、噴火しているように見えるので、ボルケーノ(volcano, 火山)プロットと呼ばれます。
プロットに用いるデータ
プロットに用いるデータは、RNA-seq などで発現変動遺伝子を算出した結果です。下記のように、 logFC と p-value (PValue) のデータを含んでいる必要があります。これらの算出が終わっていれば、リードカウントの値は不要です。
> de_result <- read_tsv("./results/edgeR.DE_results")
> de_result
# A tibble: 27,882 × 5
GeneID logFC logCPM PValue FDR
<chr> <dbl> <dbl> <dbl> <dbl>
1 MTND1P23 9.06 7.65 3.01e-51 8.39e-47
2 CD177 -9.98 8.26 1.74e-32 2.43e-28
3 CDH3 7.27 5.80 1.44e-31 1.34e-27
4 CPNE7 7.21 4.76 9.51e-25 6.63e-21
5 RP11-750B16 6.26 5.83 1.27e-24 7.08e-21
6 ETV4 5.67 5.82 7.52e-24 3.50e-20
7 MT2A -4.63 7.07 7.08e-23 2.82e-19
8 CLDN2 8.57 6.92 2.16e-22 7.54e-19
9 SLC6A19 -8.82 5.15 3.64e-22 1.13e-18
10 BEST4 -7.08 5.66 3.12e-19 8.70e-16ボルケーノプロット
散布図なので、コード自体は単純です。ggplot() の aes() で、x軸に logFC、y軸に -log10(PValue) を指定します。
g <- ggplot(de_result, aes(logFC, -log10(PValue)))
g + geom_point()*PValue を log10 変換して、さらにマイナスで符号を入れ替えている点に注意してください。

geom_point() で、ボルケーノプロットが表示されます。
